柴胡Bupleuri Radix属于伞形科(Umbelliferae)植物,有约200种[1],《中国药典》2015年版中规定的正品柴胡为伞形科植物柴胡Bupleurum chinense DC. 或狭叶柴胡B. scorzonerifolium Wlid. 的干燥根。前者俗称“柴胡”,后者俗称“南柴胡”,是我国常用大宗中药材之一[2]。柴胡是河北省的道地药材之一,每年的用量巨大。但是,由于市场上的柴胡药材来源复杂,品种较多[3],加之还有一些掺杂使假的现象,使得柴胡的临床用药安全受到很大的威胁。利用传统的理化鉴别和性状鉴别的方法不能准确鉴别柴胡药材的来源和种属,因此,需要有更科学更准确的方法来鉴别柴胡药材。
DNA条形码技术是近年来生物物种鉴定的新技术,陈士林等[4, 5]提出了以ITS2作为药用植物鉴定的通用序列,其稳定性已在多种药用植物的种属鉴定上得到肯定[6, 7, 8]。目前,已经有利用ITS和ITS2序列对柴胡进行鉴定的报道[9, 10, 11]。本实验是利用ITS2序列,对河北中药材市场的28份柴胡药材样品进行检测,从而为ITS2序列进入柴胡药材鉴定实践领域提供理论基础,为临床用药安全提供依据。
1 材料28份柴胡药材样本全部购自河北省中药材市场,经河北中医学院郑玉光教授鉴定,凭证样本保存于河北中医学院药学院,其余7个样本来源于GenBank。样品来源见表 1。
| 表 1 样品来源 Table 1 Sources of samples |
Eppendorf梯度PCR仪;电泳仪为北京六一仪器厂DYY-6D电泳系统;美国伯乐Bio-Rad GelDoc XR System 凝胶成像系统;DNA提取试剂盒购自全式金生物技术(北京)有限公司,引物由上海Invitrogen公司合成。
2.2 DNA提取、PCR扩增和测序取柴胡药材样本20 mg,液氮研磨,利用植物基因组提取试剂盒提取柴胡药材的全基因组DNA。对所提取的DNA的进行PCR扩增,所采用引物为通用引物ITS2,引物序列为ITS2F:5’-ATGCGATA- CTTGGTGTGAAT-3’;ITS2R:5’-GACGCTTCTCC-AGACTACAAT-3’,扩增反应体系为50 μL。反应体系:含有Taq PCR Mix Master 25 μL,正、反向引物各1.5 μL,DNA 5 μL,加ddH2O至50 μL。扩增程序:94 ℃预变性5 min,94 ℃变性30 s,56 ℃、30 s,72 ℃、45 s,40个循环,循环结束后72 ℃再延伸10 min,4 ℃保存。1%的琼脂糖凝胶电泳检测PCR反应结果,每份样品30 μL PCR产物直接测序,由深圳华大基因公司北京测序部完成。每份样品均进行3次生物学重复。
2.3 数据处理利用CodonCode Aligner软件对测序的序列进行校对拼接,去除引物及低质量区,同时利用隐马尔可夫模型的HMMer注释方法去除两端5.8 S和28 S区域[12, 13]。将所测得的序列用软件MEGA 6.0分析比对,基于K2P模型进行遗传距离的计算并建立邻接(NJ)树,根据 Schultz 等[14, 15]建立的ITS2数据库及其网站预测ITS2的二级结构。
3 结果 3.1 柴胡样品DNA提取和PCR扩增结果柴胡药材样品提取DNA后,利用ITS2引物进行PCR扩增,利用琼脂糖凝胶电泳检测PCR产物,发现28份样品均能扩增出单一明亮条带,对检测后的PCR产物进行测序。
3.2 序列的比对与提交将扩增的ITS2的序列经CodonCode Aligner软件校对拼接后,将序列在NCBI网站上进行Blast比对,结果发现28条序列中有4条序列与柴胡的序列差别很大,分别与苦参、龙芽草和石竹的序列相似度较高。重新取样、提取DNA并测序后结果仍是如此。因此认为柴胡药材中混入了其他的种类。将28条序列提交到NCBI数据库并获得登录号(表 1)。
 | 图 1 柴胡样品的PCR产物琼脂糖凝胶电泳 Fig.1 Agarose gel electrophoresis of PCR product from Bupleuri Radix |
柴胡实验样本的ITS2序列长度变化范围为173~223 bp,柴胡种间有5个变异位点,位置分别为50、102、105、154、170 bp,分为7种单倍型(表 2),说明柴胡样品的种间变异很小。而黑柴胡与柴胡相比变异位点有14个,苦参、龙牙草和石竹的序列与柴胡样本序列相比几乎完全不同(图 2)。
| 表 2 柴胡种内变异 Table 2 Intraspecific variation of Bupleuri Radix |
 | 图 2 柴胡及其混伪品种内、种间序列比对 Fig.2 Intraspecific and interspecific alignment of ITS2 sequences in Bupleuri Radix and its adulterants |
基于K2P模型计算柴胡与其混伪品K2P距离,柴胡种内K2P平均为0.004,种内最大K2P遗传距离为0.009。柴胡与其混伪品的种间遗传距离最小为0.041,远远大于柴胡的种内最大遗传距离,表明ITS2能够准确区分柴胡及其混伪品。
3.5 柴胡及其混伪品的聚类分析基于得到的28条ITS2序列,并从GenBank下载7条序列,利用NJ法构建系统的聚类树。从所构建的NJ树中可以看到柴胡序列单独聚为一支,黑柴胡单独聚为一支,从GenBank下载的柴胡伪品聚为一支,而鉴定出的苦参、龙牙草、石竹和作为对照的当归聚为一支,说明这些植物的科属与柴胡相差较远(图 3)。
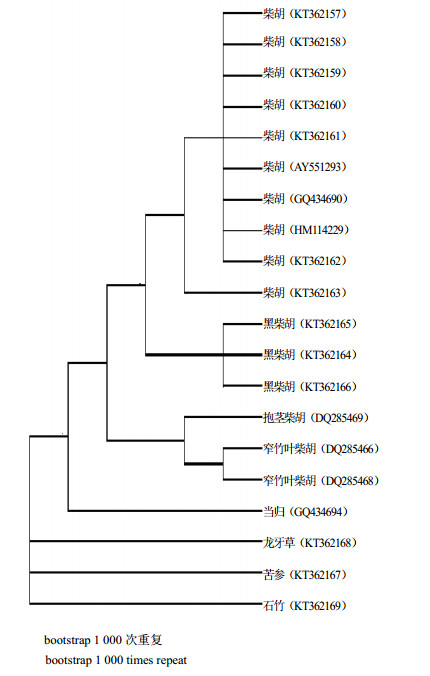 | 图 3 基于ITS序列构建柴胡及其混伪品NJ树 Fig.3 NJ dendrogram in Bupleuri Radix and its adulterants based on ITS2 sequences |
通过柴胡及其混伪品二级结构(图 4)比较可以看出,柴胡与黑柴胡的二级结构差别也是很大的,其保守区以及4个螺旋臂都有较大差异;苦参与柴胡的保守区和螺旋臂III有一些相似,但是柴胡的茎环较苦参的要大,因此二者在分子水平差异也很明显。龙牙草和石竹与柴胡在螺旋臂III稍有一些相似,但是茎环的差别很明显。因此,通过二级结构的分子形态差异可以区别柴胡及其混伪品。
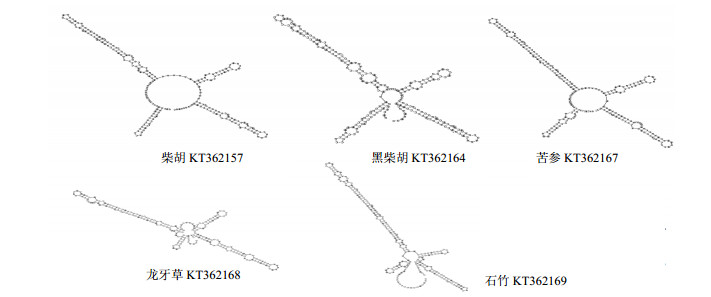 | 图 4 柴胡及其混伪品ITS2二级结构 Fig.4 Secondary structures in Bupleuri Radix and its adulterants |
我国的中药资源蕴藏量巨大,种类繁多,但是由于经济利益驱使,中药材掺假时有发生。《中国药典》2015年版中对每一种药材的鉴别方法已经达到了3种以上,DNA条形码技术也被药典列为了药材鉴定的方法之一。柴胡属作为伞形科最大的属,其作为商品流通的柴胡种类达到12种之多[16],利用传统的生药鉴定的方法已经难以满足现代物种鉴定的需求[17]。本研究证实了利用DNA条形码技术鉴定市场药材柴胡真伪的可行性。
在实验过程中,由于柴胡的药材是干燥的根,因此在DNA提取过程中应首先将其清洗干净,避免污染,其次要保证提取的DNA的浓度和纯度符合下一步PCR反应的要求。通过延长提取步骤中的水浴的时间,使干燥的柴胡根细胞充分裂解,从而达到提取较高浓度DNA的要求。本实验中涉及的28种柴胡的药材利用通用ITS2引物均有很好的扩增效率,PCR产物电泳呈现明亮单一的条带。基于K2P距离建立的NJ树以及二级结构的预测都能够很好的将柴胡及其伪品区分开来。28份市场药材中正品21份,伪品7份,正品率为78.6%,需要提及的是,伪品中检测出了苦参、龙芽草和石竹,在NJ树中和作为对照的当归聚为一支。苦参、龙芽草和石竹与柴胡在植物学上的亲缘关系较远,在原植物形态是非常容易辨认的,但是在药材中,如果没有丰富的药材辨认经验是不容易将三者区分开的,而且这三者的药性相差甚远,一旦作为柴胡入药,严重威胁到临床用药的安全。
综上所述,利用ITS2序列能够准确鉴定柴胡药材及其伪品,本实验所鉴定的28份柴胡药材与药材鉴定专家鉴定的结果一致,说明了利用DNA条形码技术能够准确、快速地完成中药材鉴定工作,在今后的药材的流通以及市场管理、临床用药等实际应用中具有极大的价值。
| [1] | Xu N, Shi Y N, Zhong X, et al. A new saikongenin from the roots of Bupleurum bicaule[J]. Chin J Nat Med, 2014, 12(4):305-308. |
| [2] | 中国药典.[S]. 一部. 2015. |
| [3] | 雷天莉, 任宇豪, 李敬, 等. 北柴胡地上茎不同分枝对根中皂苷类成分量及根产量的影响[J]. 中草药, 2014, 45(13):1920-1923. |
| [4] | 陈士林. 中药DNA条形码分子鉴定[M]. 北京:人民卫生出版社, 2012. |
| [5] | Chen S L, Yao H, Han J P, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS One, 2010, 5(1):e8613. |
| [6] | Chen X C, Liao B S, Song J Y, et al. A fast SNP identification and analysis of intraspecific variation in the medicinal Panax species based on DNA barcoding[J]. Gene, 2013, 530(1):39-43. |
| [7] | Gu W, Song J Y, Cao Y, et al. Application of the ITS2 region for barcoding medicinal plant of Selaginellaceae in Pteridophyta[J]. PLoS One, 2013, 8(6):e67818. |
| [8] | Hou D Y, Song J Y, Yao H, et al. Molecular identification of Corni Fructus and its adulterants by ITS/ITS2 sequences[J]. Chin J Nat Med, 2013, 11(2):121-127. |
| [9] | 南小洁, 郝媛媛, 赵艮贵, 等. 柴胡药材干根DNA提取及RAPD分析[J]. 中草药, 2009, 40(3):447-451. |
| [10] | 周琳, 迟莹, 张丽华, 等. 柴胡与四种伪品的DNA指纹研究[J]. 北华大学学报:自然科学版, 2013, 14(1):46-49. |
| [11] | 迟莹, 周琳, 张丽华, 等. 柴胡及其常见伪品的DNA指纹鉴定[J]. 中国实验方剂学杂志, 2013, 19(15):143-146. |
| [12] | Keller A, Schleicher T, Schultz J, et al. 5.8s-28s rRNA interaction and HMM-based ITS2 annotation[J]. Gene, 2009, 430(1/2):50-57. |
| [13] | Schultz J, Muller T, Achtziger M, et al. The internal transcribed spacer 2 database-a web serner for (not only) low level phylogenrtic analyses[J]. Nucl Acids Res, 2006, 34(3):704-707. |
| [14] | Selig C, Wolf M, Müller T, Dandekar T, et al. Homology modelling RNA structure for molecular systematics[J]. Nucl Acids Res, 2008, 36(12):377-380. |
| [15] | Koetschan C, Förster F, Keller A, et al. The ITS2 Database Ⅲ-sequences and structures for phylogeny[J]. Nucl Acids Res, 2010, 38(6):275-279. |
| [16] | 赵香妍, 刘长利, 薛文峰, 等. 北京地区野生柴胡种质资源的ISSR研究[J]. 中国现代中药, 2015, 17(10):1008-1013. |
| [17] | 刘美子, 宋经元, 罗焜, 等. DNA条形码序列对9种蒿属药用植物的鉴定[J]. 中草药, 2012, 43(7):1393-1396. |
 2016, Vol. 47
2016, Vol. 47


