2. 福建农林大学园林学院, 福建 福州 350002
2. College of Landscape, Fujian Agriculture and Forestry University, Fuzhou 350002, China
草珊瑚具有重要的药用价值和经济价值,是一种重要的植物研究材料。但是,对于草珊瑚基因组学及生长发育的调节路径的研究鲜见报道。最早对于全长cDNA文库的研究及应用出现在1994年[1],随着分子生物技术的发展,对于全长cDNA文库构建的方法越来越多,目前常用的主要有Oligo-capping法[2]、Capture法[3]、SMART法[4-5]、CapSlect法[6]、Cap-trapper法[7-8]、Cap-jumping法[9]以及mRNA差减/均一化法[10-11]等。这些全长cDNA文库构建的方法都是围绕真核生物的mRNA 5’端CAP结构进行的。虽然全长文库构建方法越来越多,但每种构建的方法都有其缺点,致使构建全长cDNA文库仍然存在不小的阻碍[12],如mRNA的完整程度,建库过程中的载体连接、小片段cDNA的聚集、反转录等问题都会导致无法构建较高质量的cDNA文库[13]。
表达序列标签(expressed sequence tag,EST)是在已构建完成的cDNA文库中随机挑选克隆组,并在5’端或3’端进行单向的测序,进而得到一段cDNA的序列数据,cDNA的任何区域的序列都可以用来测定,区域的长度在150~500 bp,EST技术的发展极大地推进了生物基因组水平研究的速度,Adams等[14]运用EST技术分析了人脑组织进行相关方面的研究。由于cDNA是由mRNA逆转录而合成的,而mRNA是由5’-UTR、3’-UTR、ORF、poly(A)尾巴4个部分组成,且mRNA的5’-UTR和3’-UTR都具有特异性,所以一个EST序列可以看作是生物的组织或细胞在某个特定时期内的一个表达基因。EST分子标记技术经常被用于遗传学图谱的构建[15]、新基因分离和鉴定[16]、基因表达谱的研究[17]、比较基因组学的研究及基因芯片的制备[18]等方面,多种应用的结合可以分析种内的遗传差异性、生物的进化联系,物种的多样性以及系统发育学。
草珊瑚Sarcandra glabra(Thunb.) Nakai为金栗兰科(Chloranthaceae)草珊瑚属SarcandraGardn.的植物,俗名草珊瑚[19]。草珊瑚在现代药理学研究中应用广泛,研究证明草珊瑚具有抗菌、抗溃疡、消炎和镇痛、促进骨折愈合、抑制流感病毒、保肝、提高免疫力、抗肿瘤等作用[20-22]。对于草珊瑚倍半萜类、有机酸类、香豆素类等化学成分已有较为系统、全面的研究[23-25]。但是,对于草珊瑚基因组学的研究鲜有报道,主要是对草珊瑚遗传种质资源多样性的研究[26-28]。在NCBI数据库中检索草珊瑚相关分子信息,未得到任何核酸序列及EST序列,同时检索到119条蛋白序列以及0条基因组。这表明对于草珊瑚在深层次的分子克隆、代谢途径及蛋白功能方面有待于进一步探究。本研究通过选取最适宜的提取方法来制备草珊瑚总RNA,再通过SMART法构建草珊瑚叶片全长cDNA文库及EST序列分析,进一步完善草珊瑚有关核酸序列、EST数据库及有效成分生物合成的功能基因的基因资源,为更深层次的草珊瑚的分子生物理论研究以及草珊瑚药用价值的基因功能提供实验基础。
1 材料与方法 1.1 材料材料取自福建农林大学工业原料林研究所苗圃,经福建农林大学林学院邹小兴教授鉴定为金栗兰科药材草珊瑚Sarcandra glabra(Thunb.) Nakai的全草。所用草珊瑚嫩叶用超纯水擦拭叶片后迅速置于液氮中速冻,然后贮存至−80 ℃冰箱中备用。
1.2 RNA的提取以及mRNA的分离采用CTAB法[29]、CTAB-Trizol法[30]、Trizol法[31]、CTAB-LiCl法[32]、RNA试剂盒法1、RNA试剂盒法2共6种方法来提取草珊瑚叶片中的总RNA,通过RNA纯化在1%~1.2%的琼脂糖凝胶上电泳检测完整性质量后[33],对符合要求的RNA进行mRNA的提取以及全长cDNA文库的构建。
1.3 cDNA文库的构建及测序对草珊瑚mRNA纯化和质检后,通过SMART法[33]构建草珊瑚cDNA全长文库。采用Clontech公司的In-Fusion SMARTer Directional cDNA文库构建试剂盒(Cat.No.634933)、第二链合成的Advantage 2 PCR试剂盒kit(Cat.No.639207)以及TaKaRa公司的电转感受态细胞DH5α,进行全长文库的构建。电转后对原始文库进行文库质量检测,包括文库滴度的测定、重组率的测定以及插入片段检测。通过检测后PCR反应后,对大于500 bp的阳性单克隆在ABI3730 DNA序列分析仪上进行EST测序。
1.4 EST序列数据分析通过DNAMAN软件以及Chromas软件结合的方法,对EST测序得到的序列去除载体、不准确的碱基以及低质量的序列,从而获得比较准确的EST片段[34]。获得EST片段后利用DNAStar软件对EST片段进行聚类拼接成重叠群(contigs)和单一序列(singlets)的单拷贝序列(unique sequences),并提交到GenBank数据库。
利用NCBI的BLAST程序对拼接后的单拷贝序列与核酸库(NT)和非冗余数据库(NR)进行比对,完成BLASTN及BLASTX的同源性比对,若BLASTN及BLASTX无法检索结果的,再用EST-others数据库比对,寻找相似性EST片段,比对时设置E值>1×10−5[35]。用BLAST2GO软件对EST序列数据进行分子功能、细胞成分以及生物过程等方面的功能分类注释[36]。利用KEGG数据库的KO标签(http://www.genome.jp/kaas-bin/kaas_main
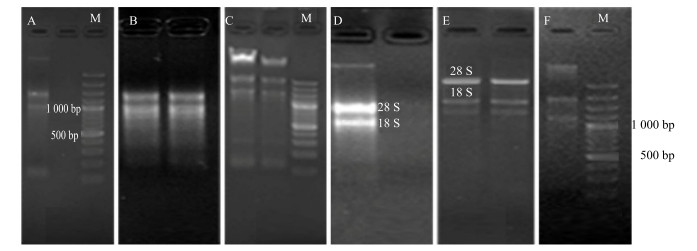
2 结果与分析 2.1 RNA的制备在NanoDrop2000c上测定6种不同草珊瑚RNA提取方法的总RNA在230、260、280 nm下的吸光度(A),得到的A260/A280、A260/A230值及总RNA的浓度(表 1),通过琼脂糖凝胶电泳检测RNA的完整性(图 1)。总体来说,CTAB-LiCl法和RNA试剂盒1 2种方法提取的RNA均符合要求,但试剂盒法得到的总RNA量及成功率较低。通过多方面比较采用CTAB-LiCl法提取草珊瑚总RNA来构建cDNA文库。CTAB-LiCl法有轻微DNA污染,需进一步消除DNA的干扰。
|
|
表 1 6种方法提取的草珊瑚叶片总RNA样品的纯度与浓度 Table 1 Comparison on purity and yield of total RNA in S. glabra by six extraction methods |
|
M-Marker A-CTAB法B-CTAB-Trizol法C-Trizol法D-CTAB-LiCl法E-RNA试剂盒法1 F-RNA试剂盒法2 M-Marker A-CTAB method B-CTAB-Trizol method C-Trizol method D-CTAB-LiCl method E-RNAprep Pure Plant Kit 1 F-RNAprep Pure Plant Kit 2 图 1 6种不同方法提取的草珊瑚叶片总RNA电泳检测结果 Fig.1 Electrophorogram of extracted total RNA from leaves of S. glabra by six different methods |
2.2 草珊瑚全长cDNA文库的构建及测序
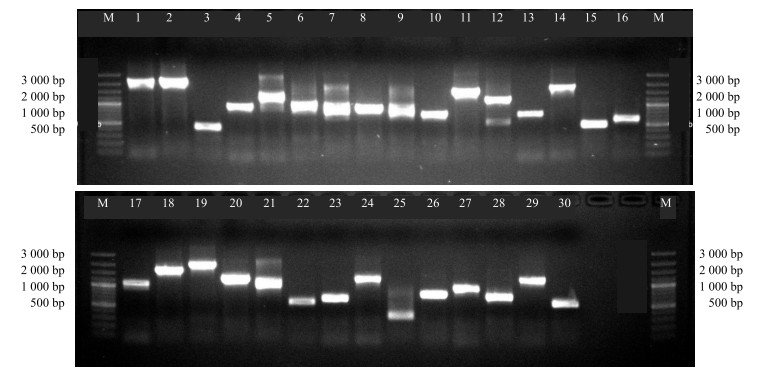
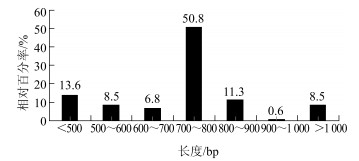
构建了以草珊瑚叶片为材料的全长cDNA文库,重组率为81.4%,文库滴度1.14×107 cfu/mL,插入的片段大小在500~3 000 bp,平均为1 000 bp,说明构建的文库包含了相对较长的cDNA(图 2)。随机挑取351个单克隆,经PCR检测去除不合格的克隆,提交共221个单克隆送铂尚公司测序,共获得有效序列177条,核糖核苷酸的G+C的总量为41.9%,说明草珊瑚叶片表达基因序列G+C的量接近于双子叶植物拟南芥(40.7%)而明显低于单子叶植物水稻(53.4%),EST序列长度主要集中在700~800 bp(图 3)。通过DNAstar软件对177条序列聚类并对同一重叠群进行拼接,共获得单一序列(unigene)151条,包含有12个EST重叠群(contig)及139条单序列(singleton),即非重复的唯一序列。文库的冗余度为14.7%,表明文库具有良好的代表性。
|
1~30-随机30个草珊瑚样本 1—30-S. glabra samples 图 2 全长cDNA文库插入片段大小 Fig.2 Average size of inserts fragments of cDNA library |
|
图 3 ESTs长度分布 Fig.3 Distribution of ESTs length |
2.3 EST序列注释及生物信息学分析
利用NCBI的BLASTX及BLASTN程序对拼接后的单序列与NR数据库进行同源性比对和基因功能注释。151条单一序列中有32条(21%)无显著的同源性,是新序列;119条(79%)有显著同源性,包括24条已知功能的蛋白和91条的预测蛋白,而未知蛋白有4条(3%),其余21%的EST为功能未知基因或新基因序列,但是这些值只是暂定的,已知功能EST序列数量必定会随着数据库中信息的增加而提高,这也从一定程度上反映出草珊瑚的研究还有待深入。
实验共获得具有KEGG注释的序列50条,根据BRITE功能等级分类得出结果:参与代谢相关的序列为39条,遗传信息加工的序列为19条,参与环境信息加工的序列为10条,参与细胞过程的序列有11条,参与生物体系统序列有25条,与人类疾病相关的序列有32条,一共126条,这说明获得注释的序列参与的过程不止一个。
将上述已知的和预测的119条有显著同源性的单一序列在BLAST2GO中进行功能学的分类,共获得了GO术语415个,每条序列平均有3.49个GO术语且归结为3类,其中生物学过程(biological process)185个,GO,术语占44.85%;分子功能(molecular function)104个GO术语占25.06%;细胞组件(cellular component)126个,GO术语占30.36%。在这119个有功能注释的单一序列中,其功能涉及细胞生长、信号转导、蛋白质合成、转录、抗逆反应以及能量代谢等(表 2),其中参与蛋白质合成和能量代谢的基因所占比例较高。
|
|
表 2 部分与已知功能基因同源的草珊瑚叶片ESTs Table 2 Some ESTs matched with known functional genes in S. glabra leaves |
3 讨论 3.1 RNA提取
草珊瑚叶片是多年生亚灌木的组织,富含多酚、多糖及其他次级代谢产物。黄酮类为草珊瑚的主要有效成分。本实验采用CTAB-LiCl法提取草珊瑚叶片总RNA,匀浆阶段加入聚乙烯吡咯烷酮(PVP)和β-巯基乙醇从而将酚类化合物去除;LiCl能选择性地沉淀高相对分子质量的RNA,可以去除大部分DNA的干扰,但有时还会有轻微DNA污染需要进一步去除和纯化;其他方法:试剂盒法1得到的总RNA量及成功率较低,不宜采用;CTAB法提取草珊瑚叶片总RNA的步骤中,没有有效去除DNA的步骤,易受DNA的污染;Trizol法是即用型试剂,主要成分有苯酚、8-羟基喹啉、异硫氰酸胍。苯酚既可裂解细胞,使细胞中的蛋白、核酸解聚而被释放,也可使蛋白质变性,但不能完全抑制RNA酶活性,可通过8-羟基喹啉、异硫氰酸胍、β-巯基乙醇等来抑制内外源RNA酶。虽然Trizol法在提取许多动植物组织或细胞中成功应用,但是不适合对多糖、多酚类物质的提取;CTAB-Trizol法在CTAB法的基础上,加入Trizol来去除DNA的污染,但容易在点样孔周围有蛋白残留,并且总RNA完整性不高,不适合提取草珊瑚叶片总RNA。
3.2 cDNA文库构建及质量评价实验采用Clontech公司SMART法文库构建试剂盒,方法操作简便易懂。在RNA进行反转录时,采用草珊瑚叶片总RNA,省去了纯化mRNA的步骤,并且可以避免在纯化mRNA过程中RNA的降解和低丰度基因的丢失,从而保证了反转录后cDNA的完整性和获得全长cDNA的可能性[37]。与常见的一步反转录不同的是,用两步法完成反转录,即72 ℃温育3 min的热变性处理,再加入反转录酶完成反转录过程,防止因RNA链过长或存在复杂的二级结构,从而使反转录酶提前终止反应,无法获得丰富的全长cDNA。dscDNA链的合成采用LD-PCR的方法,合成的双链cDNA经检测符合试验要求。
在反转录过程中以加入的Oligo(dT)的起始引物,3’末端带Oligo(dG)的SMART引物,逆转录酶及总RNA为主要合成物质。合成得到的所有cDNA单链的一端有含Oligo(dT)的起始引物序列,另一端有已知的SMART引物序列,这样合成第二链后不仅可以利用通用引物进行扩增,而且得到的cDNA能够代表原有样品中mRNA的丰度,接下来也可以应用于直接扩增基因、构建cDNA文库、RACE技术及应用于芯片检测的cDNA探针技术。
通过实验得出影响文库质量的关键因素第二链合成的最佳起始浓度、最佳循环数、cDNA片段与载体的最佳连接比例等。对于文库质量的评价方面,通过滴度检测、重组率、PCR片段检测3方面来确定。文库滴度检测能反映文库的完整性、覆盖度;重组率能通过蓝白斑的比例间接确定重组片段的导入情况,有一定的误差存在;PCR片段检测能准确直观地判断重组片段的大小。本实验没有扩增出大于3 kb以上的大片段,这是因为PCR程序的选择性不利于大片段基因的扩增,是SMART法的一个缺陷,针对这一问题后来有了相对的改进方法:(1)将PCR扩增的循环数由18减到3的Little-cycle SMART技术[38];(2)Super SMART技术;(3)以及为了获取大于3 kb的基因片段的Large-size SMART技术[5]。
生物信息学处理能够为药用植物高通量测序产生的大量生物数据进行资源整合、分析和利用,从而构建中药数据库,从中挑取与中药有效成分产生有关的基因、蛋白质进行研究。用传统的EST方法获取转录组数据,与基因组NCBI dbEST数据库通过BLAST软件进行序列比对找出同源序列,与Blast2GO、KEGG数据库比对进行基因功能注释和代谢通路分析。随着高通量测序的发展,RNA-seq在检测基因表达和转录组研究方面显示出优越的特性,其中丹参、红豆杉、甘草等药用模式植物转录组已经测序并发表,对研究次生代谢物生物合成的途径解析和关键基因的挖掘具有促进作用。今后对草珊瑚的各组织也可以通过RNA-seq技术探索控制草珊瑚中次生代谢物黄酮类与香豆素类的异嗪皮啶生物合成的具体途径和关键基因。
| [1] | Maruyama K, Sugano S. Oligo-capping:a simple method to replace the cap structure of eukaryotic mRNAs with oligoribonucleotides[J]. Gene , 1994, 138 (1/2) :171–174. |
| [2] | Suzuki Y, Sugano S. Construction of full-length-enriched cDNA libraries. The oligo-capping method[J]. Methods Mol Biol , 2001, 175 (5) :143–153. |
| [3] | Zhumabayeva B, Chenchik A, Siebert P D. RecA-mediated affinity capture:a method for full-length cDNA cloning[J]. Biol Techn , 1999, 27 (4) :834–840. |
| [4] | Dai Z, Wang B, Liu Y, et al. Producing aglycons of ginsenosides in bakers' yeast[J]. Sci Rep , 2014, 4 (4) :3698. |
| [5] | Wellenreuther R, Schupp I, Poustka A, et al. SMART amplification combined with cDNA size fractionation in order to obtain large full-length clones[J]. BMC Genomics , 2004, 5 (1) :1–8. DOI:10.1186/1471-2164-5-1 |
| [6] | Schmidt W M, Mueller M W. CapSelect:a highly sensitive method for 5'CAP-dependent enrichment of full-length cDNA in PCR-mediated analysis of mRNAs[J]. Nucl Acids Res , 1999, 27 (21) :e31. DOI:10.1093/nar/27.21.e31 |
| [7] | Yuichi S, Piero C, Masayoshi I, et al. Comparative evaluation of 5'-end-sequence quality of clones in CAP trapper and other full-length-cDNA libraries[J]. Gene , 2001, 263 (1) :93–102. |
| [8] | Carninci P, Kvam C, Kitamura A, et al. High-efficiency full-length cDNA cloning by biotinylated CAP trapper[J]. Genomics , 1996, 37 (3) :327–336. DOI:10.1006/geno.1996.0567 |
| [9] | Efimov V A, Chakhmakhcheva O G, Archdeacon J, et al. Detection of the 5'-cap structure of messenger RNAs with the use of the cap-jumping approach[J]. Nucl Acids Res , 2001, 29 (22) :4751–4759. DOI:10.1093/nar/29.22.4751 |
| [10] | Zhang Z X, Zhang F D, Tang W H, et al. Construction and characterization of normalized cDNA library of maize inbred MO17 from multiple tissues and developmental stages[J]. Mol Biol , 2005, 39 (2) :177–184. DOI:10.1007/s11008-005-0026-8 |
| [11] | Zhulidov P A, Bogdanova E A, Shcheglov A S, et al. A method for the preparation of normalized cDNA libraries enriched with full-length sequences[J]. Russ J Bioorg Chem , 2005, 31 (2) :170–177. DOI:10.1007/s11171-005-0023-7 |
| [12] | 董志敏, 张宝石, 关荣霞, 等. 全长cDNA文库的构建方法[J]. 中国农学通报 , 2006, 22 (2) :51–55. |
| [13] | 赵光耀.小麦全长cDNA文库构建、测序与分析[D].北京:中国农业科学院, 2006. |
| [14] | Adams M D, Kelley J M, Gocayne J D, et al. Complementary DNA sequencing:expressed sequence tags and human genome project[J]. Science , 1991, 252 (5013) :1651–1656. DOI:10.1126/science.2047873 |
| [15] | 张利达, 唐克轩. 植物EST-SSR标记开发及其应用[J]. 基因组学与应用生物学 , 2010, 29 (3) :534–541. |
| [16] | 崔永兰.石蒜属植物cDNA文库构建与表达序列标签(EST)分析[D].南京:南京林业大学, 2004. http://cdmd.cnki.com.cn/Article/CDMD-10298-2004092387.htm |
| [17] | 李伍举. 基因表达谱的生物信息学[J]. 军事医学科学院院刊 , 2002, 26 (1) :73–76. |
| [18] | 陈全求, 詹先进, 蓝家样, 等. EST分子标记在基因组学中应用的研究进展[J]. 中国农学通报 , 2010, 26 (3) :59–63. |
| [19] | 中国科学院中国植物志编辑委员会. 中国植物志(二十卷第一分册)[M]. 北京: 科学出版社, 1982 . |
| [20] | 中国药典[S].一部. 2010. |
| [21] | Cao H J, Tan R R, He R R, et al. Sarcandra glabra extract reduces the susceptibility and severity of influenza in restraint-stressed mice[J]. Evid Based Complement Alternat Med , 2012, 2012 :236539. |
| [22] | Li Y, Zhang D M, Li J B, et al. Hepatoprotective sesquiterpene glycosides from Sarcandra glabra[J]. J Nat Prod , 2006, 69 (4) :616–620. DOI:10.1021/np050480d |
| [23] | 胡晓茹.草珊瑚的化学成分研究[D].北京:中国协和医科大学, 2009. http://cdmd.cnki.com.cn/Article/CDMD-10023-2009150805.htm |
| [24] | 邹小燕.草珊瑚化学成分的研究[D].沈阳:沈阳药科大学, 2006. http://cdmd.cnki.com.cn/Article/CDMD-10163-2006177536.htm |
| [25] | 邵佳.草珊瑚总黄酮提取纯化及药理作用研究[D].贵阳:贵州大学, 2008. http://cdmd.cnki.com.cn/Article/CDMD-10657-2008205141.htm |
| [26] | 魏艺聪, 林培玲, 陈莹, 等. 采用ISSR标记分析草珊瑚的DNA指纹图谱与其品质相关性[J]. 中草药 , 2014, 45 (11) :1620–1624. |
| [27] | 唐美琼, 韦荣昌, 姚绍嫦, 等. 广西草珊瑚种质资源遗传多样性的AFLP分析[J]. 中草药 , 2012, 43 (7) :1398–1402. |
| [28] | Xu Y, Wei G Y, Zhou Y, et al. A set of novel microsatellite markers developed for a traditional Chinese medicinal plant, Sarcandra glabra (Chloranthaceae)[J]. J Genet , 2014, 93 (2) :e86–e88. |
| [29] | Jamalnasir H, Wagiran A, Shaharuddin N A, et al. Isolation of high quality RNA from plant rich in flavonoids, Melastoma decemfidum Roxb ex. Jack[J]. Austr J Crop Sci , 2013, 7 (7) :911–916. |
| [30] | 赵锦, 刘中成, 代丽, 等. 枣不同器官和组织RNA提取方法的研究[J]. 植物遗传资源学报 , 2009, 10 (1) :111–117. |
| [31] | King L C, Tsang W H, Lee J T Y. Simple modifications to standard TRIzol® protocol allow high-yield RNA extraction from cells on resorbable materials[J]. J Biomater Nanobiotechnol , 2011, 2 (1) :41–48. DOI:10.4236/jbnb.2011.21006 |
| [32] | 宋蓓, 赵锦, 刘孟军, 等. 改良CTAB-LiCl法提取枣总RNA体系的建立[J]. 中国农学通报 , 2007, 7 (23) :79–83. |
| [33] | Zhang D L, Hu C G, Ouyang Y D, et al. Construction of a full-length cDNA library and analysis of expressed sequence tags from inflorescence of apomictic sabaigrass (Eulaliopsis binata)[J]. Plant Mol Biol Rep , 2012, 30 (1) :46–54. DOI:10.1007/s11105-011-0316-3 |
| [34] | Gordon D, Abanjian C, Green P. Consed:A graphical tool for sequence finishing[J]. Genome Res , 1998, 8 :195–202. DOI:10.1101/gr.8.3.195 |
| [35] | 龚达平, 解敏敏, 孙玉合. 烟草叶片全长cDNA文库构建及EST序列分析[J]. 中国农业科学 , 2012, 45 (9) :1696–1702. |
| [36] | Conesa A, Gotz S, García-Gómez J M, et al. BLAST2GO:a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics , 2005, 21 (18) :3674–3676. DOI:10.1093/bioinformatics/bti610 |
| [37] | 赵桂媛, 魏志刚, 刘关君, 等. SMART策略构建小黑杨茎形成层全长cDNA文库[J]. 北京林业大学学报 , 2010, 32 (1) :52–56. |
| [38] | Zhu Y Y, Machleder E M, Chenchik A, et al. Reverse transcriptase template switching:A SMART approach for full-length cDNA library construction[J]. Biotechniques , 2001, 30 (4) :892–897. |
 2016, Vol. 47
2016, Vol. 47


