2. 广东医学院, 广东 湛江 524023 ;
3. 广州中医药大学中药学院, 广东 广州 510006
2. Guangdong Medical University, Zhanjiang 524023, China ;
3. School of Chinese Herbal Medicine, Guangzhou University of Traditional Chinese Medicine, Guangzhou 510006, China
芦荟是芦荟属Aloe L. 植物的通称,来源于单子叶植物纲百合目百合科多年生肉质草本植物。芦荟性苦、味寒,归肝、胃、大肠经,具有泻下通便、清肝泻火、杀虫疗疳的功能[1],临床上主要用于治疗热结便秘、惊痫抽搐、小儿疳积等。除此之外,芦荟还是许多美容化妆品、保健食品和药品等的重要原料。
芦荟原产于地中海、非洲,我国南方各地均有栽培,广泛用于观赏或药用。芦荟属种类繁多,各物种形态特征相近,其茎、叶形态受遗传控制和环境影响且存在动态变化[2],同时存在很多变种。利用传统的基原鉴定和性状鉴定难以准确快速地进行鉴别,因此,有必要建立一种快速、准确鉴别芦荟属物种的新方法。
DNA条形码是当前生物分类学的热门技术,是利用一段有足够变异的、易扩增且相对较短的标准DNA片段(500~1 000 bp)对物种进行快速、准确的自动鉴定[3]。2009年,生命条形码联盟植物工作组建议将rbcL和matK基因片段作为植物的核心条形码,叶绿体psbA-trnH片段和核基因ITS片段为补充条形码[4]。Chen等[5]通过分析6 600多种植物,发现ITS2序列具有较高的扩增效率,且在物种水平的鉴定成功率高于90%,提议将ITS2序列作为药用植物的通用条形码。目前,DNA条形码技术已经广泛应用于药用植物的鉴别研究[6-7]。而对芦荟的研究主要集中在芦荟的化学成分[8-9]、临床药理[10-11]、开发利用[12-13]等方面,尚未见有芦荟属植物DNA条形码研究的相关报道。
本研究选用4条热门DNA条形码序列对芦荟属6种植物进行鉴别,比较不同候选片段对该属物种的适用性和鉴别能力,以筛选能鉴别该属植物的优选DNA条形码序列,为该属植物的分子鉴定提供参考依据。
1 材料与方法 1.1 材料本研究所选用的芦荟属6个品种均采自广州中医药大学药王山,每个品种取3株不同植株的样本,具体样品信息见表 1。样品由广州中医药大学中药资源科学与工程研究中心詹若挺研究员和广东中医药博物馆张秋镇助理研究员协助采集并鉴定,凭证标本保存于广州中医药大学岭南中药资源教育部重点实验室。
|
|
表 1 材料信息表 Table 1 Information of experimental materials |
1.2 仪器与试剂 1.2.1 仪器
电泳仪(DYY-8C型),北京市六一仪器厂;电子天平(METTLER TOLEDO ME204),梅特勒托利多仪器(上海)有限公司;微量分光光度计(TGem Spectrophotometer OSE-260),天根生化科技(北京)有限公司;PCR仪(Bio-Rad PTC-200),美国;凝胶成像系统(Vilber Lourmat,INFINITY-1000),法国;掌上离心机(Eppendorf Minispin),德国;漩涡混匀器(Labnet,Vortex Mixter),美国。
1.2.2 试剂植物基因组DNA提取试剂盒(Tiangen Biotech Co.,中国)、Premix TaqTM(Ex TaqTM Version 2.0,Takara Co.,中国)、琼脂糖(Biowest,西班牙)、灭菌水、液氮、氯仿、无水乙醇、Goldview核酸染料。
1.3 方法 1.3.1 样品DNA的提取取硅胶干燥过的植物叶片约10 mg,经液氮研磨后,使用植物基因组DNA提取试剂盒(Tiangen Biotech Co.,中国)提取样品DNA,使用TGem Spectrophotometer微量分光光度计(Tiangen Biotech Co.,中国)对提取的DNA进行纯度和浓度测定。
1.3.2 序列扩增不同DNA条形码序列的通用引物、PCR反应体系及扩增程序均参考文献方法[14],实际操作时扩增条件略有调整,具体引物序列及反应条件见表 2。25 μL PCR反应体系包括:Premix Taq酶(Takara Co.,中国)12.5 μL,上游和下游引物各0.5 μL,DNA模板0.5 μL,ddH2O补齐至25 μL。扩增结束后进行2%琼脂糖凝胶电泳检测,并使用凝胶成像系统分析。
|
|
表 2 实验用引物及反应条件 Table 2 Universal primer sequences and PCR reaction conditions |
1.3.3 PCR产物测序及序列数据处理
PCR扩增成功后,各样品交由华大基因科技(广州)有限公司纯化,使用ABI 3730XL测序仪(Applied Biosystems Co.,USA)进行双向测序。测序所得的峰图采用序列拼接软件Sequencher 4.1.4进行校对拼接,去除引物区和低质量的序列。根据Genbank上序列注释,其中psbA-trnH去除psbA和trnH基因区段获得psbA-trnH间隔区[15],ITS2基于隐马尔可夫模型(Hidden Markov model,HMM)去除5.8 S和28 S基因区段获得ITS2间隔区[16-17]。运用MEGA 5.0软件对拼接后序列进行比对分析,基于Kimura 2-Parameter(K2P)双参数模型和邻位连接法(neighbor-joining,NJ),计算遗传距离,构建条形码距离(barcoding gap)图和系统发育树,系统树各分支的相对支持率采用Bootstrap法(BS)检测,自展系数为1 000。
2 结果与分析 2.1 DNA提取、PCR扩增及序列拼接按照试剂盒操作步骤,分别提取18个样品的基因组DNA,经微量分光光度计测定,A260/A280的值在1.62~2.03,表明DNA纯度较好,各样品DNA的浓度在25.28~453.76 ng/μL,表明DNA质量浓度较高,符合后续实验要求。对提取的DNA进行电泳检测,结果见图 1,条带较完整清晰。以通用引物对ITS2、psbA-trnH、rbcL、matK 4条序列进行PCR扩增,电泳结果见图 2,18个样品条带明亮单一,扩增成功率均为100%,可用于后续的测序分析。以碱基质量Q值(quality value)大于20(碱基读取错误的概率≤1%)为标准,对符合标准的测序序列进行拼接和校对。

|
图 1 样品DNA 提取产物电泳图 Fig.1 Electrophoregram of genomic DNA extracted from all samples |
|
图 2 样品4 条序列的PCR 扩增产物电泳图 Fig.2 Electrophoregram of PCR products of four barcode sequences |
2.2 候选序列的特征
如表 3所示,本研究共获得ITS2、psbA-trnH、rbcL和matK序列各18条,序列的测序成功率均为100%。不同候选序列在序列长度、GC量上有较明显的差异。比对长度均符合理想条形码序列需尽可能短(500~1 000 bp)的选择标准,比对序列长度由高到低依次为matK、psbA-trnH、rbcL、ITS2,GC量则排序相反。候选序列中ITS2序列变异最大,占总位点的4.3%,rbcL序列变异位点数最少,占总位点的1.7%,psbA-trnH序列和matK序列变异位点数相近,分别占总位点的2.8%和2.5%。
|
|
表 3 不同候选序列在芦荟属植物中的特点 Table 3 Sequence characteristics of different candidate sequences in species of Aloe L. |
2.3 候选序列的种内、种间变异分析
本研究采用MEGA 5.0软件对芦荟属不同物种进行样品间的遗传距离分析,K2P遗传距离结果表明,在ITS2、psbA-trnH、rbcL和matK序列中,所有样品的平均遗传距离分别为0.020、0.011、0.006、0.009,种间K2P遗传距离分布于0~0.040(ITS2)、0.002~0.020(psbA-trnH)、0~0.011(rbcL)、0~0.018(matK),ITS2序列的种间差异最大,psbA-trnH和matK序列次之,rbcL序列最小。ITS2序列最大种间遗传距离是0.040,分别位于皂质芦荟(80)和库拉索芦荟(83)、海岸芦荟(81)和库拉索芦荟(83)之间,psbA-trnH序列的种内最大变异小于种间最小变异,较为理想。
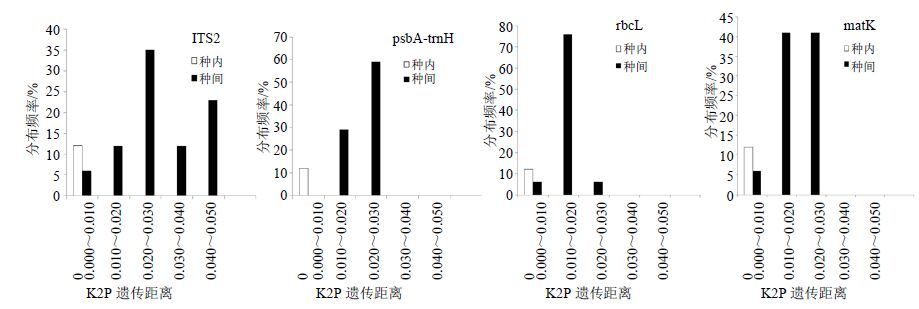
2.4 不同序列变异性的barcoding gap评估不同候选序列的barcoding gap见图 3,ITS2序列种间变异最大,rbcL和matK序列种间变异水平相对较低,ITS2、rbcL和matK序列的种内和种间变异均有重叠,没有明显的barcoding gap,psbA-trnH序列变异程度不是很高,但是序列种间和种内变异没有重叠,明显分为两大部分,有显著的barcoding gap。
|
图 3 不同候选序列的barcoding gap 图 Fig.3 Distribution for intra- and inter-specific genetic variation of different candidate sequences |
2.5 候选序列的物种鉴定有效性评价
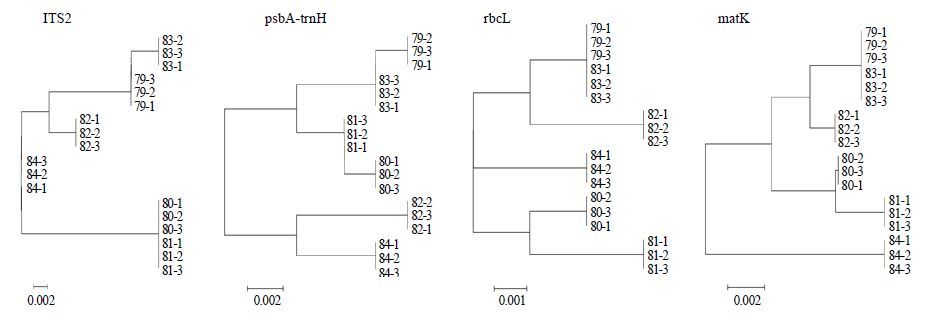
使用MEGA 5.0软件对测序结果进行分析并建立DNA条形码的系统发育树。在采用NJ法构建系统发育树时,各个序列的建树效果都较好,基本上能将不同物种分开,结果见图 4。psbA-trnH序列能将芦荟属的6个品种准确区分,其所构建的系统树各个品种不同的样本均分别聚在一起,表现出单系性。ITS2、rbcL和matK序列对其中4个品种鉴定效果良好,ITS2序列不能区分皂质芦荟(80)和海岸芦荟(81),而rbcL和matK序列不能区分中华芦荟(79)和库拉索芦荟(83)。因此,psbA-trnH序列在芦荟属6个品种的鉴定有效性方面聚类效果最好。
|
图 4 不同序列基于NJ 法构建的系统发育树图 Fig.4 Phylogenetic trees of different candidate sequences constructed using NJ method |
3 讨论
迄今国内外对芦荟属植物鉴别的报道较少,仅见少数利用RAPD技术进行芦荟遗传稳定性的研究[18-20]。RAPD分子标记实验可重复性较差不利于实际操作,而DNA条形码技术可重复性高、操作简单、易于标准化,无疑是对传统形态鉴定学的有力补充,得到了许多专家学者的肯定[21]。
DNA条形码技术是基于分子生药学进行中药鉴定的重要工具,具有足够的稳定性和准确度,能构建丰富的DNA条形码分子鉴定数据库。理想的条形码序列应当具有明显的种间变异,同时种内变异足够小,引物通用性好且易于扩增,能够通过测序得到高质量的序列[22-23],从而准确鉴别同属不同种的物种。随着十几年的发展,DNA条形码技术日趋成熟,已成功应用于药用植物栽培品种、中成药、保健食品、食用菌等领域的鉴定[24]。
本研究采用当前植物学界推荐的4个热点DNA条形码候选序列,从种内种间变异、barcoding gap、系统发育树3个方面分析不同DNA条形码对于芦荟属6种物种的鉴别能力,目的在于找到能快速、通用地鉴别芦荟属的DNA条形码序列。研究结果表明psbA-trnH序列能准确鉴别芦荟属的6个品种,可以作为鉴别芦荟属的优选DNA条形码序列。本研究选用的rbcL和matK序列采用barcoding gap法和构建系统发育树法均不能成功鉴定中华芦荟(79)和库拉索芦荟(83)这两个物种,原因是二者的亲缘关系较近,刘思言[25]提及中华芦荟Aloe vera L. var. chinensis Haw是库拉索芦荟Aloe barbadensis Miller的变种,这也就从分子水平上验证了中华芦荟和库拉索芦荟的近缘关系。ITS2序列不能将皂质芦荟(80)和海岸芦荟(81)区分开,同时ITS2序列未成功鉴别的样本没有与rbcL或者matK未鉴别出的样本重合,推测将ITS2序列与rbcL或者与matK序列组合也可以成功将芦荟属的这些物种完全区分开,但是由于psbA-trnH序列这一单序列就可以达到鉴别的目的,本研究没有进一步考察组合片段的鉴定效果。就ITS2序列和rbcL序列而言,每一个品种的3个不同植株的样品没有碱基差异;就psbA-trnH序列而言,皂质芦荟(80)的3个不同植株的样品在第559位有一个碱基差异,分别是T(80-1)、T(80-2)和碱基缺失(80-3);就matK序列而言,中华芦荟(79)的3个不同植株的样品在第739位有1个碱基差异,分别是G(79-1)、C(79-2)、C(79-3)。
由于采样条件的限制,本研究只对同一个产地的样品进行分析,并且GenBank中可用的芦荟属相关数据非常少,故没有纳入网上的数据做扩大样本量的分析。在后期的研究当中,需要适当扩大样品采集的范围,增加样品的数量。本研究虽然有一定的局限性,但仍具有鉴定意义,为芦荟属物种的准确鉴定提供科学依据,同时为其他药用植物的鉴定提供新的思路和方法。
| [1] | 中国药典[S]. 一部. 2010. |
| [2] | 马艳萍, 徐呈祥, 刘友良. 芦荟叶的特异结构、叶内生物活性成分及其与逆境适应的关系[J]. 热带作物学报, 2010,31 (4) :676. |
| [3] | Hebert P D, Gregory T R. The promise of DNA barcoding for taxonomy[J]. Syst Biol, 2005, 54 (5) :852–859 . |
| [4] | Hollingsworth P M, Forrest L L, Spouge J L, et al. A DNA barcode for land plants[J]. Proc Natl Acad Sci USA, 2009, 106 (31) :12794–12797 . |
| [5] | Chen S L, Yao H, Han J P, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS One, 2010, 5 (1) :e8613. |
| [6] | 杨培, 周红, 辛天怡, 等. 黄精属药用植物DNA条形码鉴定研究[J]. 世界中医药, 2015,10 (8) :1173–1176. |
| [7] | 张忠廉, 宋美芳, 李海涛, 等. 千斤拔属药用植物DNA 条形码鉴定研究[J]. 中草药, 2015,46 (1) :118–122. |
| [8] | 李亚辉, 马艳弘, 黄开红, 等. 芦荟多糖的超声波辅助纤维素酶提取及抗肿瘤活性研究[J]. 中国食品学报, 2015,15 (11) :91–97. |
| [9] | 何思微, 何凤林, 赵立超, 等. 库拉索芦荟中芦荟多糖提取方法的比较[J]. 食品研究与开发, 2012,33 (2) :32–35. |
| [10] | 朱蓉. 芦荟联合多种维生素治疗复发性口疮的短期疗效观察[J]. 医学理论与实践, 2015,28 (4) :488–489. |
| [11] | 崔燕. 芦荟对黄曲霉毒素B1致大鼠肝损伤的干预及其作用机制研究[D]. 无锡: 江南大学, 2014. |
| [12] | 杜国丰, 朱宝伟, 吴美毅, 等. 芦荟苹果保健醋饮料的研制[J]. 安徽农业科学, 2015,43 (31) :196–199. |
| [13] | 万金志, 徐新军, 钟佳胜, 等. 国外芦荟药品研究开发现状与趋势[J]. 今日药学, 2013,23 (1) :59–62. |
| [14] | 陈士林. 中药DNA条形码分子鉴定[M]. 北京: 人民卫生出版社, 2012 . |
| [15] | Ma X Y, Xie C X, Liu C, et al. Species identification of medicinal pteridophytes by a DNA barcode marker, the chloroplast psbA-trnH intergenic region[J]. Biol Pharm Bull, 2010, 33 (11) :1919–1924 . |
| [16] | Keller A, Schleicher T, Schultz J, et al. 5. 8S-28S rRNA interaction and HMM-based ITS2 annotation[J]. Gene, 2009, 430 (1/2) :50–57 . |
| [17] | Koetschan C, Hackl T, Muller T, et al. ITS2 database Ⅳ: interactive taxon sampling for internal transcribed spacer 2 based phylogenies[J]. Mol Phylogenet Evol, 2012, 63 (3) :585–588 . |
| [18] | 李妮亚, 高培元, 周鹏, 等. RAPD技术在芦荟属植物分类研究中的应用[J]. 西北植物学报, 2002,22 (6) :1432–1437. |
| [19] | Shioda H, Satoh K, Nagai F, et al. Identification of Aloe species by random amplified polymorphic DNA(RAPD) analysis[J]. Shokuhin Eiseigaku Zasshi, 2003, 44 (4) :203–207 . |
| [20] | Sahoo S, Rout G R. Plant regeneration from leaf explants of Aloe barbadensis Mill. and genetic fidelity assessment through DNA markers[J]. Physiol Mol Biol Plants, 2014, 20 (2) :235–240 . |
| [21] | 杨慧洁, 杨世海, 张淑丽, 等. 药用植物DNA条形码研究进展[J]. 中草药, 2014,45 (18) :2581–2582. |
| [22] | Song J Y, Yao H, Li Y, et al. Authentication of the family polygonaceae in Chinese Pharmacopoeia by DNA barcoding technique[J]. J Ethnopharmacol, 2009, 124 (3) :434–439 . |
| [23] | Yao H, Song J Y, Ma X Y, et al. Identification of Dendrobium species by a candidate DNA barcode sequence: the chloroplast psbA-trnH intergenic region[J]. Planta Med, 2009, 75 (6) :667–669 . |
| [24] | 辛天怡, 雷美艳, 宋经元. 中药材DNA条形码鉴定研究进展[J]. 中国现代中药, 2015,17 (2) :170–174. |
| [25] | 刘思言. 芦荟遗传转化受体体系的建立及转基因研究[D]. 长春: 吉林农业大学, 2007. |
 2016, Vol. 47
2016, Vol. 47


