2. 西藏藏医学院, 西藏 拉萨 850000
2. Tibetan Traditional Medical College, Lhasa 850000, China
由于多种原因,长期以来中药材一直存在不同程度的品种混乱、质量参差不齐的现象,严重影响中药的有效性、稳定性及用药的安全性。中药的“正本清源”一直是中药研究的基本任务之一。但仅仅基于传统方法进行中药基原物种的鉴定工作有其局限性。近年来,应用DNA条形码技术鉴定中药材研究进展迅速,并取得一定成果[1]。
2003年,加拿大圭尔夫大学的Paul Hebert正式提出生物学上的“barcode”概念,即从基因组标准化区间中得到的能用于鉴别、发现、区分物种以及给未确定的个体定种的一段基因短片段。DNA条形码的候选序列应具有以下特点:①适宜的长度,通常最好为600 bp左右;②较高的提取、扩增率以及测序的成功率;③在物种水平有足够的分歧度;④扩增方法和条件的通用性;⑤基因序列较稳定,即种间的差异远大于种内变异;⑥基因结构较为稳定。
植物的DNA条形码序列有3个主要来源:核基因组、叶绿体基因组和线粒体基因组,一般以前两者为主。通常认为线粒体基因较为保守,在中药DNA分子鉴定中的作用有限。事实上,线粒体基因组在不同类群(如科、属、种)间的进化速率是不相同的,对其系统学意义及其物种鉴定意义的评价应视具体情况而具体分析。目前越来越多的研究显示,线粒体基因组序列在中药基原植物鉴定中具有其特定优势;其与叶绿体基因组、核基因组相结合,可更为客观地反映物种、居群甚至个体间的遗传特性,为特定类群DNA条形码的构建打下坚实基础。本文就植物线粒体基因组特征及相关研究进展进行综述。
1 线粒体基因组基本特征 1.1 线粒体基因组的起源及其遗传特性线粒体是真核生物细胞中参与能量生产、代谢和体内平衡的半自主细胞器,在植物发育、适应和生殖方面扮演重要角色[2]。高等植物的线粒体与α-变形菌纲的紫细菌(purple bacteria)序列存在同源性,推测同叶绿体一样是来源于细菌的内共生起源[3-4]。
通常而言,被子植物线粒体同叶绿体一样为母系遗传,但也有部分父系以及双亲遗传的例子。例如,黄瓜属Cucumis Linn. 的线粒体体现了被子植物中少有的父系遗传特性[5]。裸子植物中多为母系遗传[6]。
1.2 线粒体基因组结构特征植物线粒体基因组通常类似于叶绿体基因组,为环状分子结构。而研究显示,植物线粒体基因组是由多种不同形态的DNA分子组成的混合体[7]。一般由双链主环分子(master circular chromosome)以及重复序列之间重组生成的多个亚环分子(subgenomic circular chromosome)组成[8]。Palmer和Shields建立的三环模型是其中最简单的情况,通过长约2 kb的重复序列介导的重组形成(图 1中用小箭头表示,内含cox Ⅱ基因)。整个线粒体基因组在大小为218 kb主环分子同135 kb(细线表示)和83 kb(粗线表示)的亚环分子之间转换(图 1)[9-10]。结构的复杂度与线粒体上重组活性重复区的数量、位置、方向有关[11]。虽然线粒体基因组二级结构较复杂多变,但其所含的序列相对固定,对于鉴定研究的影响不大。
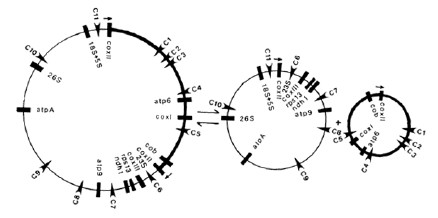
|
图 1 油菜线粒体基因组的三环结构 Fig.1 Tricircular structure of mitochondrial genome in Brassica campestris |
被子植物线粒体基因组的大小、结构以及基因顺序上变化较大[10, 12-13],即便是在较窄的分类学跨度上也存在显著差异[14]。植物线粒体基因组远大于动物线粒体基因组,介于200~2 500 kb,变异幅度达10倍以上[12],即使在同一科中也可能存在较大差异。目前已知的植物线粒体基因组长度最小的为地钱Marchantia polymorpha L.,约183 kb[15];最大的是甜瓜Cucumis melo L.,为2 500 kb[11]。大多数被子植物线粒体大小集中于300~600 kb。
1.3 线粒体基因组结构变化植物线粒体结构多变,主要体现在以下3方面:①即使亲缘关系较近物种的线粒体也很难找到分布模式的共性,且基因间位置独立、分散[4]。但线粒体种下结构比较保守[16]。②植物线粒体基因组的结构变化比较频繁,常发生高频率的同源重组[2],且间隔区还常发生加倍、替换[10]。③植物线粒体基因的丢失较为常见,尤其是核糖体蛋白以及琥珀酸脱氢酶基因(sdh基因)[17]。除rps2和rps12基因在被子植物早期少数丢失,绝大多数的核糖体蛋白基因分别独立地从线粒体基因上丢失[14]。
植物线粒体还同叶绿体、细胞核(较少)之间存在基因交换[14]。但目前还没有证据表明线粒体中的基因曾进入了叶绿体中[18]。线粒体基因组中发现过长度达12 kb的叶绿体片段[10]。地钱线粒体DNA中有11种编码不同核糖体蛋白的基因同时存在于叶绿体基因组中[19]。许多线粒体基因的细胞核转移被认为要早于叶绿体内共生事件[19]。
1.4 线粒体基因组碱基替换虽然线粒体结构具多变性,但植物线粒体基因序列进化非常缓慢,点突变速率比动物低40~100倍[11],碱基替换频率较低,尤其是在编码序列,即使是同义突变也非常少[20]。线粒体rRNA和tRNA在碱基序列以及二级结构上也非常保守[4]。因此,对研究一些较基部的分支可能具有一定优势。此外,某些属的线粒体有着较快速地进化。例如,车前属Plantago L. 植物的线粒体同义替换速率达到了其他植物的4 000倍,其中最快的一支甚至超过了动物线粒体[21],在该分类群中具有潜在的分子标记。
1.5 线粒体基因组编码基因线粒体基因主要编码4种呼吸链复合体的亚基:①细胞色素氧化酶C(cytochrome oxidase subunits),包括cox Ⅰ、cox Ⅱ、cox Ⅲ;②细胞色素b(cytochrome b);③NADH脱氢酶复合体(NADH dehydrogenase subunits),包括nad1~7,nad9;④ATP酶亚基(ATPase subunits),包括atp6、atp8、atp9、atpA[12, 22]。另外,线粒体DNA编码至少3种核糖体蛋白:①核糖体大亚基(ribosomal subunit genes of the large subunits)、小亚基(ribosomal subunit genes of the small subunits);②核糖体RNA(ribosomal RNA),包括rrn5、rrn18、rrn26;③一种或多种未知功能的开放阅读框(open reading frames)以及一部分高度分散的tRNA[10]。
综上,植物线粒体基因组有以下特点:①基因组庞大、冗余且物种间大小差异大;②与其他基因组存在基因交换;③基因组片段经历多次重复和丢失;④重复序列间的重组使其结构复杂;⑤序列结构变化迅速;⑥碱基上的变化缓慢。
2 线粒体基因在药用植物DNA条形码鉴定中的应用 2.1 线粒体基因的优势线粒体基因在中药鉴定方面具有以下优势:①在较高分类等级线粒体基因具有比较高的可比度;②一些线粒体基因的重排对于近缘物种间有一定鉴定意义;③在一些较高的分类等级中,线粒体基因及内含子的缺失可能是较好的分子标记;④线粒体通常为单亲遗传,直接测序很少出现重叠峰,便于读谱;⑤组织特异性不严格,在一些非叶组织中也能获得所需DNA模板,这一点是叶绿体分子标记所不及的。
2.2 线粒体基因的局限性线粒体基因在中药鉴定方面存在一些局限性,主要体现在:①进化速度较慢,鉴别位点有限;②在植物体中量偏低;③基因组间的转移和重排比较常见,在同源性分析上受到影响;④部分有鉴定价值的片段(如atpA、coxⅢ)在某些分类群中缺失。
2.3 线粒体片段在药用植物DNA分子鉴定中的应用2002年,Duminil为35个植物线粒体片段设计了PCR通用引物,开拓了植物线粒体片段大量运用的先河[23]。就目前而言,被子植物线粒体DNA条形码筛选较为少见,一般认为比较适宜的条形码候选序列主要有nad1、cox Ⅰ、nad2、atpA、cob和cox Ⅲ等[10]。
2.3.1 NADH脱氢酶片段该片段可用于石斛的分子鉴定,成功鉴别出9种石斛属Dendrobium Sw.植物中的8种[24]。耿丽霞等[25]应用nrDNA ITS+nad 1-intron 2新型联合片段构建了UPGMA树,与其他联合片段相比,更能有效地对中药枫斗类石斛基原植物进行鉴别,并可区分细茎石斛及霍山石斛。
根据研究报道,nad1第2内含子在稻族(tribe Oryzeae Dum.)中富含插入缺失且序列的变异呈均匀分布,无碱基偏好[26],可用于该类群的系统发育分析及鉴定。线粒体建树结果与先前形态解剖学以及matK片段[27]得出的结论一致:稻族是一快速分化类群,其下能分为两亚族:Oryzinae和Zizaniinae。对于位置存在争议的种Porteresia coarctata (Roxb.) Tateoka,nad1第2内含子同以往叶绿体DNA限制性片段分析[28]、叶绿体及核基因片段[29]所得出的结果相同。
通过此片段中的2个简单重复序列可区分挪威云杉Picea abies (L.) H. Karst. 的不同居群[30];与核基因同工酶[31]及叶绿体微卫星分析[30, 32]结果相比,其种群分化系数(GST)要明显高得多。通过进一步分析发现重复区域在植物线粒体中比较广泛,具有作为一种分子标记的潜力。
nad5的非编码区在不同种群的红松Pinus koraiensis Sieb. et Zucc. 中存在变异位点,可以利用该片段进行分子系统地理学分析[33]。其结果和线粒体cox Ⅲ[33]、叶绿体psbA-trnH[35]和trnL-FB[36]非编码区较为一致。4种分子证据均指出:红松共享单倍型主要集中于吉林、辽宁和吉林、黑龙江居群之间,推测吉林-辽宁以及吉林-黑龙江红松居群间存在基因流;野生红松在第4纪冰期的避难所位于吉林省长白山地区。线粒体同叶绿体单倍型分析结果的一致性证明了线粒体基因在分子系统学上运用的可行性。
在雪松属Cedrus Trew的分子系统发育及地理生物学研究中,母系遗传的nad1第2内含子、nad5第1内含子以及orf25的联合序列相对于父系遗传的叶绿体联合序列建立的系统树一些分支上具有更高的支持度[37]。
nad7第2内含子在红景天属Rhodiola L. 中特殊的序列长度对属内物种具有一定的鉴定意义[38]。并且支持先前核基因组ITS区的聚类分析结果:大花红景天与长鞭红景天的部分居群聚为一支,说明云南和四川境内一些地区的长鞭红景天有较高的居群分化[39]。
韩国的人参栽培种“Chunpoong”在nad7第4内含子存在一个单核苷酸多态性(SNP),能用于同其他栽培种的鉴别[40]。另2种栽培种“Gumpoong”和“Chungsun”采用多重扩增耐突变系统(multiplex amplification refractory mutation system,MARMS),利用nad7第3内含子以及结合“Gumpoong”在26 S rDNA中1个SNP位点能同其他栽培种区分 开[41]。
2.3.2 细胞色素氧化酶C片段cox Ⅱ序列由于在香蕉品种Musa×paradisiaca L. 中含有高变区,能对其16个变种进行鉴定研究[42];该片段将这些香蕉品种按基因型主要分为三大类。cox Ⅱ第一内含子在胡桃Juglans regia L. 中具有多个变异位点和插入缺失,与核基因rDNA片段相比在该种基因型的分子鉴别上有更优秀的表现,借助ARMS(amplification refractory mutation system)-PCR能够区分其种下的诸多基因型[43]。
2.3.3 其他线粒体基因片段南瓜属Cucurbita Linn. 线粒体atp4-ccmc间隔区所提供的信息同叶绿体片段相比存在一些差异。Maxima组的分支相比叶绿体基因树要更为接近多年生物种,这可能与基因片段的特异性有关。不过其内部分支与叶绿体基因树相一致[44]。
ccb256片段能用于芭蕉属Musa L. 种间以及亚种间的比较[45]。该片段主要结果与传统形态分类以及AFLP[46]、PCR-RFLP[47]等分子证据基本一致。由于芭蕉属植物线粒体独特的父系遗传特点,在推断芭蕉属父系系谱关系的研究中具有一定优势。研究者从该片段的结果中推测M. acuminata subsp. Truncata (Ridl.) Kiew可能为M. schizocarpa N. W. Simmonds的父本来源;Ice cream(ABB)可能是由野蕉M. balbisiana Colla为父本提供B基因杂交而来;小果野蕉M. acuminata Colla可能作为父本提供了A基因杂交出了除Improved lady finger(AAB)以外的栽培蕉品种。
植物线粒体DNA在药用植物分子鉴定、系统发育分析以及种群遗传多样性研究中占有不可忽视的地位。其主要原因有以下几点:①线粒体在某些种属中的超变异性;②在一部分分类群中为父系遗传,能很好地寻找父本信息;③线粒体基因扩散(gene dispersal)的减少以及高基因漂变(genetic drift)能够更为清晰地“跟踪”遗传背景复杂的栽培种或者入侵居群的起源[48];④线粒体基因序列含有的SNP方便结合ARMS-PCR等技术,更利于物种鉴定。
3 结语与展望虽然线粒体序列的进化速率慢于核基因与叶绿体基因,一般运用于较高的分类等级,如属间、科间甚至更高[49];但线粒体基因组中有着丰富的长度多态性以及自身的优势,在中药分子鉴定研究上具有一定价值,随着研究的不断深入有望进一步得到开发。
在应用DNA条形码鉴别植物时,单一的片段尚无法完美解决鉴定问题[50]。且运用单一片段进行物种鉴别存在一定的缺陷,如缺少辨识度、通用引物的扩增及测序效率不定、种下变异的存在导致鉴别位点稳定性不足、亲缘关系近的植物间易发生杂交及基因流渗入。目前尚无法找到植物某一“通用基因”作为条形码。因此,多基因组多片段的组合为必然趋势。线粒体基因组可提供多片段组合条形码的候选序列,可作为核基因以及叶绿体基因标记一个很好的补充。例如,有研究发现rbcL上的SNP结合2个线粒体特异性片段,可以快速区分落叶松属Larix Mill. 中的一些种[51]。
线粒体序列的独特优势还在于组织特异性不严格,在一些非叶组织中也能获得所需DNA模板,这一点是叶绿体分子标记所不及的。而相当一部分中药材来自于植物的地下部分,应用线粒体序列条码进行药材鉴定显然比叶绿体分子标记更为可行。
植物线粒体基因在分子鉴定中的应用受到自身进化速度的限制,但对于DNA条形码而言,标记序列的分辨率比序列变异度重要,这使得线粒体碱基替换速率慢的劣势就不那么凸显了。同时,线粒体在碱基序列上十分的稳定,在一些物种中发现的SNP有能力作为稳定的鉴别位点进行标记,并借助ARMS-PCR或CAPS(cleaved amplified polymorphic sequences)markers省去测序步骤,这种方法已经在研究中证实可行[52]。此外,植物线粒体物种间多变的基因组大小、结构上的差异及基因缺失的规律或许是中药分子鉴定研究的新思路,这些结构特性可能作为一种不同于传统基于序列变化的新型分子标记,值得进一步研究。因此,植物线粒体在中药分子鉴定上存在很大的潜力,持续探究将拥有更大的应用空间。
| [1] | 杨慧洁, 杨世海, 张淑丽, 等. 药用植物DNA条形码研究进展[J]. 中草药, 2014,45 (18) :2581–2587. |
| [2] | Gualberto J M, Mileshina D, Wallet C, et al. The plant mitochondrial genome:Dynamics and maintenance[J]. Biochimie, 2014, 100 :107–120 . |
| [3] | Burger G, Gray M W, Franz L B. Mitochondrial genomes:anything goes[J]. Trends Gene, 2003, 19 (12) :709–716 . |
| [4] | Gray M W. Origin and evolution of mitochondrial DNA[J]. Annu Rev Cell Biol, 1989, 5 :25–50 . |
| [5] | Havey M J. Predominant paternal transmission of the mitochondrial genome in cucumber[J]. J Hered, 1997, 88 (3) :232–235 . |
| [6] | Gottlieb L D, Jain S K. Plant Evolutionary Biology[M]. London: Chapman and Hall Ltd, 1988 . |
| [7] | Kubo T, Newton K J. Angiosperm mitochondrial genomes and mutations[J]. Mitochondrion, 2008, 8 (1) :5–14 . |
| [8] | Palmer J D, Herbon L A. Unicircular structure of the Brassica hirta mitochondrial genome[J]. Curr Genet, 1987, 11 (6/7) :565–570 . |
| [9] | Palmer J D, Shields C R. Tripartite structure of the Brassica campestris mitochondrial genome[J]. Nature, 1984, 307 (2) :437–440 . |
| [10] | Palmer J D. Mitochondrial DNA in Plant Systematics:Applications and Limitations[M]. New York: Springer US, 1992 . |
| [11] | Janska H, Woloszynska M. The dynamic nature of plant mitochondrial genome organization[J]. Acta Biochim Pol, 1997, 44 (2) :239–250 . |
| [12] | Levings C S, Brown G G. Molecular biology of plant mitochondria[J]. Cell, 1989, 56 (2) :171–179 . |
| [13] | Adams K L, Qiu Y L, Stoutemyer M, et al. Punctuated evolution of mitochondrial gene content:high and variable rates of mitochondrial gene loss and transfer to the nucleus during angiosperm evolution[J]. Proc Natl Acad Sci USA, 2002, 99 (15) :9905–9912 . |
| [14] | Knoop V. The mitochondrial DNA of land plants:peculiarities in phylogenetic perspective[J]. Curr Genet, 2004, 46 (3) :123–139 . |
| [15] | Oda K, Kohchi T, Ohyama K. Mitochondrial-DNA of Marchantia Polymorpha as a single circular form with no Incorporation of foreign DNA[J]. Biosci Biotechnol Biochem, 1992, 56 (1) :132–135 . |
| [16] | Palmer J D. Intraspecific variation and multicircularity in Brassica mitochondrial DNAs[J]. Genetics, 1988, 118 (2) :341–351 . |
| [17] | Adams K. Evolution of mitochondrial gene content:gene loss and transfer to the nucleus[J]. Mol Phylogenet Evol, 2003, 29 (3) :380–395 . |
| [18] | Hao W, Palmer J D. Fine-scale mergers of chloroplast and mitochondrial genes create functional, transcompartmentally chimeric mitochondrial genes[J]. Proc Natl Acad Sci USA, 2009, 106 (39) :16728–16733 . |
| [19] | Takemura M, Oda K, Yamato K, et al. Gene clusters for ribosomal-proteins in the mitochondrial genome of a liverwort, marchantia polymorpha[J]. Nucl Acids Res, 1992, 20 (12) :3199–3205 . |
| [20] | Wolfe K H, Li W H, Sharp P M. Rates of nucleotide substitution vary greatly among plant mitochondrial, chloroplast, and nuclear DNAs[J]. Proc Natl Acad Sci USA, 1987, 84 (24) :9054–9058 . |
| [21] | Cho Y, Mower J P, Qiu Y L, et al. Mitochondrial substitution rates are extraordinarily elevated and variable in a genus of flowering plants[J]. Proc Natl Acad Sci USA, 2004, 101 (51) :17741–17746 . |
| [22] | 胡德昌. 柿及其部分近缘种mtDNA和cpDNA多态性分析[D]. 武汉:华中农业大学, 2007. |
| [23] | Duminil J, Pemonge M H, Petit R J. A set of 35 consensus primer pairs amplifying genes and introns of plant mitochondrial DNA[J]. Mol Ecol Notes, 2002, 2 (4) :428–430 . |
| [24] | 张婷, 王峥涛, 徐珞珊, 等. 线粒体nad1内含子2序列在石斛属植物分子鉴定中的应用[J]. 中草药, 2005,36 (7) :1059–1062. |
| [25] | 耿丽霞, 郑瑞, 任洁, 等. 新型联合片段:nrDNAITS+ nad 1-intron 2在枫斗类石斛鉴定中的意义[J]. 药学学报, 2015,50 (8) :1060–1067. |
| [26] | 郭亚龙, 葛颂. 线粒体nad1基因内含子在稻族系统学研究中的价值——兼论Porteresia的系统位置[J]. 植物分类学报, 2004,42 (2) :333–344. |
| [27] | Ge S, Li A, Lu B R, et al. A phylogeny of the rice tribe Oryzeae (Poaceae) based on matK sequence data[J]. Am J Bot, 2002, 89 (12) :1967–1972 . |
| [28] | Duvall M R, Peterson P M, Terrell E E, et al. Phylogeny of north-american oryzoid grasses as construed from maps of plastid DNA restriction sites[J]. Am J Bot, 1993, 80 (1) :83–88 . |
| [29] | Ge S, Sang T, Lu B R, et al. Phylogeny of rice genomes with emphasis on origins of allotetraploid species[J]. Proc Natl Acad Sci USA, 1999, 96 (25) :14400–14405 . |
| [30] | Sperisen C, Buchler U, Gugerli F, et al. Tandem repeats in plant mitochondrial genomes:application to the analysis of population differentiation in the conifer Norway spruce[J]. Mol Ecol, 2001, 10 (1) :257–263 . |
| [31] | Lagercrantz U, Ryman N. Genetic structure of Norway spruce (Picea abies):concordance of morphological and allozymic variation[J]. Evolution, 1990, 44 (1) :38–53 . |
| [32] | Vendramin G G, Anzidei M, Madaghiele A, et al. Chloroplast microsatellite analysis reveals the presence of population subdivision in Norway spruce (Picea abies K.)[J]. Genome, 2000, 43 (1) :68–78 . |
| [33] | 田菲菲. 基于线粒体DNA nad5序列研究天然红松的分子系统地理学[D]. 大连:辽宁师范大学, 2011. |
| [34] | 高嵩. 红松线粒体DNA coxⅢ遗传多样性及系统地理学研究[D]. 大连:辽宁师范大学, 2009. |
| [35] | 靖晶. 基于叶绿体DNA非编码序列的天然红松分子系统地理学研究[D]. 大连:辽宁师范大学, 2009. |
| [36] | 陈家媛. 基于叶绿体DNA trnL-FB序列研究天然红松的分子系统地理学[D]. 大连:辽宁师范大学, 2010. |
| [37] | 乔才元. 雪松属的分子系统发育与生物地理学[D]. 北京:中国科学院植物研究所, 2006. |
| [38] | 邓科君, 杨足君, 刘成, 等. 红景天线粒体nad7基因内含子2序列测定及其系统发育意义[J]. 遗传, 2007,29 (3) :371–375. |
| [39] | 倪念春, 王卫华. 云南常见药用红景天的分子鉴定研究[J]. 山西中医学院学报, 2004,5 (3) :48–52. |
| [40] | Wang H, Sun H, Kwon W S, et al. Molecular identification of the Korean ginseng cultivar "Chunpoong" using the mitochondrial nad7 intron 4 region[J]. Mitochondrial DNA, 2009, 20 (2/3) :41–45 . |
| [41] | Wang H, Kwon W S, Yang D U, et al. Development of a multiplex amplification refractory mutation system for simultaneous authentication of Korean ginseng cultivars "Gumpoong" and "Chungsun"[J]. Mitochondrial DNA, 2011, 22 (1/2) :35–38 . |
| [42] | 闻盼盼, 郭泽锋, 许林兵, 等. 线粒体基因高变区应用于香蕉种属亲缘关系的鉴定研究[J]. 安徽农业科学, 2010,11 (8) :91–93. |
| [43] | Ciarmiello L F, Pontecorvo G, Piccirillo P, et al. Use of nuclear and mitochondrial single nucleotide polymorphisms to characterize English walnut (Juglans regia L.) genotypes[J]. Plant MolBiol Rep, 2013, 31 (5) :1116–1130 . |
| [44] | 郑怡鸿. 南瓜属植物的分子系统发育及分子钟研究[D]. 武汉:武汉大学, 2011. |
| [45] | 邓长娟, 王静毅, 陈友, 等. 芭蕉属植物基于线粒体ccb256序列的分类研究[J]. 热带作物学报, 2011,32 (9) :1687–1691. |
| [46] | Wong C, Argent G, Kiew R, et al. The genetic relations of Musa species from Mount Jaya, new guinea, and a reappraisal of the sections of Musa (Musaceae)[J]. Gardens Bull Singapore, 2003, 55 (1) :97–111 . |
| [47] | Nwakanma D C, Pillay M, Okoli B E, et al. Sectional relationships in the genus Musa L. inferred from the PCR-RFLP of organelle DNA sequences[J]. Theor Appl Genet, 2003, 107 (5) :850–856 . |
| [48] | Schaal B A, Olsen K M. Gene genealogies and population variation in plants[J]. Proc Natl Acad Sci USA, 2000, 97 (13) :7024–7029 . |
| [49] | Qiu Y L, Li L, Wang B, et al. Angiosperm phylogeny inferred from sequences of four mitochondrial genes[J]. J Syst Evol, 2010, 48 (6) :391–425 . |
| [50] | Fazekas A J, Kesanakurti P R, Burgess K S, et al. Are plant species inherently harder to discriminate than animal species using DNA barcoding markers?[J]. Mol Ecol Resour, 2009, 9 (1) :130–139 . |
| [51] | 管玉霞. 落叶松部分cpDNA mtDNA及ITS序列研究及其应用于落叶松种的鉴定的可行性[D]. 北京:北京林业大学, 2006. |
| [52] | Lee J W, Bang K H, Kim Y C, et al. CAPS markers using mitochondrial consensus primers for molecular identification of Panax species and Korean ginseng cultivars (Panax ginseng C. A. Meyer)[J]. Mol Biol Rep, 2012, 39 (1) :729–736 . |
 2016, Vol. 47
2016, Vol. 47


