当归属Angelica L.是伞形科(Umbelliferae)二年生或多年生草本植物,全世界约有80个物种,大部分产于北温带和新西兰地区。我国有26种5变种1变型,主要分布于我国的中部、东北部和西南部地区[1]。当归属中许多物种具有重要的药用和经济价值,其中《中国药典》2010年版一部[2]收录的当归属药材有当归Angelica sinensis (Oliv.)Diels、白芷A.dahurica (Fischer ex Hoffmann)Bentham &J.D.Hooker ex Franchet &Savatier和独活A.pubescens Maxim.F.biserrataShan.et.Yuan,它们的干燥根具有解表散寒、祛风止痛、宣通鼻窍等功效。此外,除了药典收录的上述3个物种,该属的其他物种也具有一定的药用价值,在我国的部分地区和东南亚国家的一些区域,该属的一些物种也被作为重要的中药材使用。例如在我国西部的青海和四川的部分地区,当归属的青海当归A.nitida H.Wolff和大叶当归A.megaphglla Diels被作为当归的替代品使用;在我国东北的吉林延边和日本、朝鲜、韩国等国家,东当归A.acutiloba (Siebold &Zuccarini)Kitagawa被大量栽培,并类似于当归药用[3,4]。
然而,当归属植物种类繁多,外部形态特征接近,而且物种间存在着大量的中间过渡性状,致使该属物种的特征性状难以鉴别,导致该属物种鉴定困难。而前人从植物来源、外部形状特征、显微特征以及理化性质等方面对当归属药用植物进行了鉴别研究。如潘泽惠等[5]研究了当归属及其近缘属12种植物的核型关系,结合其他性质特征,认为核型关系可以作为该属物种初步鉴别的依据。张巧英等[6]调查并整理了中国四川省当归属的植物资源,为当归属药用植物的道地性研究提出了建议。冯图等[7]对中国西南部地区的12种当归属植物的叶表皮进行了显微鉴别,结果表明显微特征也可以作为该属物种的部分鉴别依据。廖晨阳等[8]基于形态解剖学特征对当归属植物进行了分类研究,发现叶柄解剖特征可以作为该属物种进化和系统分类的依据。另外,姚惠萍[9]对当归属药用植物的化学成分进行了系统性的研究综述,为该属物种的分类鉴定提供了依据。然而,这些传统的形态鉴别方法主要依赖于专业人员的分类知识和鉴别经验,而显微鉴别和理化鉴别等方法因受植物生长区域气候、环境和地理条件的限制,同时受植物生长发育期和储存条件等诸多综合因素的影响,使得这些方法不能完全准确和快速地鉴定当归属植物。
随着分子生物学技术的快速发展,DNA条形码的概念被提出并很快应用到生物物种的分子鉴定方面[10]。这一技术的发现为药用植物物种的快速鉴定带来新的曙光,该技术使用一段标准的通用DNA条形码序列,从基因水平上进行物种鉴别,可以避免药用植物自身的复杂性状和其他外界因素对鉴定结果造成的影响。近几年来,DNA条形码已成为全球植物分类学新的研究热点方向,在Nature、Science、PANS、PLoS ONE、《中草药》等国际国内著名学术刊物上发表了大量的研究报道[11,12,13,14,15,16,17]。而在2009年,国际生命条形码联盟(CBOL)选用7个叶绿体DNA引物进行实验,从中确定rbcl和matK作为植物物种鉴定的标准条形码[18]。2011年,中国植物条形码工作组(China Plant BOLGroup)选用中国种子植物75科1 757种,共6 286个样品为研究对象,分别对叶绿体基因片段rbcl、matK和trnH-psbA以及核基因ITS引物的物种分辨率和序列扩增质量进行比较研究,发现ITS序列的物种分辨率显著高于其他3个基因片段,建议将ITS基因纳入种子植物的核心条形码[19,20]。
随着DNA条形码技术的进一步发展,这一方法也逐渐应用到当归属药用植物物种的系统进化和分子鉴定研究方面。例如薛华杰等[21]选择少量当归属及其近缘属植物,测定其ITS序列,对当归属药用植物的种间系统发育关系进行了分析,为当归属的分类提供了一定依据。辛天怡等[22]基于ITS2条形码序列对当归药材及其混伪品进行鉴定,构建的NJ树能够将当归及其混伪品区分开,结果表明ITS2条形码可作为当归物种有效的鉴别方法。张春等[23]通过对当归及其混伪品中药材ITS序列的分析,表明ITS序列可作当归药材鉴别的有效分子标记。然而,以往这些研究主要集中在当归属单一物种当归及其混伪品或者近缘植物物种的中药分子鉴定方面,或者只是取样当归属一个物种一个个体的DNA分子序列进行系统发育和种间进化关系研究。本研究将着眼于当归属整个药用植物物种水平,选取多个当归属物种多个ITS的DNA分子序列,基于群体遗传学的方法进行当归属药用植物物种的DNA分子鉴定研究,为该属植物物种的准确和快速鉴定提供理论依据。
1 材料本研究首先在野外采取当归属中青海当归的实验材料,所有材料由西北大学生命科学学院房敏峰教授鉴定为青海当归Angelica nitida H.Wolff,凭证标本存放于西北大学植物标本馆。野外采样基本覆盖了该物种整个的自然地理分布区,材料分别来自青海、四川和甘肃3个省区的5个采样地点,每个采样地采集5株新鲜健康幼嫩叶片,立即置于盛有硅胶的密封袋中干燥处理。采样时注意个体和个体之间至少间隔50 m,群体之间间隔50 km以上。同时,注意观察每个采样群体的具体生境、土壤和伴生物种等信息,详细记录每个采样地的海拔和经纬度等信息(表 1)。
| 表 1 青海当归的采样信息 Table 1 Sample information of A. nitida |
参考CTAB法[24],对提取方法稍作修改。选取青海当归5个群体共25份个体提取总DNA。用1%的琼脂糖凝胶电泳和紫外分光光度法检测所得DNA的浓度和纯度。选用CBOL推荐的ITS序列[19]进行扩增,正向引物ITS5为5’-GGAAGGAGAAGT-CGTAACAAGG-3’,反向引物ITS4为5’-TCCTCC-GCTTATTGATATGC-3’。引物由上海生工生物工程有限公司合成。PCR反应体系为25 μL,包含DNA模板20~100 ng,PCR MIX 12.5 μL(中国润德公司),2.5 μmol/L正、反向引物各0.75 μL、ddH2O8.5 μL。反应程序如下:首先94 ℃预变性5min;接着30个循环:包括94 ℃变性40s,60 ℃退火40s,72 ℃延伸90s;然后72 ℃终延伸10 min,4 ℃冰箱保存备用。PCR扩增产物利用1%的琼脂糖凝胶电泳检测后,送往上海生工生物工程公司,利用遗传分析仪ABI 3730 xl进行测序。
2.2 数据分析测序共得到了青海当归5个自然群体25份个体的ITS序列,由于同一群体5份个体的ITS基因型序列完全相同。因此数据分析时,每个群体只选择一份个体的序列作为代表。同时通过与GenBank数据库中的当归属其他物种的ITS序列进行对比分析,最终选择158条当归属药用植物物种的ITS序列,共32个物种,一并进行分析(表 2)。首先对测序获得的原始序列峰图利用软件Chromas 2.22进行人工校对。将校对后的实验数据利用软件Bioedit 7.0.9.0[25]进行多序列添加,使用MEGA6.0软件[26]去除所有序列前后两端的低质量区,只保留共同区段序列,并基于Kimura-2-parameter(K2P)模型计算种内和种间遗传距离,分别用邻接(neighbor-joiningtree,NJ)法,最大似然树(maximum-likelihood tree,ML)法和最大简约树(maximum-parsimony tree,MP)法构建系统发育树,并以Bootstrap重复1 000次,分析系统发育树各分支的支持率。
| 表 2 GenBank的ITS数据 Table 2 ITS sequences data obtained from GenBank |
序列经比对处理后,158条当归属的ITS序列总长度为530 bp,利用MEGA 6.0软件,基于K2P模型计算各物种的种间和种内遗传距离(图 1)。统计结果显示,当归属物种间遗传距离的分布范围为0.29%~12.00%,其中种间最小遗传距离为0.29%,种内遗传距离的分布范围为0~0.2%,其中种内最大遗传距离为0.2%。当归属中各物种种间距离分布大,种内距离分布小,且种间最小距离大于种内最大距离,说明ITS序列可以用于当归属药用植物的物种鉴定。
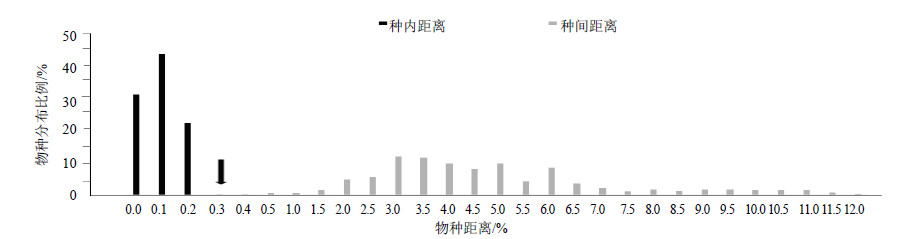 | 图 1 当归属种间和种内的遗传距离Fig.1 Intraspecific and interspecific distances of species in Angelica L. |
利用MEGA6.0软件,分别构建NJ、ML和MP,结果显示3种系统发育树的拓扑结构高度一致(图 2),每个物种的ITS基因型序列能够较好的聚为对应于该物种本身的分支,且具有中到高度的支持率,说明ITS序列能将当归属的32个物种基本全部区分。例如《中国药典》收录的当归与民间药用的其他当归属植物完全分开,且支持率为100%,同时该物种和极细当归具有较近的亲缘关系,聚为一个较大的分支(支持率为99%),位于系统发育树的最基部,显示了他们的古老性质。另外,民间广泛使用的药用植物当归的替代品大叶当归和东当归各自的ITS基因型序列也能聚为对应于该物种本身的独立遗传分支。然而,本研究同时发现,青海当归的序列没有形成独立的单系分支,实验中测定的1条序列与其他来自GenBank数据库中的青海当归的序列聚成一支;同时,其余4条序列和阿坝当归的ITS序列聚为一支,具有中度的支持率(59%),说明这个物种和阿坝当归可能具有较近的亲缘关系。
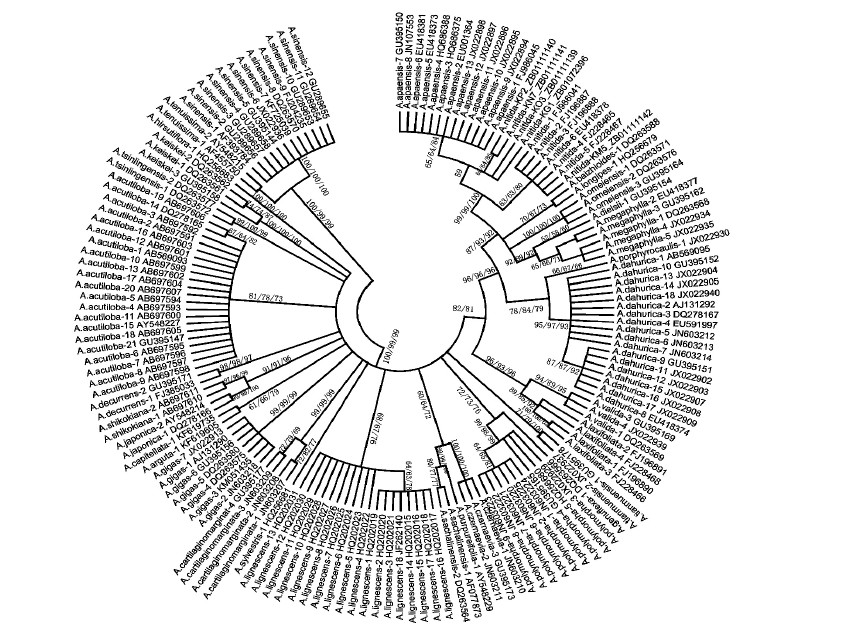 | 图 2 基于当归属32个物种的ITS序列构建的系统发育树Fig.2 Phylogenetic tree based on ITS sequences of 32 species in AngelicaL. |
ITS序列经比对排列整齐后,存在很多变异位点,这些变异位点可以作为DNA分子特征条形码,为当归属的鉴定提供依据。例如阿坝当归、小叶独活以及日本当归在系统发育树中很明显地聚在不同的分支中,在其ITS序列中寻找各自特有的条形码。
如图 3,阿坝当归分别在43 bp(碱基A)、56 bp(碱基T)、62~64 bp(碱基CCG)以及70 bp(碱基A)都存在与其他2个物种区分的特异性碱基;小叶独活在53 bp(碱基T)和60 bp(碱基T)处存在2个特异性碱基,而日本当归则在37~39 bp(碱基ATC)处与其他2个物种不同。另外,青海当归和峨眉当归作为当归的替代品和易混品,也能找到物种各自特有的条形码位点。如图 4,分别在109~111 bp、152~154 bp 2处的3个碱基存在显著的差异,这2个碱基位点可以将这3个物种完全区分。除此之外,在120 bp(碱基C)、130 bp(碱基A)和143 bp(碱基A)处为当归的特征碱基。此外,拐芹在东北被当作白芷药用,而大叶当归经常用作当归。如图 5,在336~338 bp处的3个碱基可以鉴别出白芷、大叶当归和拐芹3个物种;并且,分别在367 bp(碱基A)和371bp(碱基C)处有大叶当归和拐芹的特有碱基变异位点。
 | 图 3 阿坝当归、小叶独活和日本当归的ITS序列比对结果Fig.3 Sequence alignment of A. apaensis, A. czernaevia, and A. japonica in ITS region |
 | 图 4 青海当归、峨眉当归和当归的ITS序列比对结果Fig.4 Sequence alignment of A. nitida, A. omeiensis, and A. sinensis in ITS region |
 | 图 5 白芷、大叶当归和拐芹的ITS序列比对结果Fig.5 Sequence alignment of A. dahurica, A. megaphylla, and A. polymorpha in ITS region |
ITS序列由rDNA上的5.8 S、16 S、28 S rRNA基因序列和3个区域之间的内转录间隔区(internaltranscribed spacer,ITS)组成。一般所用的ITS条形码标记则是核糖体RNA上的非编码区序列,这段区域所承受的选择压力较小,呈中性进化模式,在物种内序列相对保守,而种间差异比较明显,进化速率较快,因此适合于近缘物种间和种内水平上的分析鉴定[27]。近年来,利用ITS序列作为药用植物分子鉴定的研究已广泛报道,是研究物种间系统进化和近缘种间关系的重要手段。例如张宏意等[28]选择不同产地的何首乌药材,分析和比对其ITS序列,结果证明ITS序列能够有效鉴别何首乌的道地药材和其野生种质资源。陶刚等[29]选择贵州地区类似钩藤的4种药用植物进行ITS序列分析,结果表明所选植物与药用钩藤属有很近的亲缘关系,为药用钩藤寻找到了有效的替代品。而张晓南等[30]通过比较黄草乌及其混淆品的ITS序列,发现了大量的物种特异性变异位点,可以作为药材混伪品鉴别的重要依据。彭禄等[31]选择同样是当归属的独活为研究对象,涉及2科5属17个种共26个居群的独活样品材料,进行ITS序列分析,综合构建系统发育树和单倍型式样进行分析,得出ITS序列可以鉴定独活物种。本研究中,基于当归属32种植物的ITS条形码序列,利用NJ、ML和MP法分别构建该属物种的系统发育树,结果显示,不同建树方法得到了高度一致的拓扑结构,而且每种植物的ITS基因型序列基本能够聚为一个对应于该物种本身的独立遗传分支,说明ITS序列可以很好的鉴别当归属植物物种。同时,研究发现了当归属物种在ITS区域存在大量的物种特异性变异位点,也可以作为该属物种鉴别的特异性位点。
4.2 ITS条形码鉴定当归属药用植物的可靠性通过对当归属32个物种共158条的ITS序列进行分析,结果说明基于群体遗传学的方法,利用ITS序列来鉴定当归属药用植物是可行的。构建的系统发育树将所选的32个当归属物种成功区分,并存在中到高度水平的支持率。其中当归和极细当归聚在一起,和其他物种能够明显区分,通过对比这2个物种的ITS序列,发现当归在多处有特异性碱基位点变异,如在82~84 bp的碱基为TAC,109~111 bp为GTA,399~401 bp为TGA,468~470 bp为TCT等,这些位点均可与极细当归进行区分。而在阿坝当归和青海当归形成的分支中,来自于GenBank数据库的青海当归序列和通过实验得到的序列(KM5_ZB01111142)形成一个分支,而另外4条序列形成了一个小分支,出现这个结果的原因可能是青海当归种内序列出现了遗传变异或分化。对比青海当归的ITS序列,发现青海当归存在3个种内变异位点,分别在31、334、444 bp,而实验序列(KM5_ZB01111142)与数据库下载的青海当归ITS序列完全一致。进一步分析系统发育树表明,青海当归和阿坝当归聚为一支,说明两物种间具有较近的亲缘关系,推测两物种间可能存在着基因渐渗(introgression)或杂交现象。野外调外也显示,这两个物种的自然地理分布区域存在着明显重叠现象,而且物种间具有大量的中间过渡形态,暗示这2个物种之间可能存在近期的基因流或渐渗。
一般来说,作为DNA条形码标记,要求种间遗传距离大于种内距离,只有种间差异尽可能的大,而种内差异尽可能的小,才能保证可以尽量不受到种内变异的影响,进一步更准确的鉴别物种之间的关系。本研究通过对当归属种内和种间遗传距离的计算,发现当归属的种间距离明显大于种内距离。虽然有些物种出现了种内变异,但是通过对比ITS序列,发现种间的变异位点可以很好地将所选的当归属物种区分开,例如阿坝当归和青海当归有2处变异位点,即31 bp(G-T)和187bp(T-C)可以将这2个种鉴定开来;长序当归、峨眉当归、城口当归和大叶当归共有14处变异位点,可以将这聚在一支的4个种区分开来;东当归、秦岭当归、明日叶和滨当归共有21个变异位点,可以将这4个种区分开来。
通过对当归属ITS序列的分析研究以及系统发育分析结果,说明以ITS序列作为DNA分子条形码,可以准确地对当归属药用植物进行区分和鉴定,为进一步利用当归属药用植物资源提供了重要科学依据。
| [1] | 陈书坤, 李恒, 陈邦余. 中国植物志(第55卷, 第3分册)[M]. 北京:科学出版社, 1997. |
| [2] | 中国药典[S]. 一部. 2010. |
| [3] | 晋玲, 吴迪, 崔治家, 等. 当归药材资源种类及分布研究[J]. 中兽医医药杂志, 2013(1):74-77. |
| [4] | 马瑞君, 王钦, 陈学林, 等. 当归的研究进展[J]. 中草药, 2002, 33(3):280-282. |
| [5] | 潘泽惠, 庄体德, 姚欣梅, 等. 当归属及近缘小属的核型演化及地理分布研究[J]. 植物分类学报, 1994, 32(5):419-424. |
| [6] | 张桥英, 何兴金. 四川当归属植物资源及其开发利用[J]. 中国野生植物资源, 2003, 22(1):17-19. |
| [7] | 冯图, 刘爽, 何兴金. 西南地区当归属植物叶表皮微形态研究[J]. 四川大学学报:自然科学版, 2009, 46(6):1798-1804. |
| [8] | 廖晨阳, 何兴金, 马祥光. 中国广义当归属及其相关类群的叶柄结构研究[J]. 西北植物学报, 2012, 32(1):90-98. |
| [9] | 姚惠萍. 当归属药用植物化学成分研究概况[J]. 现代中药研究与实践, 2003, 17(2):56-58. |
| [10] | Hebert P D N, Ratnasingham S, de Waard J R. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc Royal Soc London B:Biol Sci, 2003, 270(Suppl):S96-S99. |
| [11] | Gregory T R. DNA barcoding does not compete with taxonomy[J]. Nature, 2005, 434(7037):1067. |
| [12] | 陈士林, 宋经元, 姚辉, 等. 药用植物DNA条形码鉴定策略及关键技术分析[J]. 中国天然药物, 2009, 7(5):322-327. |
| [13] | 刘美子, 宋经元, 罗焜, 等. DNA条形码序列对9种蒿属药用植物的鉴定[J]. 中草药, 2012, 43(7):1393-1396. |
| [14] | Chen S L, Yao H, Han J P, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS One, 2010, 5(1):e8613. |
| [15] | 李栎, 肖憬, 苏振宇, 等. ITS2条形码序列对茜草科黎药植物的鉴定[J]. 中草药, 2013, 44(13):1814-1818. |
| [16] | Gao T, Yao H, Song J, et al. Evaluating the feasibility of using candidate DNA barcodes in discriminating species of the large Asteraceae family[J]. BMC Evol Biol, 2010, 10(1):324-329. |
| [17] | 张忠廉, 宋美芳, 李海涛, 等. 千斤拔属药用植物DNA条形码鉴定研究[J]. 中草药, 2015, 46(1):118-122. |
| [18] | CBOL Plant Working Group. A DNA barcode for land plants[J]. Proc Natl Acad Sci USA, 2009, 106(31):12794-12797. |
| [19] | Li D Z, Gao L M, Li H T, et al. Comparative analysis of a large dataset indicates that internal transcribed spacer (ITS) should be incorporated into the core barcode for seed plants[J]. Proc Natl Acad Sci, 2011, 108(49):19641-19646. |
| [20] | Hollingsworth P M. Refining the DNA barcode for land plants[J]. Proc Natl Acad Sci USA, 2011, 108(49):19451-19452. |
| [21] | 薛华杰, 闫茂华, 陆长梅, 等. 基于ITS序列的东亚当归属植物的分类学研究[J]. 植物分类学报, 2007, 45(6):783-795. |
| [22] | 辛天怡, 姚辉, 韩建萍, 等. 当归药材及其混伪品ITS2条形码鉴定[J]. 科研信息化技术与应用, 2013, 4(3):76-82. |
| [23] | 张春, 王晓丽, 朱烨, 等. 中药当归及其混伪品的rDNA ITS序列分析与鉴别[J]. 四川农业大学学报, 2011, 29(2):218-224. |
| [24] | Doyle J J, Doyle J L. A rapid DNA isolation prodedure for small quantities of fresh leaf material[J]. Phytochem Bull, 1987, 19:11-15. |
| [25] | Hall T A. A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 1999, 41:95-98. |
| [26] | Sudhir K, Masatoshi N, Joel D, et al. MEGA:Abiologist-centric software for evolutionary analysis of DNA and protein sequence[J]. Brief Bioinf, 2008, 9(4):299-306. |
| [27] | 于华会, 杨志玲, 杨旭, 等. 药用植物种质资源ITS序列研究进展[J]. 中草药, 2010, 41(3):491-495. |
| [28] | 张宏意, 石祥刚. 不同产地何首乌的ITS序列研究[J]. 中草药, 2007, 38(6):911-914. |
| [29] | 陶刚, 刘涛, 朱英, 等. 贵州中药材钩藤属植物的分子鉴定[J]. 中药材, 2008, 31(6):825-828. |
| [30] | 张晓南, 杜春华, 付德欢, 等. 黄草乌及其混淆品ITS序列的分析鉴别[J]. 中药材, 2012, 35(9):1410-1413. |
| [31] | 彭禄, 余岩, 王志新, 等. 基于ITS序列对独活17个种的分子鉴定[J]. 中草药, 2013, 44(12):1648-1653. |
 2016, Vol. 47
2016, Vol. 47


