浙贝母是中国传统中药之一,原产地为宁波象山,现产地主要为鄞州、磐安、南通、东阳等地,是著名的“浙八味”之一,具有清热散结、化痰止咳的功效,主治痰火咳嗽、肺痈、乳痈、疮毒、瘰疬等[1],在祛痰[2]、平喘和镇咳[3]上的效果显著,以及在抗肿瘤[4]及逆转多药耐药方面[5]作用明显。
贝母属植物部分类群的特征性状相似性高,如浙贝母与其近缘品种湖北贝母、东贝母性状相似性高,川贝母与米贝母、砂贝母、太白贝母等相似性较高,且市场流动的贝母多为干燥鳞茎切片,给药用植物的鉴定带来了困难,导致药材品质不一[6]。近年来遗传物质多样性的鉴别方法技术十分热门,包括RAPD、AFLP、ISSR等分子标记方面的研究,都为浙贝母及其贝母属其他植物的物种鉴定提供了可行的方法[7, 8]。
DNA条形码技术是一种建立在PCR技术及DNA测序技术基础之上的新型分子鉴定技术,它利用基因组中一段通用的标准短序列进行物种鉴定及种间亲缘关系的分析[9, 10, 11, 12]。动物DNA条形码的研究已经趋向成熟[13],而在植物界,还未能找到一个能覆盖所有植物,具有高度通用性和高鉴定能力的DNA序列[14, 15]。但已有研究发现,ITS区间在种间的变异距离较大[16],是潜在的优秀DNA条形码候选序列[17, 18]。
本实验通过DNA条形码技术,利用ITS1和ITS2序列对包括浙贝母在内的贝母属主要物种进行物种鉴定及聚类关系分析,评价其鉴定效果。
1 材料与方法 1.1 材料本实验的8种贝母属植物分别为浙贝母F. thunbergii Miq.、暗紫贝母F. unibracteata Hsiao.、卷叶贝母F. cirrhosa D. Don.、梭砂贝母F. delavayi Franch.、湖北贝母F. hupehensis Hsiao.、伊贝母F. pallidiflorae、东贝母F. thunbergii Miq.Var. Chekiangensis.、平贝母F. ussuriensis Maxim.,其中浙贝母包括5个主要栽培品种(多子、狭叶、宽叶、三子、轮叶)及5个变种(变种A、变种B、变种C、变种D、变种E)共21份样品,样品信息及来源见表 1。其中样品P01~P11采集地为宁波市章水镇养殖基地;样品P12采集地为江苏南通市养殖区;样品P13采集地为象山县下峙后村养殖地;样品P14、P15为磐安县新渥镇种植基地采集,其余样品均由宁波中药制药有限公司提供。上述样品均由浙江省中药研究所沈晓霞高级工程师鉴定。
|
|
表 1 贝母样品信息及其来源 Table 1 Samples information and sources |
浙贝母新鲜鳞茎切片后,置于洗净灭菌的塑料方盒中,编号,65 ℃烘干3~5 h。称取新鲜叶片200~300 mg,加入适量液氮充分研磨;称取烘干的鳞茎样品80~100 mg,直接研磨成粉末;所有浙贝母叶片和鳞茎样品使用北京康为世纪公司的植物基因组DNA提取试剂盒提取总DNA。在凝胶成像仪下观察拍照并筛选出提取效果较好的样品用于下一步的PCR扩增。
PCR反应体积为30 μL,体系内包含:ddH2O 17 μL、2×Taq mastermix 10 μL、DNA模板1 μL、正向引物1 μL、反向引物1 μL。ITS1序列正向引物为5’-AAGGATCATTGTCGAGAACCG-3’,反向引物为5’-TTGCGTTCATAGACTCGATGG-3’;ITS2序列正向引物为5’-GCATCGATGAAGAACGCAG-C-3’,反向引物为5’-TCCTCCGCTTATTGATATGC-3’。
将反应体系中的所有成分准确移取至PCR管中并充分混匀后放入PCR仪中。扩增程序:ITS1为94 ℃变性10 min,再进行35个循环(94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸45 s),最后72 ℃延伸10 min。ITS2为94 ℃变性10 min,再进行35个循环(94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸45 s),最后72 ℃延伸10 min。
选择PCR扩增成功的DNA样品交由英潍捷基(上海)贸易有限公司进行测序。
1.2.2 数据处理为了更准确地评价ITS序列的鉴定能力,从GeneBank中下载了12种其他贝母属序列,见表 2。并运用各种基础软件及生物技术软件对DNA测序结果进行分析,其主要分析步骤及所用软件见表 3。除了PC端的软件外,还利用BLAST在线搜索页面(http://blast.ncbi.nlm.nih. gov/ Blast.cgi?PROGRAM=blastn&BLAST_PROGRAMS=megaBlast&PAGE_TYPE=BlastSearch&SHOW_DEFAULTS=on&LINK_LOC=blasthome)对测序结果进行初步搜索,确认测序准确性。在序列信息分析步骤,运用SMS网站的在线DNA和蛋白质序列处理工具(http://www.91bio.com/ SMS2/dna_stats.htmL),对各个序列的碱基组成、G+C量进行计算。
|
|
表 2 12种其他贝母属序列信息 Table 2 Sequence information of 12 species of Fritillaria L. downloaded from GeneBank |
|
|
表 3 数据处理及软件 Table 3 Data processing and software |
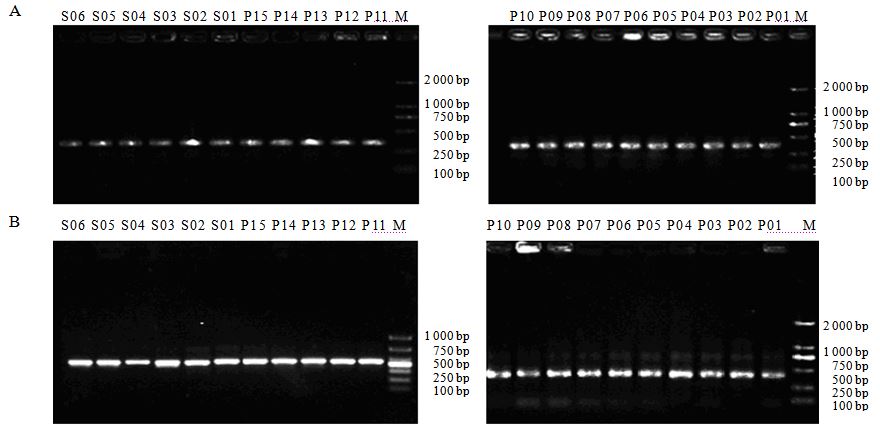
本实验对10份叶片样品,6份鳞茎干品和5份鳞茎鲜品提取了总DNA,电泳鉴定结果见图 1,DNA长度在15 000 bp左右,与预期片段大小相符。挑选出条带较完整清晰的DNA样品,进行ITS1、ITS2序列的PCR扩增,ITS1和ITS2序列扩增后的片段长度分别为400 bp和350 bp左右,部分样品的PCR产物电泳图见图 2。整理PCR扩增成功的DNA样品交由英潍捷基(上海)贸易有限公司进行测序。

|
图 1样品DNA提取产物电泳图 Fig. 1 Electrophoregram of DNA extracted from some samples |
|
图 2样品中ITS1 (A)和ITS2 (B) 序列的PCR产物电泳图 Fig. 2 Electrophoregram of PCR products from samples ITS1 (A) and ITS2 (B) |
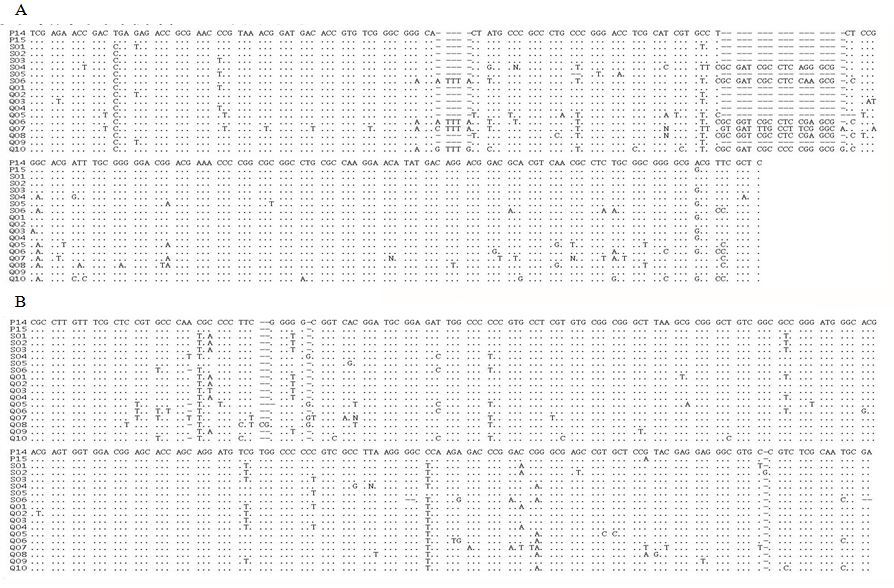
对测序所得序列进行修剪,ITS2序列去除两端5.8 S和26 S区段,ITS1序列去除两端18 S和5.8 S区段,获得ITS1、ITS2间隔区序列。其中ITS1序列的片段长度在205~229 bp,ITS2序列的片段长度在237~244 bp。选取本实验材料中的8个不同种贝母(P14、P15、S01、S02、S03、S04、S05、S06)加上12条网上下载的贝母序列,计算保守位点、信息位点、突变位点及转换/颠换值,通过Blast1搜索法计算序列的鉴定效率,计算结果见表 4,通过MEGA的Sequence Data Explorer功能比对序列信息见图 3。
|
|
表 4 ITS1和ITS2序列基本信息 Table 4 Main information of ITS1 and ITS2 sequences |
|
图 3贝母属不同物种ITS1 (A) 和ITS2 (B) 序列对比图 Fig. 3 Sequence comparison on ITS1 (A) and ITS2 (B) of different species of Fritillaria L. |
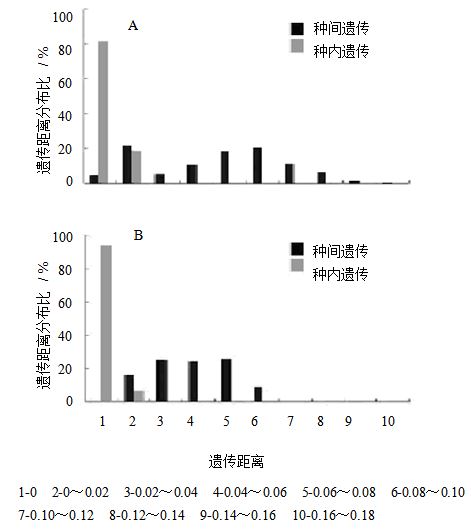
通过MEGA软件分别计算ITS1、ITS2序列种内(浙贝母)及种间(贝母属不同物种)的遗传距离,基于Kimura 2-parameter(K2-P)距离模型,建立种内及种间遗传距离矩阵[19]。通过Overall mean distance算法,计算出ITS1序列的种内及种间平均遗传距离分别为0.001 8和0.066 1,而ITS2序列的种内及种间平均遗传距离分别为0.000 5和0.046 8。
再利用Matlab软件结合遗传距离矩阵的数据,计算ITS1、ITS2序列在种内及种间的遗传距离分布,生成遗传距离分布柱状图,该柱状图代表了条形码序列两两对比的遗传距离在各距离范围内所占的比值,体现了该DNA条形码序列在不同物种中遗传距离的分散度和在同一物种中的稳定度。
从ITS1、ITS2序列遗传距离分布图可见(图 4),ITS1序列的种内与种间变异分布重叠度较高,其种内各样品遗传距离在0~0.02的序列占比高达18.75%,种间则为21.57%,表明ITS1序列在种内的稳定性不高,重叠度较高,影响鉴定效果。使得种间遗传距离在0.10~0.18内均有分布。ITS2的遗传距离高峰集中在0.04~0.08,种内种间遗传距离分布重叠度较低。ITS2序列种内高度保守,变异序列仅占6.25%。ITS1序列过大的遗传距离可能使其聚类分析结果远离同属物种而接近近缘属物种,而ITS2序列更为保守稳定,更适合作为条形码序列。
|
图 4 ITS1 (A) 和ITS2 (B) 序列种内及种间遗传距离分布 Fig. 4 Intraspecific and interspecific genetic distance distribution of ITS1 (A) and ITS2 (B) |
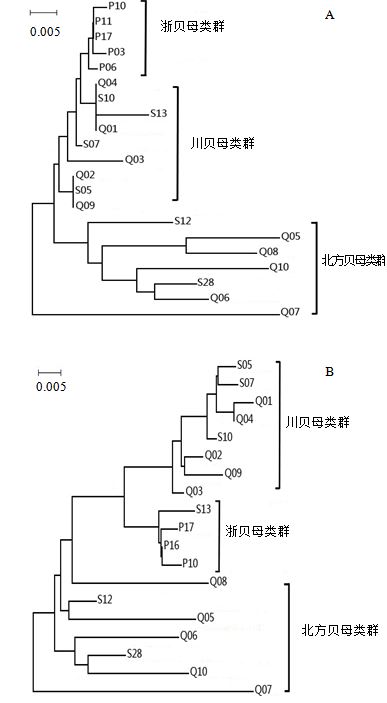
选取本实验材料中的8个不同种贝母(P14、P15、S01、S02、S03、S04、S05、S06)与种内发生变异的浙贝母序列(P03、P07、P10),加上12条网上下载的贝母序列,运用MEGA 4.0软件,基于K2-P距离模型进行ITS1、ITS2序列的NJ树构建,并进行Bootstrap consensus tree显著性筛选[20]。结果见图 5。
|
图 5 基于ITS1 (A) 和ITS2 (B) 序列构建的NJ树 Fig. 5 NJ tree based on ITS1 (A) and ITS2 (B) sequences |
对比ITS1序列与ITS2序列所构建的NJ树,发现ITS1序列在浙贝母中的遗传稳定性较差,样品P03、P07、P10均有一定程度变异,其亲缘关系接近于P15,且ITS1系统发育树中,浙贝母类群被划分为川贝母类群的一个分支,而川贝母类群中的S03和甘肃贝母(GQ205115.1)、S01和大金贝母(GQ205114.1)等均由于序列无差异而难以被区分。ITS2序列构建的聚类直观图,在区分北方贝母类群、川贝母类群、浙贝母类群的效果明显,区分度和准确度高,不同贝母类群的变异速度较均匀。但是无论是ITS1序列还是ITS2序列,都难以区分主产于新疆地区的阿尔泰贝母、伊贝母等和主产于东北地区的平贝母、轮叶贝母等。推测其两类居群为同一个祖先且在相似环境中遗传距离较小,差异较少。通过考察各个贝母物种的地理分布和形态学差异[22],对比NJ树聚类分析结果认为ITS2在其物种鉴定及聚类分析效果上具有简单、准确、高效的特点,在大量样本的物种鉴定中,具有一定的优势。
3 讨论 3.1 种内遗传情况对比将本实验计算得出的ITS1与ITS2序列的平均遗传距离、可变位点占比,与柑橘属DNA条形码鉴定[20]中的实验结果进行对比,发现同为ITS区域的序列,贝母属的平均遗传距离ITS1为0.066 1、ITS2为0.046 8,要高于柑橘属的0.030 5和0.025 7。但是信息位点占比ITS1为11.8%、ITS2为8.2%,略低于柑橘属的14.7%和14.6%。原因可能是选取的贝母属物种聚类关系较分散,而文献中柑橘属样品含有较多的近缘物种,聚类关系较为集中,但两者都表现出ITS1序列的信息位点及平均遗传距离要高于ITS2序列的特点。matK与rbcL序列种间差异则显著低于ITS区域序列。
3.2 ITS1与ITS2的鉴定效果对比本实验中,单一片段的鉴定能力中以ITS2的鉴定效率最高,为100%;ITS1序列在区分属内近缘物种的能力较差,模糊鉴定率高达27.8%。在早期的研究中已发现,ITS2序列在物种水平上的遗传稳定性高、种间差异稳定、对近缘物种的区分度高,在物种鉴定上的综合能力高于ITS1序列,在柑橘属[20]、木兰属[23]、鸟足兰属[23]等均达到鉴定成功率在90%以上;在蔷薇科[24]、豆科[26]、菊科[27]等较为庞大的科系中,鉴定效率有所下降,如菊科的鉴定效率仅为76%。即使如此,也远高于其他条形码候选序列的鉴定效果。
3.3 系统发育树的构建与物种聚类情况分析结合不同贝母在地理分布、形态特征上的差异,并通过系统发育树对贝母属不同物种的聚类能力进行对比,认为ITS2构建的NJ树、ITS1与ITS2组合序列构建的NJ树对参与实验物种的聚类情况分析准确度最高,能在一定程度上准确判断不同地理位置、形态特征的贝母种的遗传距离,区分浙贝母类群、川贝母类群、北方高纬度地区贝母类群3个中国贝母主要类群。但是对于新疆地区贝母和东北地区的贝母无明显区分能力。
本实验的聚类结果与4种浙贝母与其他品种贝母的RAPD分析结果[28]相一致,伊贝母区别于南方贝母类群,而南方贝母类群分成了川贝母系与浙贝母系。
| [1] | 中国药典[S]. 一部. 2010. |
| [2] | 汪丽燕, 韩传环. 皖贝、川贝和浙贝止咳化痰的药理作用比较[J]. 安徽医学, 1993, 14(3): 57-58. |
| [3] | 李 萍. 鄂贝定碱的镇咳作用研究[J]. 中草药, 1993, 24(9): 475-477. |
| [4] | 谢艳华, 孙纪元, 王四旺, 等. 二贝母胶囊抑瘤作用[J]. 第四军医大学学报, 2003, 24(16): 1463-1465. |
| [5] | 胡凯文, 郑洪霞. 浙贝母碱逆转白血病细胞多药耐药性的研究[J]. 中华血液学杂志, 1999, 20(12): 650. |
| [6] | 丁丽娟. 川贝母及其常见伪品鉴别[J]. 成都中医药大学学报, 2009, 32(2): 83-84. |
| [7] | Wang L Q, Dai X Z, Li X F. Genetic Diversity of Velvetleaf Germplasm by RAPD and ISSR Markers[J]. J Plant Gen Resour, 2009(1): 126-132. |
| [8] | Powell W, Morgante M, Andre C. The comparison of RFLP, RAPD, AFLP and SSR (microsatellite) markers for germplasm analysis[J]. Mol Breeding, 1996, 2(3): 225-238. |
| [9] | Chen S L, Yao H, Han J P, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS One, 2010, 5(1): e8613. |
| [10] | 石召华, 陈士林, 姚 辉, 等. 娑罗子基原物种的DNA条形码鉴定研究[J]. 中草药, 2013, 44(18): 2593-2599. |
| [11] | Casiraghi M, Labra M, Ferri E, et al. DNA barcoding: a six-question tour[J]. Brief Biginform, 2010, 11(4): 440-453. |
| [12] | 孙稚颖, 陈士林, 姚 辉, 等. 基于ITS2序列的羌活及其混伪品的DNA条形码鉴定[J]. 中草药, 2012, 43(3): 568-571. |
| [13] | Alex Smith M, Wood M, Janzen D H. DNA barcodes affirm that 16 species of apparently generalist tropical parasitoid flies (Diptera, Tachinidae) are not all generalists[J]. Cur Issue, 2007, 104(12): 4967-4972. |
| [14] | 任保青, 陈之端. 植物DNA条形码技术[J]. 植物学报, 2010, 45(1): 1-12. |
| [15] | Pettengill J B, Neel M C. An evaluation of candidate plant DNA barcodesand[J]. Am J Bot, 2010, 97(8): 1391-1406. |
| [16] | Pang X H, Song J Y, Zhu Y J, et al. Applying plant DNA barcodes for Rosaceae species identification[J]. Cladistics, 2011, 27(2): 165-170. |
| [17] | Chiou S J, Yen J H, Fang C L, et al. Authentication of medicinal herbs using PCR-amplified ITS2 with specific primers[J]. Planta Med, 2007, 73(13): 1421-1426. |
| [18] | Hollingsworth P M, Graham S W, Little D P. Choosing and Using a Plant DNA Barcode[J]. PLoS One, 2011, 6(5): e19254. |
| [19] | 陈士林. 中药DNA条形码: 分子鉴定[M]. 北京: 人民卫生出版社, 2012. |
| [20] | 于 杰. 柑橘及其近缘属植物DNA条形码研制及其物种的鉴定研究[D]. 重庆: 西南大学, 2011. |
| [21] | Lahaye R, Bank M V D, Bogarin D. DNA barcoding the floras of biodiversity hotspots[J]. PNAS, 2008, 105(8): 2923-2928. |
| [22] | 唐 进, 汪发缵. 中国植物志(第14卷)[M]. 北京: 科学出版社, 2010. |
| [23] | 罗 焜, 陈士林, 陈科力. 基于芸香科的植物通用DNA条形码研究[J]. 中国科学: 生命科学, 2010, 40(4): 342-358. |
| [24] | Pang X H, Song J Y, Zhu Y J, et al. Using DNA barcoding to identify species within Euphorbiaceae[J]. Planta Med, 2010, 76: 1784-1786. |
| [25] | Gao T, Yao H, Song J Y, et al. Identification of medicinal plants in the family Fabaceae using a potential DNA barcode ITS2[J]. Ethnopharmacology, 2010, 130: 116-121. |
| [26] | Gao T, Yao H, Song J Y, et al. Evaluating the feasibility of using candidate DNA barcodes in discriminating species of the large Asteraceae family[J]. BMC Evol Biol, 2010, 10: 324-331. |
| [27] | 陆 含, 朱世华, 周书记. 浙贝母4品种及5种贝母遗传多样性的RAPD分析[J]. 宁波大学学报: 理工版, 2009, 22(1): 44-47. |
 2014, Vol. 45
2014, Vol. 45


