2. 湖南农业大学 兽用中药资源与中兽药创制国家地方联合工程研究中心, 湖南 长沙 410128;
3. 湖南农业大学动物医学院, 湖南 长沙 410128;
4. 湖南中医药大学药学院, 湖南 长沙 410128
2. National and Local Union Engineering Research Center for the Veterinary Herbal Medicine Resources and Initiative, Hunan Agricultural University, Changsha 410128, China;
3. Veterinary Faculty of Hunan Agricultural University, Changsha 410128, China;
4. Pharmacy college of Hunan University of Chinese Medicine, Changsha 410128, China
长久以来,酵母一直被广泛应用于人类的生活,如食品生产(酿酒、面包)等方面。酵母菌具有安全可靠、培养周期短、生长迅速、操作简单等特点,且相较于原核生物,它能够识别真核基因帮助其进行转录翻译后修饰,表达的外源蛋白更接近于天然蛋白质,并将蛋白分泌至胞外,能弥补原核表达系统在表达真核基因方面的不足等优点。随着酵母基因组的解析和重组DNA技术的不断进步,目前酵母被应用于更加广泛的方面,如生产燃料、化学物质、胰岛素、人血清白蛋白等[1-2],近年来更被用于植物次生代谢产物合成基因的异源表达和生产高价值的次生代谢产物[3-5],如Ro等[6]和Paddon等[7]在酵母中合成青蒿素重要前体青蒿酸,Galanie等[8]在酵母中从葡萄糖开始合成吗啡,Fossati等[9]通过在酵母中重组10个基因合成重要的苄基异喹啉类生物碱(BIAs)类生物碱血根碱等。由于酵母异源表达体系适合许多植物中的酶,如细胞色素P450、氧化还原酶等,因此大量的天然产物合成基因的验证工作都是在酵母体内完成的[9-12]。
BIAs是分布于被子植物中的一类重要次生代谢产物,是一些重要医用药物的主要成分之一,比如吗啡、帝巴因、可待因、罂粟碱、小檗碱和血根碱。目前BIAs生物合成基因的主要来源植物是罂粟Papaver somniferum L.、花菱草Eschscholzia californica Cham.、墨西哥蓟罂粟Argemone Mexicana L.、日本黄连Coptis japonica Franch.[13]以及博落回属Macleay spp.植物[14]等。近些年来BIAs在合成生物学上取得了许多突破性的进展,Hawkins等[15]通过构建异源表达载体使得一些苄基异喹啉生物碱在酵母中实现表达,转入酵母菌株中的功能酶来源于罂粟、花菱草、日本黄连3种植物,它们通过对不同来源的功能酶组合使得网状番荔枝碱产量从10 mg/L增加到150 mg/L。另外,Fossati等[9]将罂粟中血根碱合成通路上的10个基因重组到酿酒酵母中,并能从(R,S)-去甲劳丹碱产生二氢血根碱及其氧化衍生物血根碱,这项工作也是当今最长的重组到酵母中的生物碱通路并且证明了微生物系统生产高价值生物碱的可行性。然而,酵母作为基因功能验证的受体细胞依然存在以下问题:(1)一些复杂的P450酶很难在酵母中表达[16],有些酶虽然能够在酵母中表达但是蛋白表达量很低[9]。(2)酵母中缺乏合成某些目标化合物前体的能力,如生物碱[15]。因此需要对酵母进行复杂的工程改造或进行前体饲喂[17-19],而不同的前体饲喂方式也会影响酵母生物转化的产量[20]。因此,找到一种高效的酵母前体饲喂方式对于验证基因功能来说具有十分重要的意义。本研究以罂粟原阿片碱6-羟化酶(P6H)催化原阿片生成二氢血根碱为例,首先通过合成罂粟中已验证的PsP6H基因和PsCPR基因,并将2个基因重组到酵母中构建工程菌。然后通过4种不同的前体饲喂方式饲喂工程菌,最后通过质谱检测下游产物二氢血根碱来判断哪种前体饲喂方法二氢血根碱量最高。
1 材料 1.1 菌种和质粒酵母表达载体(PYES2)和酵母菌株(ivf)购自Thermofisher,大肠杆菌购自天根生物有限公司。
1.2 PCR引物设计引物序列见表 1,根据infusion引物的设计原则分别设计PsP6H、PsCPR基因的引物,由上海生工生物工程股份有限公司合成。
|
|
表 1 PsP6H和PsCPR基因扩增和同源重组引物序列 Table 1 Primers used for amplification and homologous recombination of PsP6H and PsCPR genes |
1.3 试剂及抗生素
质粒提取试剂盒、琼脂糖凝胶DNA回收试剂盒购自天根生物有限公司,Taq酶、infusion酶购自宝生物工程有限公司,Q5酶、限制性内切酶KpnI-HF、XbaI购自New England Biolabs(NEB)公司。原阿片碱、二氢血根碱由本实验从博落回植物中分离纯化,质量分数均大于96%。氨苄青霉素、醋酸锂、PEG 3350、Trisbase、EDTA购自Sigma公司。鲑鱼精(H1060)购于索莱宝生物科技有限公司。基因PsP6H(GenBank KC154002)、PsCPR(GenBank KF661328)序列信息来源于NCBI,由苏州金唯智生物科技有限公司合成。
2 方法 2.1 培养基的配制 2.1.1 LB培养基1 L超纯水,10 g蛋白胨,5 g酵母提取物,10 g NaCl,pH 5.8。
2.1.2 YPDA液体培养基10 g酵母提取物,20 g Bacto-蛋白胨,20 g葡萄糖,0.03 g腺嘌呤,1 L超纯水,pH 5.8。
2.1.3 SD/Dropout培养基6.7 g无氨基酸酵母氮源(YNB),相应的SD/Dropout,20 g葡萄糖(或者半乳糖),1 L超纯水,pH 5.8。
2.2 母液的配制 2.2.1 氨苄青霉素(Amp)母液(100 mg/mL)1 g Amp溶于无菌蒸馏水,定容至10 mL,0.22 μm滤膜滤过除菌,分装成每管1 mL,-20 ℃保存。
2.2.2 50% PEG母液5 g PEG 3350溶于无菌蒸馏水,定容至10 mL,0.22 μm滤膜滤过除菌,分装成1 mL每管,-20 ℃保存。
2.2.3 1 mol/L LiAc母液10.2 g醋酸锂溶于9 mL无菌蒸馏水,用稀冰醋酸调pH至7.5,定容至10 mL,0.22 μm滤膜滤过除菌,分装成每管1 mL,-20 ℃保存。
2.2.4 TE(Tris+EDTA)母液1.21 g Trisbase+0.37 g EDTA,溶解于90 mL无菌蒸馏水,用1 mol/L NaOH调pH至5.8或8.0,补蒸馏水至100 mL。
2.2.5 原阿片碱母液精密称取原阿片碱对照品3.00 mg于1.5 mL离心管中,用1 mL DMSO溶解即得原阿片碱母液。
2.2.6 二氢血根碱母液精密称取二氢血根碱对照品适量,加甲醇制成质量浓度为4.0 mg/L的母液,即得二氢血根碱母液。
2.3 基因克隆和表达载体的构建Q5酶克隆反应体系:Q5 Reaction Buffer,40 μL;10 mmoL/L dNTP Mix,4 μL;正向引物10 μL;反向引物10 μL;cDNA,8 μL;Q5 High Gc Enhancer,40 μL;Q5 DNA聚合酶,2 μL;ddH2O,86 μL;总体积200 μL。轻轻混匀,短暂离心5 s,平均分装至4个PCR管中(每管50 μL)。扩增条件:98 ℃预变性30 s,98 ℃、10 s,58 ℃、30 s,72 ℃、1 min,72 ℃、2 min,4 ℃保持。扩增完成后,用琼脂糖凝胶DNA回收试剂盒回收基因。另外,用限制性内切酶KpnI-HF、XbaI对Leu与Ura空质粒进行双酶切(37 ℃,30 min),酶切反应结束后用琼脂糖凝胶DNA回收试剂盒回收线性载体。基因和线性载体都回收完成后,用infusion同源重组酶连接目的基因与线性载体(50 ℃,20 min),转入大肠杆菌感受态中,过夜培养。第2天挑阳性菌落进行菌落PCR(Taq酶),根据琼脂糖凝胶电泳结果,挑取与目的基因大小匹配的菌落过夜培养,次日提取质粒并送测序。
2.4 酵母转化首先挑取ivf的单菌落于3 mL YPDA液体培养基中,30 ℃,200 r/min过夜培养。第2天用新的YPDA液体培养基稀释过夜培养的菌液,600 nm检测吸光度(A600),控制A600于0.3,30 ℃,200 r/min继续培养3 h左右(A600为0.4~0.6)。3 h后,用离心机以5 000 r/min离心5 min,收集菌体,用1 mL无菌水重悬后再次用离心机以5 000 r/min离心5 min,收集菌体,用1 mL 1×LiAc-1×TE-40% PEG3350重悬菌体。
根据转化样品数目准备灭菌的1.5 mL离心管,每管加入100 μL上一步重悬起来的菌液,再每管都加入0.1 μg DNA,10 μL鲑鱼精和700 μL 1×LiAc-1×TE-40% PEG3350,充分震荡混匀。放于摇床30 ℃,200 r/min,摇动30 min。然后每管加入88 μL无菌DMSO,轻轻混匀,42 ℃放置7 min。7 min后,用离心机以12 000 r/min离心20 s,弃上清,用1 mL 1×TE重悬后以同样的方式再次离心,弃上清,达到清洗的目的。最后,用50 μL无菌水或是1×TE重悬菌体,涂于相应的SD/Dropout培养皿上。长出菌落后,挑选酵母单菌落摇菌,进行下游验证。
2.5 酵母前体饲喂 2.5.1 前体饲喂方法(1)在超净工作台挑取PsP6H+PsCPR酵母单菌落于装有2 mL尿嘧啶与亮氨酸葡萄糖缺陷培养基的15 mL无菌离心管中,放于摇床,30 ℃,200 r/min过夜培养。(2)培养液用离心机以5 000 r/min离心1 min,收集菌体。用1 mL Ura与Leu半乳糖缺陷培养基重悬菌体,加入到装有19 mL Ura与Leu半乳糖缺陷培养基的100 mL无菌三角瓶中培养。(3)检测A600值,按照比例用半乳糖培养液将菌液A600值稀释至0.8,总体积为10 mL,将A600=0.8的培养液继续培养7 h。(4)7 h后,培养液用离心机以5 000 r/min离心5 min,弃上清,加入前体溶液,震荡使菌体重悬,继续培养。
2.5.2 测试样本提取(1)培养液用15 000 r/min离心1 min,将上清转移至5 mL离心管中,剩下菌体再提取。(2)菌体中加入500 μL甲醇,100 μL玻璃珠,震荡涡旋10 min。再用5 000 r/min离心5 min,吸取上清于上一步的5 mL离心管中。将提取液经0.22 μm滤膜滤过,待上机检测。
2.5.3 空白对照样本制备空白菌株为转入空质粒酵母菌株,培养以及提取方法与上述方法相同。
2.6 不同半乳糖诱导时间前体饲喂在前体饲喂步骤(2)中,分别将菌株在100 mL无菌三角瓶中培养24 h和31 h(培养24 h后再稀释A600至0.8再继续培养7 h,共31 h)。每组样品平行重复3份。
2.7 不同培养方式下前体饲喂 2.7.1 半乳糖培养方式下前体饲喂方案在“2.5.1”项前体饲喂的步骤(4)中,将分离后的菌体转入总体积为2 mL含有终浓度为10 μmol/L的原阿片碱半乳糖培养液中继续培养24 h。样品平行重复3份。
2.7.2 TE溶液培养方式下饲喂前体方案在“2.5.1”项前体饲喂的步骤(4)中,将分离后的菌体转入总体积为2 mL含有终浓度为10 μmol/L的原阿片碱TE溶液中继续培养24 h。样品平行重复3份。
2.8 不同pH溶液中前体饲喂在“2.5.1”项前体饲喂的步骤(4)中,分别加入pH为5.8和8.0的总体积为2 mL含有终浓度为10 μmol/L的原阿片碱TE溶液继续培养24 h。每组样品平行重复3份。
2.9 不同的孵育时间前体饲喂在“2.5.1”项前体饲喂的步骤(4)中,将酵母在总体积为2 mL含有终浓度为10 μmol/L的原阿片碱TE溶液中分别培养24 h和48 h。每组样品平行重复3份。
2.10 二氢血根碱的测定[21] 2.10.1 色谱条件华谱XAqua C18色谱柱(150 mm×2.1 mm,5 μm),流动相为0.1%甲酸水(A)-0.1%甲酸乙腈(B);柱温35 ℃;检测波长287 nm;体积流量0.2 mL/min;进样量2 μL。
2.10.2 标准曲线的制备精密吸取适量二氢血根碱母液,按比例稀释成质量浓度梯度为2.0、1.0、0.5、0.2、0.1、0.02、0.01 mg/L的对照品溶液,采用UPLC Q-TOF MS/MS进行测定,记录色谱峰面积。以峰面积积分值为纵坐标(Y),质量浓度为横坐标(X)绘制标准曲线,得到回归方程Y=432.78 X-531.24,r2=0.999 7,线性范围为0.01~2.0 mg/L。
3 结果 3.1 目的基因的克隆与表达载体构建以合成基因PsP6H和PsCPR质粒为模板,通过RT-PCR扩增。结果显示分别获得特异扩增条带PsP6H(1 626 bp)和PsCPR(2 052 bp)与预期大小相一致(图 1)。经切胶回收后将纯化PCR产物通过同源重组酶(infusion)与线性载体PYES2-ura、PYES2-leu进行连接。并转入感受态大肠杆菌中,在Amp抗性板上过夜培养,次日挑取阳性单克隆菌落进行菌落PCR。结果显示分别获得与目的条带相符合的阳性菌株。将阳性菌株在Amp培养基中过夜培养提取质粒送测序检测得到完全匹配目的质粒用于下一步酵母转化。
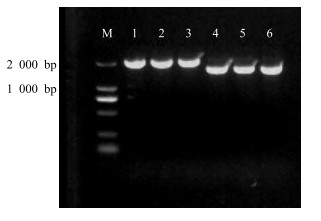
|
M-Marker 1~3-PsCPR基因4~6-PsP6H基因 M-Marker 1-3-PsCPR gene 4-6-PsP6H gene 图 1 PsP6H和PsCPR基因扩增电泳图 Fig.1 Electrophoregram of PsP6H and PsCPR genes amplification |
3.2 酵母转化子营养缺陷筛选
将PYES2-Leu重组质粒单独转入酵母(ivf)菌株中,PYES2-PsP6H组合PYES2-PsCPR重组质粒转入酵母(ivf)中获得酵母工程菌株PSY-1000(PYES2-Leu),PSY1001(PYES2-PsP6H+PYES2-PsCPR),分别在亮氨酸缺陷以及亮氨酸与尿嘧啶缺陷的SD-U选择培养基上培养48 h,得到直径约1 mm的单菌落。挑取单菌落酵母在相应缺陷培养基中过夜培养用于下一步前体饲喂。
3.3 酵母前体饲喂方法的影响 3.3.1 不同半乳糖诱导时间下前体饲喂的影响半乳糖的诱导时间也是对前体饲喂结果产生影响的因素之一,选择了2种诱导时间一种是24 h半乳糖诱导,另一种是24 h半乳糖诱导之后测定所有酵母的A600值,并统一稀释到0.8然后再诱导7 h。确定方法后,把酵母工程菌PSY-1000、PSY-1001在以上的不同方法中进行诱导,诱导结束后再在TE溶液中继续孵育24 h。反应结束后用UPLC-Q-TOF MS/MS检测不同诱导时间处理酵母提取物中二氢血根碱的量,对结果进行统计学分析,经过半乳糖诱导24 h后再稀释A600值为0.8再继续诱导7 h的方法产生的二氢血根碱的量为(0.430±0.008)mg/L,高于半乳糖诱导24 h的量(0.314±0.009)mg/L(图 2)。经SPSS 19.0软件分析,P < 0.05,两者差异显著,实验结果具有统计学意义。酵母工程菌PSY-1000在所有条件下饲喂前体后均未产生二氢血根碱,证明酵母本身不会对实验产生影响。
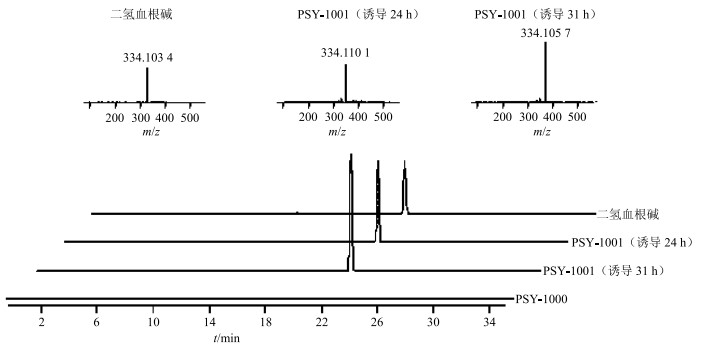
|
图 2 不同半乳糖诱导时间下酵母前体饲喂中二氢血根碱的UPLC-Q-TOF MS/MS分析 Fig.2 Analysis on dihydrosanguinarine at different time of galactose induction cell feeding in S. cerevisiae by UPLC-Q-TOF MS/MS |
3.3.2 不同培养方式下前体饲喂的影响
酵母工程菌PSY-1000、PSY-1001经过24 h半乳糖诱导后在含有同等浓度原阿片碱的TE缓冲液与半乳糖培养基中继续孵育24 h。反应结束后用UPLC Q-TOF MS/MS分析不同培养方式下的酵母提取物中二氢血根碱的量,对结果进行统计学分析,TE溶液培养方式中的二氢血根碱的量为(0.498±0.012)mg/L,高于半乳糖培养中二氢血根碱的量(0.282±0.009)mg/L(图 3)。经SPSS 19.0软件分析,P < 0.05,两者差异显著,实验结果具有统计学意义。酵母工程菌PSY-1000在2种培养方式中饲喂前体后均未产生二氢血根碱,证明酵母本身不会对实验产生影响。
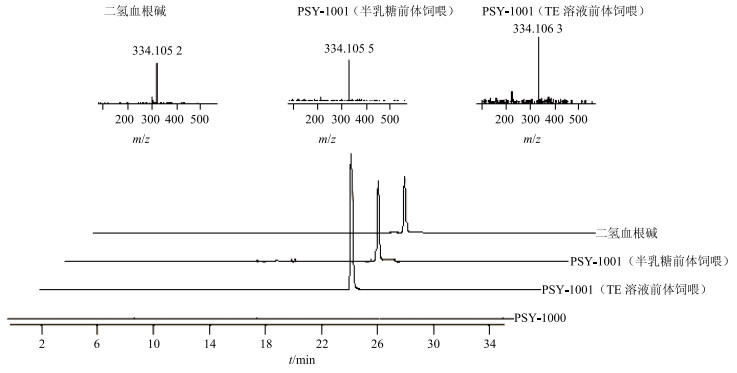
|
图 3 不同培养方式下酵母前体饲喂中二氢血根碱的UPLC-Q-TOF MS/MS分析 Fig.3 Analysis of dihydrosanguinarine at different cultivation cell feeding in Saccharomyces cerevisiae by UPLC-Q-TOF MS/MS |
3.3.3 不同pH值环境下前体饲喂的影响
在确定了TE缓冲液为最佳培养条件后,后期的前体饲喂方法均采用TE缓冲液的方法。为找到最适合进行前体饲喂的TE溶液pH值,把酵母工程菌PSY-1000、PSY-1001在含有同等浓度原阿片碱的pH值为5.8和8.0的TE溶液中继续孵育24 h。反应结束后用UPLC Q-TOF MS/MS分析不同pH值条件下的TE溶液中酵母提取物中二氢血根碱的量,对结果进行统计学分析,pH值为8.0的TE溶液中二氢血根碱的量为(0.382±0.017)mg/L,高于pH为5.8的TE溶液中二氢血根碱的量(0.331±0.018)mg/L(图 4)。经SPSS 19.0软件分析,P < 0.05,两者差异显著,实验结果具有统计学意义。酵母工程菌PSY-1000在2种pH值条件下饲喂前体后均未产生二氢血根碱,证明酵母本身不会对实验产生影响。
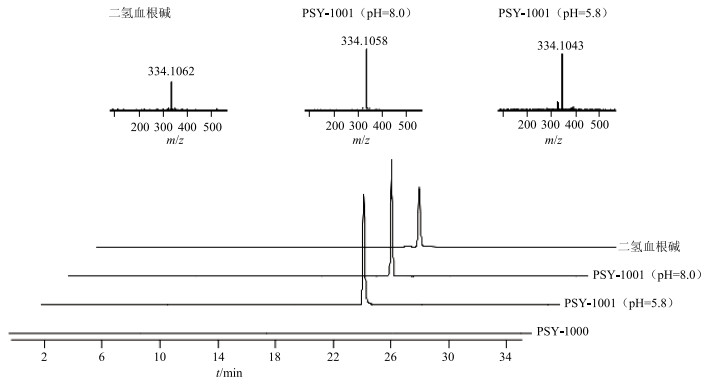
|
图 4 不同pH值环境下酵母前体饲喂中二氢血根碱的UPLC-Q-TOF MS/MS分析 Fig.4 Analysis of dihydrosanguinarine at different pH cell feeding in Saccharomyces cerevisiae by UPLC-Q-TOF MS/MS |
3.3.4 不同的孵育时间下前体饲喂的影响
选择了24 h和48 h 2个时间进行比较。酵母工程菌PSY-1000、PSY-1001在经过同等浓度原阿片碱的TE溶液中按照以上两个时间长度孵育。反应结束后用UPLC-Q-TOF MS/MS分析经过不同时间孵育的酵母提取物中二氢血根碱的量,对结果进行统计学分析,孵育24 h产生的二氢血根碱的量为(0.485±0.011)mg/L,高于孵育48 h所产生的二氢血根碱的量(0.256±0.021)mg/L(图 5)。经SPSS19.0软件分析,P < 0.05,两者差异显著,实验结果具有统计学意义。酵母工程菌PSY-1000在所有条件下饲喂前体后均未产生二氢血根碱,证明酵母本身不会对实验产生影响。
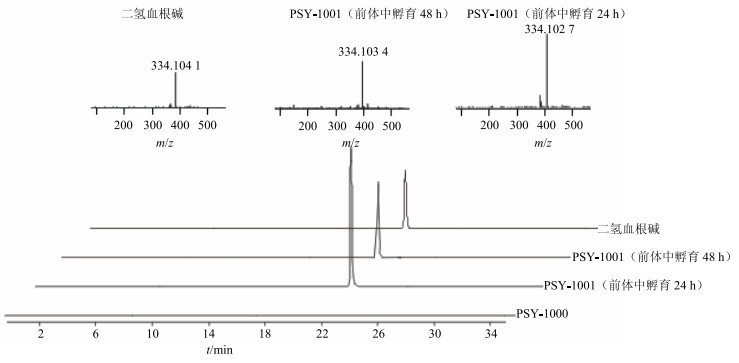
|
图 5 不同的孵育时间下酵母前体饲喂中二氢血根碱的UPLC-Q-TOF MS/MS分析 Fig.5 Analysis of dihydrosanguinarine at different incubation time cell feeding in Saccharomyces cerevisiae by UPLC Q-TOF MS/MS |
4 讨论
近年来,大量的植物次生代谢产物合成通路基因得到了功能验证。为植物次生代谢产物的异源表达和工程化菌株的构建奠定了研究基础。长期以来,酵母广泛应用于植物次生代谢产物的异源表达,被认为是实现新的资源获取途径的重要途径之一。本实验在前人实验方法[9, 15, 20]的基础上进行改进,设计出4种不同的酵母前体饲喂方式并进行比较,最终找到了最适合的一种酵母前体饲喂方式:首先将酵母在葡萄糖培养基中进行过夜慢摇,第2天在酵母生长充分后便转入半乳糖培养基中诱导和扩大培养24 h,经过24 h培养后统一测定所有酵母的A600值,然后再根据测得的稀释A600值到0.8。这一步的目的是使所有工程菌株样本都处于同一个水平的浓度,然后在这一浓度再经过7 h的半乳糖培养后分离培养基上清和菌体,得到菌体后便开始进行前体饲喂。前体用DMSO溶解后计算取一定量加入到2 mL的pH=8.0的TE溶液中进行稀释,再将稀释后的前体TE溶液全部加入到酵母菌中并重悬。然后放置到摇床上过夜孵育24 h。结束后便对酵母菌体进行提取然后上UPLC-Q-TOF MS/MS进行检测。
另外,本课题组对这4种不同的前体饲喂方式产生效果不同的原因进行了分析。首先,酵母在培养基中前体饲喂产生的下游产物量低有2个原因,一个是前体孵育的时间过长可能导致前体化合物发生变化,影响了酵母转化的效率。另外与培养基的pH值有关,在不同的pH条件下饲喂的实验中本课题组已经证实酵母在pH 5.8时所产生的下游产物要低于pH 8.0,这也许跟酵母细胞在pH 8.0的环境下细胞膜的通透性有关,在pH 8.0时酵母更容易与外界物质进行交换。由于实验酵母使用的是半乳糖诱导型启动子,所以酵母只有在半乳糖诱导培养基的作用下外源基因才能表达,而诱导时间并非时间越长越好,因为酵母中异源基因的表达量是有上限的。因此发现诱导24 h后再统一菌种浓度诱导7 h是最合适的诱导时间长度。而孵育时间的长短也并非越长越好,因为酵母中表达的异源蛋白也是有消耗的,当进入平台期后随着时间的推移并不会对转化率有帮助,而转化生成的化合物如果不稳定的话可能会转化生成其他的物质,这也会对计算外源基因的转化率产生影响。本方法为酵母体内验证植物次生代谢产物合成基因前体饲喂提供方法学指导,采用本方法优化后的前体饲喂酵母验证比较高效稳定,重复性好。
| [1] | Nielsen J B. Yeast cell factories on the horizon[J]. Science, 2015, 349(6252): 1050–1051. DOI:10.1126/science.aad2081 |
| [2] | Billingsley J M, Denicola A B, Tang Y. Technology development for natural product biosynthesis in Saccharomyces cerevisiae[J]. Curr Opin Biotechnol, 2016, 42: 74–83. DOI:10.1016/j.copbio.2016.02.033 |
| [3] | Mills E, Truong K. Ca2+-mediated synthetic biosystems offer protein design versatility, signal specificity, and pathway rewiring[J]. Chem Biol, 2011, 18(12): 1611–1619. DOI:10.1016/j.chembiol.2011.09.014 |
| [4] | Zhang Y H. Production of biofuels and biochemicals by in vitro synthetic biosystems:Opportunities and challenges[J]. Biotechnol Adv, 2015, 33(7): 1467–1483. DOI:10.1016/j.biotechadv.2014.10.009 |
| [5] | Facchini P J, Bohlmann J, Covello P S, et al. Synthetic biosystems for the production of high-value plant metabolites[J]. Trends Biotechnol, 2012, 30(3): 127–131. DOI:10.1016/j.tibtech.2011.10.001 |
| [6] | Ro D K, Paradise E M, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast[J]. Nature, 2006, 440(7086): 940–943. DOI:10.1038/nature04640 |
| [7] | Paddon C J, Keasling J D. Semi-synthetic artemisinin:a model for the use of synthetic biology in pharmaceutical development[J]. Nat Rev Microbiol, 2014, 12(5): 355–367. DOI:10.1038/nrmicro3240 |
| [8] | Galanie S, Thodey K, Trenchard I J, et al. Complete biosynthesis of opioids in yeast[J]. Science, 2015, 349(6252): 1095–1100. DOI:10.1126/science.aac9373 |
| [9] | Fossati E, Ekins A, Narcross L, et al. Reconstitution of a 10-gene pathway for synthesis of the plant alkaloid dihydrosanguinarine in Saccharomyces cerevisiae[J]. Nat Commun, 2014, 5: 3283–3290. |
| [10] | Brown S, Clastre M, Courdavault V, et al. De novo production of the plant-derived alkaloid strictosidine in yeast[J]. Proc Natl Acad Sci USA, 2015, 112(11): 3205–3210. DOI:10.1073/pnas.1423555112 |
| [11] | Zhao J, Bao X, Li C, et al. Improving monoterpene geraniol production through geranyl diphosphate synthesis regulation in Saccharomyces cerevisiae[J]. Appl Microbiol Biotechnol, 2016, 100(10): 4561–4571. DOI:10.1007/s00253-016-7375-1 |
| [12] | Hori K, Okano S, Sato F. Efficient microbial production of stylopine using a Pichia pastoris expression system[J]. Sci Rep, 2016, 6: 22201–22207. DOI:10.1038/srep22201 |
| [13] | Dang T T, Onoyovwi A, Farrow S C, et al. Biochemical genomics for gene discovery in benzylisoquinoline alkaloid biosynthesis in opium poppy and related species[J]. Methods Enzymol, 2012, 515: 231–266. DOI:10.1016/B978-0-12-394290-6.00011-2 |
| [14] | Zeng J, Liu Y, Liu W, et al. Integration of transcriptome, proteome and metabolism data reveals the alkaloids biosynthesis in Macleaya cordata and Macleaya microcarpa[J]. PLoS One, 2013, 8(1): e53409. DOI:10.1371/journal.pone.0053409 |
| [15] | Hawkins K M, Smolke C D. Production of benzylisoquinoline alkaloids in Saccharomyces cerevisiae[J]. Nat Chem Biol, 2008, 4(9): 564–573. DOI:10.1038/nchembio.105 |
| [16] | Hagel J M, Beaudoin G A, Fossati E, et al. Characterization of a flavoprotein oxidase from opium poppy catalyzing the final steps in sanguinarine and papaverine biosynthesis[J]. J Biol Chem, 2012, 287(51): 42972–42983. DOI:10.1074/jbc.M112.420414 |
| [17] | Galanie S, Smolke C D. Optimization of yeast-based production of medicinal protoberberine alkaloids[J]. Microb Cell Fact, 2015, 14(1): 144–150. DOI:10.1186/s12934-015-0332-3 |
| [18] | Trenchard I J, Siddiqui M S, Thodey K, et al. De novo production of the key branch point benzylisoquinoline alkaloid reticuline in yeast[J]. Metab Eng, 2015, 31: 74–83. DOI:10.1016/j.ymben.2015.06.010 |
| [19] | Deloache W C, Russ Z N, Narcross L, et al. An enzyme-coupled biosensor enables (S)-reticuline production in yeast from glucose[J]. Nat Chem Biol, 2015, 11(7): 465–471. DOI:10.1038/nchembio.1816 |
| [20] | Trenchard I J, Smolke C D. Engineering strategies for the fermentative production of plant alkaloids in yeast[J]. Metab Eng, 2015, 30: 96–104. DOI:10.1016/j.ymben.2015.05.001 |
| [21] | 刘秀斌.博落回属植物中生物碱代谢累积研究[D].长沙:湖南农业大学, 2012. |
 2017, Vol. 48
2017, Vol. 48


