2. 北京协和医学院 中国医学科学院医学生物学研究所, 云南 昆明 650118;
3. 昆明学院 化学科学与技术系, 云南 昆明 650224
2. Institute of Medical Biology, Chinese Academy of Medical Sciences and Peking Union Medical College, Kunming 650118, China;
3. Department of Chemical Science and Technology, Kunming University, Kunming 650224, China
蛭类(leeches)很早就被应用于医蛭疗法(hirudotherapy)和作为中药用于治疗心脏疾病、妇科疾病、泌尿疾病、外科手术、口腔疾病及眼科疾病[1]。世界上有600余种蛭类,中国有100余种。但是被FDA正式批准的治疗水蛭仅欧洲医蛭Hirudo medicinalis一种[2];《中国药典》2015年版中规定水蛭药材源动物仅包含水蛭科水蛭、蚂蟥、柳叶蚂蟥。李军德等[3]对蛭类药材来源考证认为《中国药典》中规定的此3种水蛭正式中文名为日本医蛭Hirudo nipponia,归属于医蛭科Hirudinidae医蛭属HirudoLinnaeus;蚂蟥正式中文名为宽体金线蛭Whitmania pigra Whitman、柳叶蚂蟥正式中文名为尖细金线蛭Whitmania acranulata Whitman,二者归属于黄蛭科(Haemopidae)金线蛭属Whitmania Whitman。并建议扩大水蛭药材来源包含:菲牛蛭Poecilobdell amanillensis Lesson、或山蛭科动物天目山蛭Haemadipsa tianmushana Song的。随着心血管等疾病对蛭类药材资源需求的日益增长,扩大蛭类用药资源仍然迫切。分布在云南等中国西南的医蛭科牛蛭属棒纹牛蛭Poecilobdella javanica Wahlberg[4],以及蝙蝠血为食物的陆生山蛭类新种洞穴山蛭Heamadipsa cavatuses sp. nov.[5],是抗凝血活性极强的吸血蛭,据中药中蛭类使用原则推测[6]和《中国药典》对药用蛭质量鉴定的抗凝血酶活性标准,棒纹牛蛭和洞穴山蛭是具有潜在药用价值的动物资源,其吸血过程中唾液包含大量抗凝血功能物质,可发展为治疗心血管疾病及脑部血栓等疾病药物。
由于水蛭具有活血化瘀等功效,在中药中主要用于治疗心血管疾病,《中国药典》2010年版规定利用抗凝血酶活性来定义水蛭的效价。但是由于水蛭药用成分中发挥抗凝血功能的物质远比抗凝血酶复杂得多[7],吸血蛭类中吸血相关的大量功能物质都可能参与蛭类抗凝血过程。因而以凝血酶为靶点不能有效控制水蛭临床应用的有效性及安全性,更不能作为药用蛭资源鉴定和扩大的标准。
本实验利用RNA-Seq技术对宽体金线蛭、棒纹牛蛭、洞穴山蛭、日本医蛭进行测序并从头组装分别得到各个物种的转录组数据,通过MISA软件分析各转录组数据获得的各物种的EST-SSR信息。并比较各种物EST-SSR在转录组中的分布特点及规律;另一方面,对4种蛭类中相同的抗凝血分子基础进行重叠分析;综合2部分数据挖掘4种蛭中抗凝血功能特有的通用功能SSR标记,为蛭类药用资源鉴定和遗传监测、质量控制的分子标记开发提供参考数据。
SSR序列一般是指由1、2、3、4、5和6核苷酸重复单元串联排列的简单重复序列,其广泛分布于各类生物基因组内[8-9]。对于不同的生物而言,其基因组内SSR的密度、类型及其分布区域都存在较大的差异[10]。大量研究表明,基因不同区域存在的SSR序列对物种的进化、基因表达的调节以及基因翻译产生的蛋白功能都有一定的影响[11-13],因此SSR可以作为特定功能的分子标记。
1 材料与方法 1.1 材料所用蛭类样本包括棒纹牛蛭、日本医蛭、洞穴山蛭和金线蛭均采自云南省西南地区德宏州。其中棒纹牛蛭在处理时按其习性分别设置饥饿状态、吸血后30 min、10 d、20 d、30 d共8个样本;洞穴山蛭包括饥饿状态、吸血后30 min、10 d共3个样本;日本医蛭包含饥饿和吸血30 min 2个样本(表 1)。所有样本采回后切开,并用去离子水反复清洗体表及体腔,然后用75%无水乙醇再次清洗后立即提取RNA用于后续分析。
|
|
表 1 用于转录组分析的各物种样品数量 Table 1 Number of samples prepared for transcriptomic analysis |
1.2 RNA提取及转录组分析准备
在进行Illumina测序前,先提取总RNA,用Agilent 2100 Bioanalyzer RNA Nanochip进行质量监控,确保总RNA质量浓度≥400 ng/μL,A260/A280在1.8~2.2,RNA 28 S:18 S≥1,且RNA完整度指数RNA Integrity Number(RIN)≥7.0。接着用NEBNext Poly(A)mRNA Magnetic Isolation Module(NEB,E7490)富集mRNA,并NEBNext mRNA Library Prep Master Mix Set for Illumia(NEB,E6110)和NEBNext Multiplex Oligos for Illumina(NEB,E7500)试剂盒构建测序文库,用1%琼脂糖凝胶电泳检测合格后,用Library Quantification Kit-Illumina GA Universal(Kapa,KK4824)试剂盒进行qPCR定量检测,检测合格的文库用Illumina cbot进行聚类,之后用Illumina HiSeqTM 2500(Biomarker biotechnological Co. Beijing)进行测序。
1.3 Illumine reads加工及从头组装经测序得到的原始pair-end read通过质量分数、碱基分布、数据评估和统计等校验后,进一步滤过掉接头、未识别的核酸和带模糊序列“N”的低质量reads,最终获得的高质量reads用Trinity[7]进行各个物种分别从头组装(3个物种均无参考基因组信息)。所得到的unigene进行下一步的注释和分析。
1.4 基因注释和SSR分析对所得unigene用BLAST软件(E值 < 1×10-5)以下公共蛋白数据库中进行同源查找,按相似基因进行功能注释。包括:NCBI非冗余数据库(http://www.ncbi.nlm.nih.gov/)、Swiss-Prot蛋白数据库(http://www.expasy.ch/sprot/)、COG(Clusters of Orthologous Groups)数据库(http://www.ncbi.nlm.nih.gov/cog/)和KEGG数据库(http:www.genome.jp/keg/)。GO功能分配和分类分别用Gene Ontology(GO)(http://www.geneontology.org/)和WEGO(http//wego.genomics.org.cn/cgi-bin/wego/index.pl)。利用MISA软件筛选到的1 kb以上的unigenes做SSR分析。通过SSR简单重复序列的查找得到单核苷酸重复次数在10次或10次以上,二核苷酸重复次数在6次或6次以上,2~6核苷酸重复次数在5次或5次以上的完全重复SSR以及中间被少数碱基(间隔≤100)打断的不完全重复的SSR。所有得到的SSR序列通过EXCEL分析,得到本实验结果。参考已有的研究分析[9],本实验将所有可循环序列及与其互补序列归为一类,将单核苷酸重复序列归为A、C 2类;二核苷酸重复序列归为AC、AG、AT和CG 4类:3核苷酸重复序列归为10类;4核苷酸重复序列归为16类;5核苷酸重复序列归为50类;6核苷酸重复序列可归为160类。如3核苷酸重复序列中,AAG基序代表了所有AAG、AGA、GAA、CTT、TCT和TTC的SSR序列。随后利用blast+算法将拼接得到的非重复序列基因与CDD库中的COG库进行比对,对饥饿洞穴山蛭含SSR的不同非重复序列基因进行COG功能分类及分析。
2 结果与分析 2.1 4种蛭类转录组测序及组装对宽体金线蛭(1个样本)、棒纹牛蛭(吸血前及吸血后不同时间段的8个样本)、洞穴山蛭(吸血前及吸血后不同状态下的3个样本)及日本医蛭(吸血前及吸血后2个样本)各样本分别构建cDNA文库后,用Illumina HiSeqTM 2500进行双末端RNA测序。原始双末端reads经数据评估后,滤过掉低质量的数据和rRNA,并用Trinity软件分别对不同物种的各个样品数据分别进行混合组装,获得对应的转录本序列(Transcripts),在聚类单元中选取最主要的转录本作为unigenes序列,用cd-hit聚类去冗余,采用TGICL聚类组装策略最终得到unigene文库。金线蛭cDNA总共获得5.29 Gb数据,去除低质量数据后使用4.23 Gb数据;棒纹牛蛭8个样本测序总共数据量为33.6 Gb,使用28.3 Gb;洞穴山蛭总共获得14.95 Gb数据,除去低质量数据后使用12.55 Gb;日本医蛭则获得和使用14.58 Gb数据。除日本医蛭一组的3个样本Q30(测序错误概率为0.01)值为88%外,其余样本均大于93%。最终棒纹牛蛭组装获得57 410个unigene,平均长度1 413 bp;洞穴山蛭组装获得37 465个unigene,平均长度1 362 bp;日本医蛭组装获得39 812个unigene,平均长度1 012 bp;宽体金线蛭组装获得22 875个unigene,平均长度1 214 bp(表 2)。从4种蛭类片段分布长度来看,日本医蛭与其他3类蛭有明显差异,其200~300 bp短片段分布明显高于长片段分布,而其他3种蛭类正好相反。
|
|
表 2 4种蛭中RNA测序后unigene的数量及分布 Table 2 Number and distribution of assembled unigenes in four leech species |
2.2 4种蛭SSR分布特点及分析
利用MISA软件对筛选到的1 kb以上的unigenes进SSR分析。结果从棒纹牛蛭unigene文库选出14 262个SSR,SSR的频率为35.6%,平均每隔4 723 bp;洞穴山蛭文库中共筛到5 150个SSR,SSR的频率为24.9%,平均间隔8 144个unigene;相同的参数在日本医蛭和宽体金线蛭中分别是SSR总数4 451个和2 698个,SSR频率分别为25.2%和22.3%,平均间隔长度分别为6 026 bp和8 059 bp。从SSR分布密度特点来看,棒纹牛蛭unigene中SSR出现的频率较高,平均间隔最短;其后依次是日本医蛭、宽体金线蛭和洞穴山蛭(表 3)。日本医蛭unigene文库中由于低于1 kb片段较多(表 2),因此该结果可能低估了日本医蛭中SSR分布频率;另外,棒纹牛蛭由于测序所用的8个样本能涵盖更广泛的mRNA,因此获得更多的表达基因,较大的数据本底可能是导致检出高频率的SSR原因之一。
|
|
表 3 4种蛭中SSR在unigene中密度分布比较 Table 3 Comparision of SSR density distribution in unigene of four leeches |
进一步对4种蛭中SSR基序组成类型分布分析发现,4种蛭中单碱基重复,二碱基重复和三碱基重复占整个SSR类型的绝大多数,其中三碱基重复占绝大多数,体现了蛭类SSR碱基重复的共同规律。对4种蛭中SSR类型分布比率作图发现,在4种蛭中同时占比最高的单碱基因类型(11%~27%)和三碱基类型(47%~68%)中,洞穴山蛭明显不同于其余3种,其单碱基比率远低于其余3种,而三碱基比率远高于其余三者。进一步证实了洞穴山蛭的SSR分布规律与其余3种蛭明显不同(图 1)。
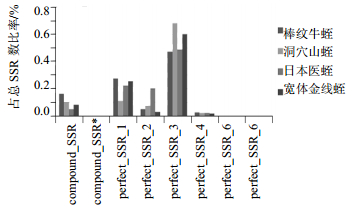
|
perfect_SSR_1、perfect_SSR_2、perfect_SSR_3、perfect_SSR_4、perfect_SSR_5、perfect_SSR_6分别为1~6碱基重复的SSR类型;compound_SSR为混合类型的SSR;compound_SSR*为混合导致的不确定的SSR perfect_SSR_1, perfect_SSR_2, perfect_SSR_3, perfect_SSR_4, perfect_SSR_5, perfect_SSR_6 represent mononucleotide to hexanucleotide repeat respectively; compound_SSR* represent uncertain SSR because of mixture 图 1 不同碱基类型的SSR类型所占比率比较 Fig.1 Comparision of ratios of SSR type for four leeches |
进一步对三碱基类型的SSR的不同基序深入分析发现,AAC/GTT、AAT/ATT、ATC/GAT 3个基序所占比例在4个蛭中最大,针对这3个基序类型作图发现,洞穴山蛭所占比率明显不同于其余三者,尤其AAC/GTT明显低于其余三者,在ATC/GAT基序明显高于其余三者(图 2)。
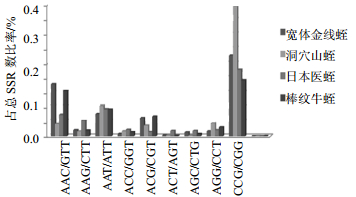
|
图 2 三碱基重复的SSR中各类基序在4种蛭中的比率分布 Fig.2 Distribution of ratios of all trinucleotide repeat motif in four leeches |
从4个蛭种的SSR分布密度及组成特点可以发现,医蛭科牛蛭属的棒纹牛蛭与日本医蛭的SSR分布密度最为相近,但在SSR组成上黄蛭科金线蛭属的宽体金线蛭和医蛭属的棒纹牛蛭最为相近;无论从密度和组成来看,山蛭科的洞穴山蛭与其余三者相对差异明显。由于日本医蛭可能由于测序时cDNA片段质量因素,因此在SSR组成与棒纹牛蛭差异较大。因此综合SSR分布密度和组成来看,棒纹牛蛭和日本医蛭最近,其次是宽体金线蛭,最远的是洞穴山蛭。SSR分布规律与蛭类分类关系完全一致。
2.3 抗凝血相关分子标记的挖掘药用蛭类主要用于治疗心血管疾病及脑部血栓等疾病,从分子机制上,蛭类药理作用主要与抗凝血相关分子相关,吸血蛭类都具有丰富的抗凝血相关分子,而非吸血蛭宽体金线蛭同样具有极强的抗凝血活性。因此可从4种蛭中挖掘抗凝血相关的分子标记。为了确定4种蛭中抗凝血的分子是否相似,将棒纹牛蛭、洞穴山蛭、日本医蛭吸血前后的unigene进行比对,获得吸血前后RPKM值(Reads Per Kilobase per Million mapped reads,反映对应unigene的表达丰度)显著差异的unigene分别为:棒纹牛蛭1 187个,洞穴山蛭897个,日本医蛭716个。经swissprot蛋白数据库注释后,分别获得无冗余的功能597、486、347个。功能比对发现,在这3类吸血差异基因中,棒纹牛蛭与洞穴山蛭有169个相同、洞穴山蛭与日本医蛭有124个相同、日本医蛭与棒纹牛蛭有143个相同(表 4)。虽然金线蛭为非吸血蛭,但其转录组中注释得到的1 107个功能中,仍有178、145、111个分别与棒纹牛蛭、洞穴山蛭和日本医蛭的抗凝血相关分子功能相同(图 3)。该结果表明4种蛭中有共同的抗凝血相关分子基础。其中宽体金线蛭和日本医蛭抗凝血功能分子最为接近,随后是洞穴山蛭和棒纹牛蛭。有趣的是
|
|
表 4 4种蛭之间抗凝血功能分子的重叠数据 Table 4 Number of overlap unigenes in four kinds of leeches |

|
图 3 4种蛭类中抗凝血相关功能分子重叠分析 Fig.3 Overlap analysis for number of anticoagulating unigenes in four kinds of leeches |
抗凝血相关功能分子并不因为洞穴山蛭亲缘关系较远而出现较大差异,相对于医蛭科中棒纹牛蛭,洞穴山蛭在吸血相关的抗凝功能分子上与作为药用蛭的宽体金线蛭和日本医蛭更相似。这与实际应用情况相似,即宽体金线蛭和日本医蛭都作为中药使用,而山蛭中很早就发现山蛭素[14]而被广泛应用,相对而言牛蛭的应用较少。由此推测从4种蛭类相同抗凝相关差异分子能较好地反映水蛭应用于心血管等疾病治疗的情况,因此从中筛选SSR标记,可以较好地反映蛭类的药用价值。
基于以上推测,进一步进行重叠分析得到4种蛭中具有相同的抗凝血相关分子功能48个,每个功能均来自unigene在Swissprot数据库的序列相似性比对结果,因而每个注释功能在4种蛭中具有数量不等的包含SSR的unigene片段。每个功能在每个物种中对应的含SSR的unigene片段越多,意味着等位基因越丰富,即SSR滑动突变导致的基因越丰富,因此SSR分子标记多态性越高,按照该假设筛选到22个功能分子,包括Uncharacterized histidine-rich protein DDB_G0274557(Precursor),Putative leucine-rich repeat-containing protein DDB_G0290503,ADAM 17-like protease(Precursor),Ankyrin,Epidermal growth factor receptor substrate,Filamin-A,Serine/threonine-protein kinase par-1 Low-density lipoprotein receptor-related protein(Precursor),A disintegrin and metalloproteinase with thrombospondin motifs gon-1(Precursor)等抗凝血相关Swissprot注释功能,其中等位基因片段最丰富的2个,分别为histidine-rich protein和leucine-rich repeat-containing protein,均为SSR三碱基基序重复导致的组氨酸重复序列和亮氨酸重复序列。以SSR出现频率最低的宽体金线蛭为本底,获得22个功能对应的164个主要以包含ACC/GTT、AAT/ATT、ATC/GAT基序的SSR及其对应的137个unigene,作为开发药用蛭类质量鉴定和监测等分子标记的EST-SSR片段。
3 讨论 3.1 三碱基重复基序在蛭类中EST-SSR组成中占绝大多数通过对蛭类3科4属4个物种14个样本的转录组测序分析,发现蛭类EST-SSR分布特点极为相似,三碱基基序出现频率接近或超过50%,其次是单碱基10%~30%和少量的二碱基。SSR碱基基序的分布在不同物种中有不同的特点。EST-SSR分析发现鸡中90%以上为单碱基和二碱基的SSR序列[15];山羊中EST-SSR分析结果发现二碱基、三碱基和六碱基出现的比例最高,其中二碱基比例最高[16],另武昌鱼Megalobrama amblycephala Yih和黄鱼Larimichthys polyactisBleeker转录组分析表明二碱基重复达绝大多数,其次是占较小比例的三碱基[17];可见三碱基基序在SSR类型中占较大比例是蛭类SSR分布的重要特点。
3.2 EST-SSR在蛭类中密度和分布反映物种间亲缘关系从4种蛭类SSR分布特点可以发现洞穴山蛭在EST-SSR密度和组成分布特点上与医蛭科其他3种区别较大,而金线蛭又可与日本医蛭和棒纹牛蛭区别。SSR的密度及组成分布特点甚至可以区分科间、属间关系。通过基因组中SSR分布分析发现,SSR基序分布特点各物种有所不同,如按基序所占比例的先后顺序排序的话,人基因组中顺序是一、四、二,即中SSR分布主要是单碱基重复、其次是四碱基和二碱基重复;果蝇Drosophila melanogasterMeigen基因组中SSR基序比例分别是二、三、一、四碱基重复;秀丽隐杆线虫Caenorhabditis elegansBergerac碱基基序比例顺序是一、二、三、四;拟南芥Arabidopsis thaliana(L.) Heynh.基因组中SSR基序比例也是一、二、三、四,但一、二、三占绝大部分;酿酒酵母Saccharomyces cerevisiaeMeyen exE.C. Hansen则为一、三、二、四[18]。由此可见不同的物种可以按SSR基序的分布特特点加以区分。SSR作为DNA指纹图谱和多样性研究的理想的遗传标记,主要因为微卫星位点存在的片段长度多态性[19-20]。同时,大量研究表明SSR密度、组成及分布特点在不同物种间都有所区别[21-23]。基于抗凝血共同分子基础筛选的SSR标记可以作为医蛭药用资源鉴定的标记。
3.3 抗凝血关联的功能SSR候选unigene在基因组中,SSR可以出现在编码区,也可以分布在非编码区。SSR出现在编码区域时,就会导致编码蛋白的随机重复[24],许多研究认为编码区中包含SSR的基因群,可能是进化速度较快的蛋白[25-26],这些SSR具有与染色体稳定、RNA折叠、氨基酸重复或人类疾病、重组热点、转座子相关的生物学功能[18]。功能SSR通常出现在应对选择压力的基因群中,如包含SSR的EST序列在肺癌组织中具有不同的氨基酸组成和蛋白表达模式[28]。以吸血为主的蛭类尤其是医蛭科和山蛭科种属,抗凝血需求是其进化中的主要进化压力。
4种蛭中包含SSR的unigene有很大部分与抗凝血相关基因重叠。以等位基因多态性进行排序获得的前22个功能分子几乎与抗凝血机制相关联。其中出现的多态性最高的2个分别为富含组氨酸重复和亮氨酸重复的蛋白,可能是直接由三碱基的SSR重复序列编码的结果。这些迹象显示抗凝血机制关联的包含SSR的unigene中,可能是筛选抗凝血功能SSR的unigene类群。
| [1] | Baskova I P, Kostrjukova E S, Vlasova M A, et al. Proteins and peptides of the salivary gland secretion of medicinal leeches Hirudo verbana, H.medicinalis, and H.orientalis[J]. Biochemistry (Mosc), 2008, 73(3): 315–320. DOI:10.1134/S0006297908030127 |
| [2] | 鞠利雅. 法国蚂蟥获准FDA医用许可证书[J]. 中国中药杂志, 2004, 29(10):1022–1023. |
| [3] | 李军德, 黄璐琦, 唐仕欢, 等. 《中国药典》2010年版一部部分动物药材来源探讨[J]. 中国中药杂志, 2010, 35(16):2051–2055. |
| [4] | 王德斌, 仝向荣, 王斌, 等. 云南省蛭类动物及其区系的重要性[J]. 昆明学院学报, 2009, 31(6):49–51. |
| [5] | 杨潼, 莫潇, 王德斌, 等. 洞穴吸血陆蛭一新种在中国云南省西陲的发现[J]. 动物分类学报, 2009, 34(1):125–129. |
| [6] | 吕莉, 李欣燕, 韩国柱, 等. 重组水蛭素体内抗凝活性的实验研究[J]. 中草药, 2002, 33(6):520–522. |
| [7] | Corral R M, Macedo R S, Pereira P J, et al. Leech-derived thrombin inhibitors:from structures to mechanisms to clinical applications[J]. J Med Chem, 2010, 53(10): 3847–3861. DOI:10.1021/jm901743x |
| [8] | Emmanuel B, Neil J. The rise, fall and renaissance of microsatellites in eukaryotic genomes[J]. BioEssays, 2006, 28(10): 1040–1050. DOI:10.1002/(ISSN)1521-1878 |
| [9] | Gur-Arie R, Cohen C J, Eitan Y, et al. Simple sequence repeats in Escherichia coli:abundance, distribution, composition, and polymorphism[J]. Genome Res, 2000, 10(1): 62–71. |
| [10] | Li Y C, Korol A B, Fahima T, et al. Microsatellites within genes:structure, function, and evolution[J]. Mol Biol Evol, 2004, 21(6): 991–1007. DOI:10.1093/molbev/msh073 |
| [11] | Verstrepen K J, An J, Lewitter F, et al. Intragenic tandem repeats generate functional variability[J]. Nature Genetics, 2005, 37: 986–990. DOI:10.1038/ng1618 |
| [12] | Rocha E P, Blanchard A. Genomic repeats, genome plasticity and the dynamics of Mycoplasma evolution[J]. Nucleic Acids Res, 2002, 30(9): 2031–2042. DOI:10.1093/nar/30.9.2031 |
| [13] | Li Y, Korol A B, Fahima T, et al. Microsatellites:genomic distribution, putative functions and mutational mechanisms:a review[J]. Mol Ecol, 2002, 11(12): 2453–2465. DOI:10.1046/j.1365-294X.2002.01643.x |
| [14] | Strube K H, Kröger B, Bialojan S, et al. Isolation, sequence analysis, and cloning of haemadin.An anticoagulant peptide from the Indian leech[J]. J Biol Chem, 1993, 268(12): 8590–8595. |
| [15] | Bakhtiarizadeh M R, Arefnejad B, Ebrahimie E, et al. Application of functional genomic information to develop efficient EST-SSRs for the chicken (Gallus gallus)[J]. Genet Mol Res, 2012, 11(2): 1558–1574. DOI:10.4238/2012.May.21.12 |
| [16] | 赵传志, 李爱芹, 李长生, 等. 山羊EST资源的SSR分析[J]. 山东农业科学, 2009(6):6–9. |
| [17] | Gao Z, Luo W, Liu H, et al. Transcriptome analysis and SSR/SNP markers information of the blunt snout bream (Megalobrama amblycephala)[J]. PLoS One, 2012, 7(8): e42637. DOI:10.1371/journal.pone.0042637 |
| [18] | Mukund V K, Prabhakar K R, Vidya S G. Differential distribution of simple sequence repeats in Eukaryotic genome sequences[J]. Mol Biol Evol, 2001, 18(7): 1161–1167. DOI:10.1093/oxfordjournals.molbev.a003903 |
| [19] | Beckmann J S, SOLLER M. Toward a unified approach to genetic mapping of eukaryotes based on sequence tagged microsatellite sites[J]. Biotechnology, 1990, 8: 930–932. DOI:10.1038/nbt1090-930 |
| [20] | Morgante M, Olivieri A M. PCR-amplified microsatellites as markers in plant genetics[J]. Plant J, 1993, 3: 175–182. DOI:10.1111/j.1365-313X.1993.tb00020.x |
| [21] | Galindo C L, Mciver L J, Mccormick J F, et al. Global microsatellite content distinguishes humans, primates, animals, and plants[J]. Mol Biol Evol, 2009, 26: 2809–2819. DOI:10.1093/molbev/msp192 |
| [22] | Mayer C, Leese F, Tollrian R. Genome-wide analysis of tandem repeats in Daphnia pulex-a comparative approach[J]. BMC Genomics, 2010, 11(19): 277–285. |
| [23] | Pannebakker B, Niehuis O, Hedley A, et al. The distribution of microsatellites in the Nasonia parasitoid wasp genome[J]. Insect Mol Biol, 2010, 19(Suppl 1): 91–98. |
| [24] | Katti M V, Sami-Subbu R, Ranjekar P K. Amino acid repeat patterns in protein sequences:their diversity and structural-functional implications[J]. Protein Sci, 2000, 9(5): 1203–1209. |
| [25] | Green H, Wang N. Codon reiterations and the evolution of proteins[J]. Proc Natl Acad Sci USA, 1994, 91: 4298–4302. DOI:10.1073/pnas.91.10.4298 |
| [26] | Huntley M, Golding G B. Evolution of simple sequence in proteins[J]. J Mol Evol, 2000, 51: 131–140. DOI:10.1007/s002390010073 |
| [27] | Bakhtiarizadeh M R, Ebrahimi M, Ebrahimie E. Discovery of EST-SSRs in lung cancer:Tagged ESTs with SSRs lead to differential amino acid and protein expression patterns in cancerous tissues[J]. PLoS One, 2011, 6(11): e27118. DOI:10.1371/journal.pone.0027118 |
 2017, Vol. 48
2017, Vol. 48


