物种多样性是生物多样性的重要组成部分,随着全球生物多样性程度的丧失,物种多样性保护已经成为公众关注的热点问题,也是当前生命科学领域研究的重要方向。对物种数目统计及其相互关系的研究是物种多样性保护的前提与基础,传统依赖于形态学标准的物种鉴定往往需要专门知识,只有少数分类学专家才能有效掌握。生物多样性的保护急需新的、标准化的、高效的物种鉴定手段。2003年Hebert等[1]提出将DNA条形码应用于分类学研究,希望能够利用一个或多个DNA片段实现物种的快速、准确鉴定,用来探讨区系组成、评估生物多样性水平。2009年,CBOL Plant Working Group提出将叶绿体rbcL+matK片段组合作为陆地植物核心条形码,叶绿体基因psbA-trnH和核基因片段ITS作为补偿[2]。2011年中国植物条形码研究团队(China Plant BOL Group)建议将ITS纳入核心条形码当中[3]。由于叶绿体rbcL和matK片段进化速率较慢,适合于科属及以上水平类群的分析,而对于经历快速辐射或最近分化的类群,这2个条形码标记就往往缺乏有效性[4]。ITS现已广泛应用于系统发育分析中,但是鉴于ITS是多拷贝基因,在许多类群中存在致同进化不完全现象以及PCR扩增的偏好性,容易导致系统发育关系的不确定性,为物种鉴定带来很大的难度[5]。
与多拷贝核基因相比,单拷贝核基因优势在于可直接区分直系同源(orthologous)和旁系同源(paralogous),易建立物种间直系同源的关系,其兼具核基因的双亲遗传以及序列保守性的特点,进化速率比质体基因组序列更快而且能够选取的序列片段更加丰富。单拷贝核基因条形码开发所面临的最大困难是大多数植物缺乏基因组信息,DNA条形码的通用性较差。随着新一代测序(next generation sequencing)技术的飞速发展,大规模转录组测序变得简便,核基因数据呈几何式增长,使开发不同类群通用的单拷贝核基因DNA条形码成为可能。Duarte等[6]发现4个属植物(拟南芥属Arabidopsis Heynh.、杨属Populus Linn.、稻属Oryza Linn.和葡萄属VitisLinn,APOV)之间共享的959个单拷贝核基因,Zhang等[7]进一步开发出1 083个高度保守的低拷贝基因。这些单拷贝基因已经应用于多种植物类群系统关系研究中,如杨柳科[8]、山龙眼科[9]等,并为单拷贝核基因DNA条形码的筛选提供了有效工具。
地黄属Rehmannia Libosch. ex Fischer & Mey.是隶属于玄参科(Scrophulariaceae),亦有研究认为应该独立成地黄科(Rehmanniaceae)[10]。《中国植物志》记载地黄属有6个种,包括中国著名的传统中药地黄R. glutinosa Libosch. ex Fisch. & C. A. Mey.、天目地黄R. chingii Li、裂叶地黄R. piasezkii Maxim.、高地黄R. elata N. E. Br. ex Prain、湖北地黄R. henryi N. E. Br.和茄叶地黄R. solanifolia Tsoong et Chin,其中地黄和茄叶地黄是四倍体[11]。地黄属植物的研究主要集中在传统药用植物地黄的药理作用、物质基础、繁育体系、遗传变异等方面[11],而缺乏属内其他物种的研究,加上该属植物分布范围广,外观形态差异较大,经常造成分类学上的争议。比如,最近的形态、化学成分以及分子证据显示高地黄和裂叶地黄实为同一物种[11-12],茄叶地黄与地黄在遗传上高度一致,也应该予以归并[11]。当前已有研究探讨DNA条形码在地黄属的适用性。程芳婷等[13]利用trnL-F、trnMV、trnS-G 3个叶绿体片段与核基因ITS对地黄属物种进行分析后,推荐ITS+trnS-G作为地黄属的标准条形码。该研究没有说明psbA-trnH、matK、rbcL等国际标准候选条形码在地黄属应用的可行性。最近,夏至等[4]补充了该标准条形码的数据,认为psbA-trnH也可以作为地黄属的核心条形码。遗憾的是,除地黄外这一研究没有开展其他物种种内遗传变异分析,无法确定这些标记的种内最大变异是否显著小于种间最小变异范围,其物种鉴定的可靠性受到质疑。此外,采用单一个体代表物种所进行系统发育分析,往往会对种间亲缘关系的确定构成负面影响[14]。本研究目的在于通过大量的群体数据,对比多个叶绿体与核基因DNA标记的差异性,筛选出适合地黄属的DNA条形码,确立地黄属各物种的亲缘关系,并探讨单拷贝核基因作为物种鉴定DNA条形码的可行性。
1 材料与方法 1.1 材料由于高地黄与裂叶地黄的遗传组成相一致,本研究将二者作为一个物种处理[11]。实验选取了地黄属5个物种13个居群共65份个体作为研究对象[13](表 1)。所用实验材料均为野外采集的叶片,经硅胶快速干燥,常温保存备用,标本保存于西北大学生命科学学院。
|
|
表 1 材料来源 Table 1 Information of materials |
利用CTAB法从叶片中提取基因组DNA,叶绿体基因片段引物参照文献方法[13]设计,单拷贝核基因由地黄转录组开发获得[11],PCR扩增程序按照相关文献方法[11, 13]操作,PCR产物由上海生工生物工程技术服务有限公司完成测序工作。根据先前的研究成果,本研究总共对12个单拷贝核基因(R10、R15、R56、R84、R118、R124、R146、R163、R254、R255、R256、R257)、1个多拷贝核基因(ITS)和6个叶绿体基因(matK、psbA-trnH、rbcL、trnL-F、trnM-V、trnS-G)进行对比分析。
1.2 数据处理原始序列采用BioEdit ver. 7.0.9.1软件[15]编辑并进行人工校对。利用MEGA ver. 6.0软件[16]统计每个基因片段的长度、插入/缺失(indels)、变异位点数、简约信息位点数等序列特征;分析各DNA片段种内、种间的Kimura-2-parameter(K2P)遗传距离,同时以崖白菜Triaenophora rupestris (Hemsl.) Solereder为外类群构建单一片段以及片段组合的邻接(neighbor-joining,NJ)树。采用建树法和距离法这2种不同的方法评估DNA条形码的物种鉴定效果。建树法认为只有同一个物种的不同个体在构建的NJ系统树上形成单系分支才认为物种鉴定成功;距离法采用PWG距离(PWG-distance)法,认为种间最小的遗传距离大于种内最大的遗传距离,则物种鉴定成功[3];此外,还应用TaxonDNAver.1.7.8软件[17-18],通过最佳匹配(Best match)计算每个个体间的相似性,判断每个个体所属物种的准确率。只有当最佳匹配值大于0.4时,该标记才会用于条形码组合分析。由于本研究侧重于分析叶绿体基因与单拷贝核基因在地黄属物种鉴定成功率的差异性,因此仅以ITS为参照,构建了叶绿体基因与单拷贝核基因的独立数据矩阵,并没有将二者进行联合数据集分析。考虑到前期研究均表明茄叶地黄往往包含在地黄系统分支内,把地黄-茄叶地黄分支组成单系群确定为地黄鉴定成功的标准。
2 结果 2.1 序列特征本研究所采用的19个基因片段在地黄属5个种13个居群65个个体均能够进行成功扩增与测序。除R84外,其余11个单拷贝核基因片段序列均仅出现位点的碱基替代,没有插入/缺失现象。变异位点比率与简约信息位点比率变化范围均在0.77%~4.33%,平均值分别为2.44%和2.34%,R146变异最低,而R84最高(表 2)。裂叶地黄(除R15、R56基因外)与天目地黄(除R56基因外)在绝大多数核基因中都表现出较高水平的种内居群间变异,湖北地黄只在R118和R257中存在变异,而地黄与茄叶地黄序列完全相同,在所有基因中均呈现单态现象。多拷贝核基因ITS则体现出种内较高的多态性,每个物种种内均存在变异,变异位点、简约信息位点比率分别为3.50%和3.07%。在6个叶绿体基因片段中,除matK和rbcL,其他基因均存在碱基的插入/缺失,其中psbA-trnH与trnS-G插入/缺失十分频繁,各有10个和8个,最长缺失分别长达76 bp和30 bp(表 2)。叶绿体基因的变异位点比率、简约信息位点比率的变化幅度分别为0.15%~4.02%和0.15%~3.62%,平均值为1.67%和1.39%,稍低于单拷贝核基因。rbcL变异率最低,仅在天目地黄种内有1个变异位点,其他物种均共享一个单倍型;psbA-trnH基因有20个变异位点,序列变异比率最高;其次是trnS-G,而trnL-F与trnM-V在地黄属内变异水平相当(表 3)。在所有地黄属物种中,多数叶绿体基因片段表现出居群个体间的多态性。但也有例外,比如rbcL个体间同源性近乎100%,湖北地黄也表现出种内低的变异水平,仅在psbA-trnH基因上存在个体间遗传变异。
|
|
表 2 核基因片段遗传特征以及物种鉴定成功率 Table 2 Characteristics of each nuclear or chloroplast fragment and their successful identification rates for species inRehmannia Libosch. ex Fisch. et Mey. |
|
|
表 3 叶绿体基因片段遗传特征以及物种鉴定成功率 Table 3 Characteristics of chloroplast fragment and their successful identification rates for species inRehmannia Libosch. ex Fisch. et Mey. |
2.2 地黄属DNA条形码鉴定
利用距离法(PWG距离法和Best match法)和建树法比较不同DNA条形码对地黄属物种的鉴定成功率。ITS作为推荐的核心条形码在3种方法中都具有最高的分辨率。PWG距离法分析表明,多数单拷贝核基因片段存在种内最大遗传距离大于种间最小遗传距离,单拷贝核基因使用有一定的限制性。只有R255、R256和R257能够与ITS媲美,符合种内最大遗传距离小于种间最小遗传距离的DNA条形码的标准(表 2)。而依据Best match法,物种分辨率最高的R255、R257在地黄属也只有57%的鉴定成功率。建树法结果则表明单拷贝核基因不能单独地100%完成物种鉴定,分辨率最高的R257也只能成功分辨75%的物种。在本研究涉及的6个叶绿体基因片段中,PWG距离法、Best match法和建树法都显示rbcL没有分辨率,matK的物种分辨率也较低。虽然在不同条形码分析方法中得到的物种分辨率略有差异,但是psbA-trnH、trnL-F、trnM-V与trnS-G都显示较高水平的物种鉴定成功率(50%~100%),见表 3和图 1-a。
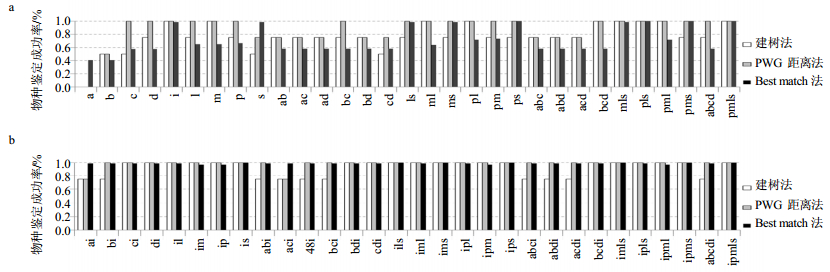
|
a=R84 b=R254 c=R255 d=R257 l=trnL-F m=trnM-V s=trnS-G p=psbA-trnH i=ITS 图 1 不包含ITS的条形码组合(a)和包括ITS的条形码组合(b)的物种鉴定成功率 Fig.1 Successful identification rates of barcode combinations without ITS fragment (a) and barcode combinations containing ITS (b) |
通过Best match法分辨率大于0.4的标准进一步筛选标记,单拷贝核基因与叶绿体基因各自获得4个片段用于构建基因片段组合数据集,分别是核基因的R84、R254、R255、R257和叶绿体基因的psbA-trnH、trnL-F、trnM-V与trnS-G(表 1)。DNA条形码分析表明,随着基因片段组合数目的增加,叶绿体基因物种分辨率快速增加,按照PWG距离法2个叶绿体片段的组合就能达到100%的分辨率,而按照建树法则需要3个叶绿体片段组合。与之相对,2、3与4个单拷贝核基因组合数据得到的物种分辨率没有显著区别,只有R255+R257+R84组合能够依照PWG距离法和建树法100%将地黄属各物种区分开(图 1-a)。当把ITS作为必需的DNA条形码组合时,几乎所有的基因片段组合方式都有近乎100%的分辨率(图 1-b),这进一步证实了ITS作为核心条形码的必要性[19]。
2.3 地黄属的系统发育关系以崖白菜为外类群,利用12个单拷贝核基因、ITS和6个叶绿体基因片段构建了地黄属5个物种的NJ树。结果表明,裂叶地黄、天目地黄、湖北地黄均为单系群,地黄与茄叶地黄为并系群,每个分支都有98%以上的支持率。湖北地黄与天目地黄是姊妹类群,裂叶地黄处于系统树的基部(图 2)。
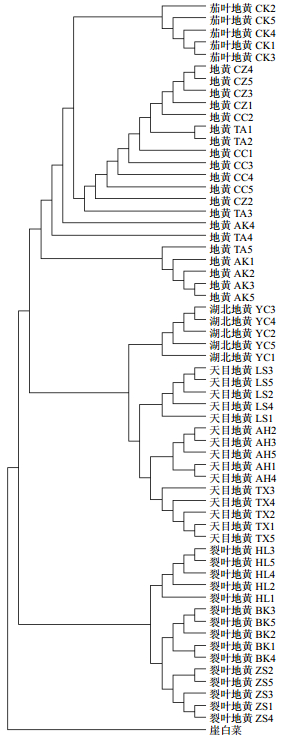
|
图 2 基于6个叶绿体基因与13个核基因构建的地黄属植物NJ树 Fig.2 NJ tree constructed with six chloroplast genes and 13 nuclear gene fragments |
3 讨论 3.1 DNA条形码的比较
有研究认为,叶绿体基因进化速率慢,使用叶绿体基因片段对植物类群鉴定存在上限,即增加片段组合也不能提高分辨率[19]。研究结果与此稍有不同,基因数目的增加有助于物种鉴定成功率的提高。以建树法为例,2个基因片段组合的物种分辨率稍高于单个基因(组合前后平均值分别为75%与83%),但多于2个基因组合后,物种鉴定成功率快速增加(3、4个基因组合的分辨率分别为93.7%、100%,图 1)。究其原因,可能与所选叶绿体基因具体的进化规律有关。生命条码联盟植物工作组推荐的DNA条形码,比如matk和rbcL进化速率较慢,适合科属以上水平的系统学分析,对属下水平的物种鉴定提供的信息非常有限[20]。而本研究分析所用的psbA-trnH、trnL-F、trnM-V、trnS-G片段全部属于非编码片段,进化速率快于基因编码区。特别是trnS-G基因片段,序列中含有类似小卫星重复区域,插入/缺失反复出现,拥有这一特征的基因其进化速率可能比普通叶绿体基因进化速率高几个数量级[21]。
与叶绿体基因结果不同,单拷贝核基因片段数目增加并不能显著提高地黄属物种的鉴定成功率,这主要有2个原因:一是本研究所选单拷贝核基因是根据APOV等模式植物筛选的,基因在类群间高度保守,并且绝大部分区段都是基因编码区,进化速率较慢[22];另一个原因是不同基因在地黄属种内种间的变异程度不同,进化规律的冲突也会导致片段组合后也会降低物种的鉴定效果。
本研究中,ITS在3种分析方法中均表现出很高的物种分辨率(98%~100%),证明ITS确实应该作为地黄属的DNA核心条形码,这与先前的研究相一致[3-4, 13]。4个叶绿体基因片段(psbA-trnH、trnL-F、trnM-V、trnS-G)在地黄属表现的分辨率相近,种间最小距离都大于种内最大距离(表 2),ITS与其中任何一个序列组合均能将地黄属物种100%鉴定成功,因此它们均可以作为备选DNA条形码。同时建议单拷贝核基因R255与R257也可以考虑作为备选项。虽然这些核基因在地黄属物种鉴定中表现逊于叶绿体备选条形码,但依据PWG距离法仍能100%鉴定地黄属5个物种,并且这2个基因能各自独立地把地黄与其他二倍体物种区分开来(表 2),这为中药地黄的分子鉴定提供了更多有效手段。随着基因组数据的增长与开发,单拷贝核基因将会提供更多合适的单拷贝核基因DNA条形码。
3.2 地黄属种间亲缘关系通过19个基因片段构建的地黄属NJ树强烈支持地黄属二倍体物种都是单系群(图 2),支持率茄叶地黄与地黄关系密切,这2个四倍体,在遗传上显著区别于本属二倍体物种,这与前人研究相一致[4, 11, 13, 23]。茄叶地黄处于地黄分布区西南缘,前者可能只是地黄地理扩张过程中新形成的生态型。先前叶绿体证据,支持湖北地黄与天目地黄是并系群[24]。本研究综合了大量的核基因与叶绿体基因数据,不支持上述结论,NJ树显示湖北地黄与天目地黄是姊妹类群,二者组成单系群,可能拥有最近共同祖先。以前研究均认为天目地黄是本属基本类群[13, 23-24],但本研究认为裂叶地黄可能更近于祖先状态。裂叶地黄现今主要分布于大巴山东缘,其分布范围与地黄属模拟得到的祖先分布区相一致[14],裂叶地黄可能是地黄属祖先类群原地分化的产物,而天目地黄可能是祖先物种向东扩张过程中与祖先形成地理隔离产生的衍生种,其具体机制有待深入研究。关于四倍体地黄-茄叶地黄的起源方式一直受到各项研究的关注,前人研究推测地黄-茄叶地黄可能源于天目地黄与裂叶地黄的杂交再经过基因组加倍而形成的异源多倍体[14, 23]。本研究的NJ树显示地黄-茄叶地黄分支与天目地黄-湖北地黄祖先是姊妹类群。这一系统发育关系既可以解释为裂叶地黄与天目地黄-湖北地黄的祖先物种分化后再杂交形成四倍体的地黄-茄叶地黄,也可以解释为地黄-茄叶地黄是裂叶地黄一部分群体直接加倍后而形成的同源四倍体。因此关于四倍体的起源方式仍需要更多的研究工作进行深入探讨。
4 结论通过比较12个单拷贝核基因、ITS与6个叶绿体基因片段作为DNA条形码的可行性,确定了ITS作为核心条形码的地位,同时推选叶绿体片段psbA-trnH、trnL-F、trnM-V、trnS-G作为备选项,核基因RR255与R257可作为地黄专用鉴定条形码。这些DNA标记为探讨地黄属物种间的亲缘关系提供了新的资料。
| [1] | Hebert P D H, Cywinska A, Ball S L, et al. Biological identifications throught DNA barcodes[J]. Proc Roy Soc Lond, Ser B:Biol Sci, 2003, 270(1512): 313–321. DOI:10.1098/rspb.2002.2218 |
| [2] | Group CPW. A DNA barcode for land plants[J]. Proc Nat Acad Sci USA, 2009, 106: 12794–12797. DOI:10.1073/pnas.0905845106 |
| [3] | Li D Z, Gao L M, Li H T, et al. Comparative analysis of a large dataset indicates that internal transcribed spacer (ITS) should be incorporated into the core barcode for seed plants[J]. Proc Nat Acad Sci USA, 2011, 108(49): 19641–19646. DOI:10.1073/pnas.1104551108 |
| [4] | 夏至, 王璐静, 黄勇, 等. 地黄属植物DNA条形码鉴定及地黄栽培起源研究[J]. 中草药, 2016, 47(4):648–654. |
| [5] | 胡兆锋, 向秋云, 黄宏文. 四照花亚属的nrDNA ITS致同进化不完全[J]. 热带亚热带植物学报, 2013, 21(1):29–37. |
| [6] | Duarte J M, Wall P K, Edger P P, et al. Identification of shared single copy nuclear genes in Arabidopsis, Populus, Vitis and Oryza and their phylogenetic utility across various taxonomic levels[J]. BMC Evol Biol, 2010, 10: 61. DOI:10.1186/1471-2148-10-61 |
| [7] | Zhang N, Zeng L, Shan H, et al. Highly conserved low-copy nuclear genes as effective markers for phylogenetic analyses in angiosperms[J]. New Phytol, 2012, 195(4): 923–937. DOI:10.1111/j.1469-8137.2012.04212.x |
| [8] | Du S, Wang Z, Zhang J. A novel set of single-copy nuclear DNA markers for the genetic study of Salicaceae[J]. Gen Mol Res, 2014, 13(3): 4911–4917. DOI:10.4238/2014.July.4.5 |
| [9] | Tonnabel J, Olivieri I, Mignot A, et al. Developing nuclear DNA phylogenetic markers in the angiosperm genus Leucadendron (Proteaceae):a next-generation sequencing transcriptomic approach[J]. Mol Phylogenet Evol, 2014, 70: 37–46. DOI:10.1016/j.ympev.2013.07.027 |
| [10] | Reveal J L. Summary of recent systems of angiosperm classification[J]. Kew Bulletin, 2011, 66(1): 5–48. DOI:10.1007/s12225-011-9259-y |
| [11] | Huang J, Zeng S Y, Zhao J H, et al. Genetic variation and phylogenetic relationships among Rehmannia (Scrophulariaceae) species as revealed by a novel set of single-copy nuclear gene markers[J]. Biochem Syst Ecol, 2016, 66: 43–49. DOI:10.1016/j.bse.2016.03.011 |
| [12] | 李宏庆, 刘国丽. 地黄属的一个新异名[J]. 广西植物, 2012, 32(1):23–26. |
| [13] | 程芳婷, 李忠虎, 刘春艳, 等. 地黄属植物的DNA条形码研究[J]. 植物科学学报, 2015, 33(1):25–32. |
| [14] | 刘占林, 李建芳. 地黄属分子系统学分析[J]. 西北植物学报, 2014, 34(1):77–82. |
| [15] | Hall T A. BioEdit:a user-friendly biological sequence alignment editor program for Windows 95/98/NT[J]. Nuclear Acids Symposium Series, 1999, 41(1): 95–98. |
| [16] | Tamura K, Stecher G, Peterson D, et al. MEGA6:Molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725–2729. DOI:10.1093/molbev/mst197 |
| [17] | Meier R, Shiyang K, Vaidya G, et al. DNA barcoding and taxonomy in Diptera:a tale of high intraspecific variability and low identification success[J]. Syst Biol, 2006, 55(5): 715–728. DOI:10.1080/10635150600969864 |
| [18] | Vaidya G, Lohman D J, Meier R. SequenceMatrix:concatenation software for the fast assembly of multi-gene datasets with character set and codon information[J]. Cladistics, 2011, 27(2): 171–180. DOI:10.1111/cla.2011.27.issue-2 |
| [19] | Lahaye R, van der Bank M, Bogarin D, et al. DNA barcoding the floras of biodiversity hotspots[J]. Proc Nat Acad Sci USA, 2008, 105(8): 2923–2928. DOI:10.1073/pnas.0709936105 |
| [20] | Dong W, Liu J, Yu J, et al. Highly variable chloroplast markers for evaluating plant phylogeny at low taxonomic levels and for DNA barcoding[J]. PLoS One, 2012, 7(4): e35071. DOI:10.1371/journal.pone.0035071 |
| [21] | Cozzolino S, Noce M E, Musacchio A, et al. Variation at a chloroplast minisatellite locus reveals the signature of habitat fragmentation and genetic bottlenecks in the rare orchid Anacamptis palustris (Orchidaceae)[J]. Am J Bot, 2003, 90(12): 1681–1687. DOI:10.3732/ajb.90.12.1681 |
| [22] | Zeng L, Zhang Q, Sun R, et al. Resolution of deep angiosperm phylogeny using conserved nuclear genes and estimates of early divergence times[J]. Nat Commun, 2014, 5: 4956–4965. DOI:10.1038/ncomms5956 |
| [23] | Albach D C, Li H Q, Zhao N, et al. Molecular systematics and phytochemistry of Rehmannia (Scrophulariaceae)[J]. Biochem Syst Ecol, 2007, 35(5): 293–300. DOI:10.1016/j.bse.2006.11.003 |
| [24] | Xia Z, Wang Y Z, Smith J F. Familial placement and relations of Rehmannia and Triaenophora (Scrophulariaceae) inferred from five gene regions[J]. Am J Bot, 2009, 96(2): 519–530. DOI:10.3732/ajb.0800195 |
 2017, Vol. 48
2017, Vol. 48


