黄芪Astragalus membranaceus(Fisch.) Bunge是豆科(Leguminasae)黄芪属Astragalus Linn.多年生草本药用植物[1]。黄芪是我国40种常用大宗药材品种之一,广泛用于中成药之中,是一种重要的主原料药材之一,药用量大且历史悠久。黄芪经典名方包括玉屏风散、补中益气汤、黄芪建中汤和归脾汤等[2]。《神农本草经》提出黄芪具有补气升阳、固表止汗、利水消肿、生津养血等功效。现代临
床黄芪常用于治疗心气虚损、血脉瘀阻之病毒性心肌炎、心功能不全及脾虚湿困之肝炎等疾病[1]。近年来,人们对保健需求的进一步增加导致黄芪需求量剧增,我国各地出现了多个黄芪栽培品种,黄芪药材品质参差不齐,研究黄芪药用活性成分的形成和调控机制,加强种质资源鉴定、保存和良种选育尤为必要。
MicroRNA(miRNA)是动植物中广泛分布的一类非编码内源性单链小分子RNA,长度在19~25个核苷酸,被美国《科学》杂志列为2002年世界十大科技突破之首[3]。最早发现的miRNA是秀丽隐杆线虫Caenorhabditis elegans的lin-4和let-7[4-5]。植物miRNA最早在拟南芥中被发现[6]。到目前为止,已有206个物种的24 521种miRNA被发现[7]。miRNA主要在转录后水平通过与靶基因的碱基互补配对,进而介导靶基因的降解或翻译抑制来调控基因表达。在植物中,miRNA通过负调控SPL(SQUAMOSA promoter-binding protein-like)、TCP和MYB等转录因子和其他重要的功能基因,广泛参与植物生长发育、形态建成、逆境胁迫应答和次生代谢调控等多种生物学过程,发挥重要的作用[8]。
已进行的植物miRNA研究主要针对拟南芥、水稻等模式植物和作物进行。近年来,对药用植物miRNA的研究受到越来越多的重视,研究人员利用高通量测序、miRNA芯片、生物信息学分析等方法研究了人参、红豆杉、半夏、千里光和毛地黄等中药的miRNA,从这些药用植物中鉴定出一些miRNA,对部分miRNA在次生代谢中的调控作用进行分析,取得了初步进展[9-10]。黄芪作为中国传统的大宗药材,在我国中医药中占重要的地位。迄今为止,研究者们对于黄芪的化学成分、药理作用、炮制和贮藏养护方法方面进行了大量研究,并取得了较大进展[11];但是对于黄芪的药用活性成分的形成、调控的分子机制还不清楚,对于与这些调控机制相关的重要遗传因子--miRNA的相关研究也未见报道。本实验拟应用生物信息学技术与实验方法,鉴定黄芪的miRNA,并分析干旱胁迫对黄芪miRNA表达的影响,为进一步研究黄芪miRNA的生物学功能,特别是在次生代谢途径调控中的作用奠定基础。
1 材料黄芪种子来自内蒙古自治区鄂尔多斯市,其植株经中央民族大学生命与环境科学学院刘博博士鉴定为黄芪Astragalus membranaceus (Fisch.) Bunge。
Trizol试剂购自美国英杰生命技术有限公司(Invitrogen),逆转录试剂和荧光定量PCR试剂盒购自天根生化科技(北京)有限公司,引物由生工生物工程(上海)股份有限公司合成。其余试剂均为分析纯。
2 方法 2.1 胁迫处理播种前先将黄芪种子于50 ℃温水中浸泡12~24 h,然后放在湿毛巾中催芽,待露白后播种。土壤由蛭石和营养土混合而成(体积比3:1)。黄芪生长8周后用20% PEG6000进行干旱处理3 d,取对照组和干旱处理组的根与叶用液氮速冻后于-80 ℃保存备用。
2.2 黄芪核酸数据的获取和转录本数据拼接在NCBI网站(http://www.ncbi.nlm.nih.gov)下载黄芪的高通量测序数据。数据登录号为ERR706814和SRR923811。数据滤过使用软件Trimmomatic(http://www.usadellab.org/cms/index.php?page=trimmomatic)[12]和SolexaQA(http://solexaqa.sourceforge.net/)[13]。拼接使用Trinity(http://trinityrnaseq.github.io/)[14],拼接使用默认参数进行。
2.3 黄芪miRNA及其靶基因的预测和注释miRNA的具体预测流程按照文献方法[15]进行。使用psRNAtarget软件进行靶基因的预测[16],预测使用默认参数,即最大期望值(maximum expectation)为3.0,互补打分序列长度(length for complementtarity scoring)为20 bp,靶基因的最大数量(top target genes for each small RNA)为200,靶基因可及性(target accessibility)为25。通过将靶基因序列比对到拟南芥蛋白序列来注释靶基因,截取标准为E值 < 1×10-5。
2.4 茎环引物实时荧光定量PCR用Trizol试剂提取植物组织总RNA,具体方法参照说明书。分别提取对照组和干旱组的黄芪幼苗根、叶的总RNA,使用NanoDropTM2000分光光度计(Thermo Fisher,USA)检测总RNA的浓度和纯度,1.2%琼脂糖凝胶电泳检测总RNA的完整性。
miRNA的表达检测采用茎环引物实时荧光定量PCR法。随机选取8个miRNA,分别设计逆转录茎环引物及荧光定量PCR引物(表 1)。使用逆转录引物进行第一链反转录,然后用荧光定量PCR检测miRNA的表达情况。荧光定量PCR反应在MyiQ2型荧光定量PCR仪(Bio-Rad)上运行,按照FastFire快速荧光定量PCR预混试剂(TIANGEN,CHINA)说明书步骤在冰上配制反应体系:2×FastFire qPCR PreMix 10 μL,PCR正向引物(10 μmol/L)0.6 μL,PCR反向引物(10 μmol/L)0.6 μL,cDNA 2.0 μL,ddH2O(灭菌蒸馏水)补足至20 μL,设置温度梯度摸索PCR最佳退火温度,最终确定反应条件为95 ℃预变性1 min,95 ℃变性5 s,56 ℃退火延伸15 s,40个循环,延伸阶段收集信号,从56 ℃到95 ℃,每个循环增加0.5 ℃,持续0.05 s获得解链温度,采集溶解曲线荧光信号。反应结束后确认实时荧光定量PCR的扩增曲线和熔解曲线,记录Ct值。最后利用3.5%琼脂糖凝胶电泳对茎环引物实时荧光定量PCR产物进行验证。定量PCR反应以U6作为内参,其正向引物序列为5’-GAGTTGAGCAACATGGAAACC-3’,反向引物序列为5’-TGGATTTGTGCAGCTTCTTTT-3’。每个反应设置3个平行,每个实验组设置3个生物学重复。采用2-ΔΔCt法来计算干旱处理前后miRNA的相对表达量[17]。
|
|
表 1 用于实验验证的miRNA及其引物 Table 1 miRNAs used for experimental validation and their primers |
3 结果与分析 3.1 黄芪转录本数据的拼接
首先用Trimmomatic软件下载的黄芪高通量数据进行滤过,获得去除测序接头、去除低质量数据、去除冗余序列的可用于拼接的数据。然后用Trinity拼接。拼接结果的主要参数见表 2。
|
|
表 2 拼接获得的黄芪转录本数据基本情况 Table 2 Statistics of assembled transcripts of A. membranaceus |
3.2 预测到的黄芪miRNA、前体及其二级结构
以已知植物miRNA前体序列为探针与拼接获得的86 647条黄芪转录本序列进行BLASTN比对分析,筛选出546条转录本序列。去除其中的蛋白编码基因后,得到109条潜在的miRNA前体序列,进一步对这些转录本进行手动筛选,确定了其中能够用Mfold软件折叠形成稳定的茎环结构,并符合相应筛选标准的核酸序列共17条,代表17个茎环前体序列(表 3和图 1)。在这些前体序列上分布着分属于17个不同家族的17个miRNA的成熟序列(表 4)。其中,家族成员最多的是miR156家族,有3个成员;其次为miR159、miR166和miR482家族,有2个成员。
|
|
表 3 黄芪中新发现miRNA前体 Table 3 Stem-loop precursors of predicted miRNA from A. membranaceus |
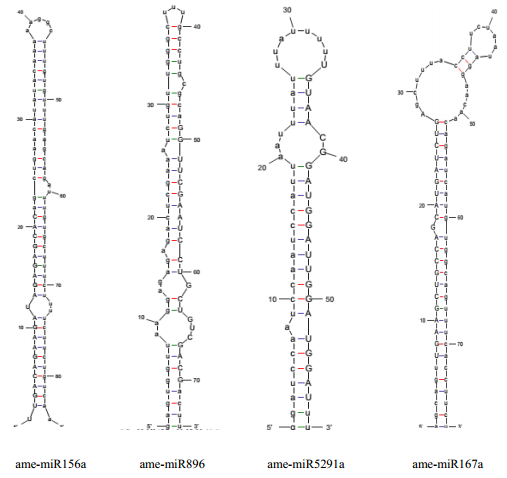
|
miRNA的成熟形式用大写字母表示 The miRNA sequences are shown in capitals 图 1 部分miRNA前体的二级结构 Fig.1 Secondary structures of four predicted stem-loop precursors |
|
|
表 4 黄芪中新发现的miRNA Table 4 Novel miRNAs predicted from A. membranaceus |
在黄芪中预测到的17条成熟miRNA的长度范围为20~24 nt,其中21 nt长度的miRNA最多(7/17,41%),其次为22 nt的miRNA(5/17,29%),其他长度的miRNA数量较少。预测的17个茎环前体的序列长度介于58~211 nt,这些前体都能折叠成稳定的二级结构,最小自由能(MFE)介于76.94~18.8 KJ/moL,最小自由能指数(MFEI)介于0.57~1.68(表 3)。
本研究考察了17个预测的黄芪miRNA与其他植物来源的同源miRNA的序列相似性(表 4)。59%(10/17)的miRNA与其他植物中的对应miRNA的序列完全相同,12%(2/17)的miRNA出现1个差异碱基,出现多于1个差异碱基的miRNA共有5个,占29%。这说明本研究预测的miRNA多数为保守miRNA。此外还考察了预测的17个黄芪miRNA前体与其他植物来源的miRNA前体的序列相似性(表 3)。发现所有前体与其他植物来源的miRNA前体都有程度不同的相似性,E值最小为3×10-23,最大为9×10-8。这一结果说明,与蛋白编码基因类似,同源miRNA的成熟形式和茎环结构前体在序列上也具有保守性。
3.3 黄芪miRNA的验证为了确定生物信息学预测中的miRNA真实存在,本研究随机选取了8条新发现的miRNA,通过茎环引物实时荧光定量PCR实验进行验证。结果显示,8条miRNA荧光定量扩增产物的熔解曲线均为单峰,将8条miRNA荧光定量PCR产物进行琼脂糖凝胶电泳,8条miRNA均出现明显单一条带,大小约60 bp,与荧光定量PCR扩增产物的实际大小一致(图 2),证明生物信息学预测的miRNA在黄芪中确实存在。
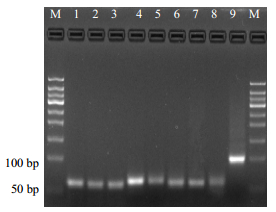
|
M-Marker 1-miR156b-3p 2-miR159e-3p 3-miR168a-3p 4-miR156a-5p 5-miR166l-3p 6-miR167a-5p 7-miR2118-3p 8-miR3630-3p 9-U6 图 2 miRNA茎环引物实时荧光定量PCR产物电泳图 Fig.2 Electrophoresis of miRNA stem-loop qRT-PCR products |
3.4 黄芪miRNA靶基因的预测及功能分析
根据预测的17条黄芪miRNA序列以及拼接获得的转录本序列,通过在线预测软件psRNATarget对获得的黄芪保守miRNA的潜在靶基因进行了预测,共预测出17个miRNA的145个靶基因。
采用Blast将这些靶基因在拟南芥基因组中比对,通过分析靶基因在拟南芥中的同源基因来解析其功能(表 5)。结果表明多数靶基因为转录因子编码基因,部分基因编码受体蛋白激酶,糖、脂类、氨基酸等物质代谢相关酶类分子以及防御反应和胁迫响应等相关蛋白。这些miRNA在其他植物中的靶基因多为黄芪miRNA靶基因的同源基因,说明这些miRNA在不同物种中的功能具有一定的保守性。对靶基因的功能分类分析表明,在黄芪中,预测的miRNA参与了转录调节、物质代谢、信号转导、物质运输、胁迫应答以及蛋白翻译后修饰等多种生物学过程。
|
|
表 5 黄芪保守miRNAs靶基因预测结果 Table 5 Novel miRNA targets predicted from A. membranaceus |
3.5 黄芪miRNA的表达模式分析
从预测的黄芪17个miRNA中随机选取8个miRNA进行实时定量PCR,分析干旱胁迫对于这些miRNA表达的影响(图 3和图 4)。图 3的数据显示,干旱胁迫后黄芪叶中miR156b-3p、miR159e-3p、miR168a-3p、miR3630-3p、miR156a-5p、miR166l-3p和miR167a-5p的表达上调,其中miR166l-3p的表达极显著上调(P < 0.01),而miR2118-3p的表达显著下调(P < 0.05)。从图 4可以看出,干旱胁迫后黄芪根中miR159e-3p、miR168a-3p、miR166l-3p的表达表现为下调的趋势,其中miR168a-3p的表达显著下调(P < 0.05),而miR2118-3p的表达则极显著上调(P < 0.01)。表明miR2118、miR166l和miR168a可能参与黄芪的干旱胁迫应答过程。
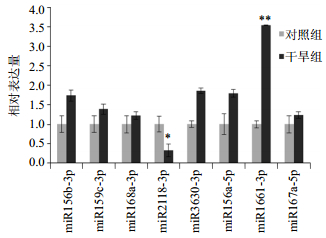
|
与对照组比较:P < 0.05; **P < 0.01,下同 P < 0.05; **P < 0.01 vs control group, same as below 图 3 黄芪叶中8个miRNA在干旱胁迫下的表达情况 Fig.3 Expression of eight miRNAs in A. membranaceus leaves under drought stress |
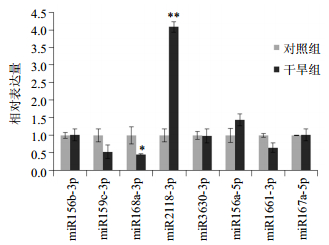
|
图 4 黄芪根中8个miRNA在干旱胁迫下的表达情况 Fig.4 Expression of eight miRNAs in A. membranaceus roots under drought stress |
4 讨论
由于缺乏基因组用于寻找miRNA的前体,对于缺乏基因组信息的植物物种来说,其miRNA鉴定工作难度较大。但随着高通量测序技术的发展,这一局面有所改变。在最近的一些报道中,研究者利用转录组测序技术、小RNA测序以及基因组调查测序(GSS)技术对一些中草药植物进行了测序,并在此基础上进行了miRNA的鉴定,取得了较好的效果。如Qiu等[18]运用高通量测序技术从红豆杉中鉴定出56个保守miRNA。Wu等[19]使用高通量测序技术鉴定人参小分子RNA及其靶基因,鉴定了一些非保守miRNA,其中,miR6136b、miR6135k、miR6139、miR6140d、miR6140与高温和干旱胁迫应答有关。Wu等[20]利用转录组测序分析,从毛地黄中鉴定出13个保守的miRNA,预测到25个miRNA的靶基因。这些研究说明,随着组学技术的快速发展,转录组测序、小RNA测序等高通量测序技术可为非模式植物的miRNA的鉴定提供数据支持,促进中草药miRNA的研究。
本研究利用黄芪高通量测序数据和生物信息学技术鉴定到17个miRNA,预测到145个响应靶基因,这些靶基因参与生长发育、物质代谢、胁迫应答等一系列重要的生物学过程。本研究对其中8个miRNA的干旱胁迫表达模式进行了分析,为理解miRNA在黄芪中的功能奠定了基础。保守miRNA在不同植物中调控的靶基因也存在同源性。例如拟南芥中miR169的靶基因可以编码NF-YA(Nuclear transcription factor Y subunit alpha)转录因子,本研究预测的黄芪miR169的靶基因也包括此靶基因。此外,与其他植物类似,黄芪miRNA的部分靶基因参与植物的抗逆境胁迫过程,例如miR482和miR2118的靶基因所编码的NBS-LRR型抗性蛋白参与植物防御应答过程中的抗病反应。
黄芪的药用部分根部含有黄芪皂苷、多糖、黄酮等多种有效成分,这些药效活性成分是黄芪的次生代谢产物,当受到干旱、冷害等环境胁迫后,植物体内可以通过信号转导、基因表达、酶的合成及各种酶促反应等不同层次影响次生代谢过程,从而显著提高次生代谢物产量[21],而miRNA作为植物逆境胁迫适应过程中的一类重要的调控因子,直接参与胁迫应答过程,分析黄芪miRNA对干旱胁迫的应答对于研究黄芪次生代谢产物的调控机制具有重要意义。本研究对黄芪miRNA进行干旱表达模式分析,发现miR168a、miR2118和miR166l可能在黄芪干旱逆境应答中发挥调控作用。
有研究结果显示,miR168在植物干旱胁迫响应中起到重要的调控作用,如玉米的miRNA168在干旱胁迫后也呈现差异性表达[22];在拟南芥中miR168受到干旱胁迫诱导[23];丹参在干旱胁迫处理后,miR168在丹参叶中的表达呈现出显著性上调[24]。与其他植物类似,本研究中黄芪在干旱胁迫后,miR168亦受到干旱胁迫诱导,在叶中的表达呈现出上调的趋势,在根中是显著下调表达,表明miR168在黄芪的干旱胁迫应答过程中发挥着重要作用。
miR2118在豆科植物中是高度保守的,其功能是调节编码TIR-NBS-LRR抗病蛋白的基因的表达,并且在豆科植物中与干旱胁迫应答密切相关。有研究发现大豆miR2118受干旱胁迫诱导后,在根和叶中的表达均是上调的,这表明miR2118可能参与大豆苗期的干早应答[25]。对miR2118在豆科植物中间锦鸡儿中对干旱胁迫响应的作用进行研究,发现miR2118及其靶基因在根、茎和叶中随着干旱的时间增加,均表现出上调表达,表明miR2118及其靶基因受干旱胁迫诱导,并且能通过调控miR2118及其靶基因的表达使植物适应干旱环境[26]。但在本研究中,在干旱胁迫诱导之后,miR2118在黄芪的根和叶中的表达存在差异,在叶中的表达是显著下调的,而在根中则是极显著上调的,由此说明miR2118在黄芪叶片和根中响应干旱的具体调控作用存在差异。
MIR166基因家族是高度保守、成员众多的一个miRNA基因家族[27],广泛存在于多种植物中。经长期进化而产生的MIR166基因家族在植物生长发育和胁迫响应中发挥非常重要的调控作用[28]。研究表明,miR166是有一个高度保守的靶基因,即编码Ⅲ类同源结构域的亮氨酸拉链蛋白家族(HD-ZIP Ⅲ),HD-ZIP Ⅲ转录因子家族含有4个基因,包括PpHB14、PpHB15、PpHB8和PpREV,这些基因是植物特有的,调节植物发育的关键部分,包括侧器官极性、顶端和侧生分生组织的形成和血管发育等[29]。而本研究预测的miR166的其中2个靶基因就分别含有ATHB15和ATHB14,是属于HD-ZIP Ⅲ转录因子家族基因编码的蛋白,这与已有研究报道相似,说明miR166及其靶基因的高度保守性。在本研究中,受干旱胁迫诱导之后,miR166l在黄芪叶中的表达是极显著上调的,在根中表现出下调的趋势;而miR166在其他植物的干旱胁迫响应研究也有报道,如干旱可以诱导蒺藜苜蓿、野生二粒小麦和大麦中miR166的表达[28];丹参在干旱胁迫处理后,miR166在丹参叶中的表达呈现出显著性上调[24]。综上所述,表明miR166在植物干旱胁迫响应过程中发挥重要的调控作用。
随着药用植物miRNA研究的不断深入,以及高通量测序技术的进一步发展,将发掘到更多的黄芪miRNA及其靶基因。这些工作将为研究miRNA对黄芪次生代谢产物调控提供基础,为进一步利用基遗传工程技术改良黄芪的遗传品质,培育抗逆性好、有效成分量高的黄芪新品种提供科学依据。
| [1] | 中国药典[S].一部.2015. |
| [2] | 达美君. 补药之长--黄芪的名方今用[J]. 家庭健康:医学科普, 2015(2):53. |
| [3] | 艾佳, 李永光, 王涛. 植物逆境microRNA的研究进展[J]. 基因组学与应用生物学, 2014, 33(5):1154–1160. |
| [4] | Lee R C, Feinbaum R L, Ambros V. The Caenorhabditis elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 75(5): 843–854. DOI:10.1016/0092-8674(93)90529-Y |
| [5] | Reinhart B J, Slack F J, Basson M, et al. The 21 nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans[J]. Nature, 2000, 403(6772): 901–906. DOI:10.1038/35002607 |
| [6] | Reinhart B J, Weinstein E G, Rhoades M W, et al. MicroRNAs in plants[J]. Genes & Development, 2002, 16(13): 1616–1626. |
| [7] | Kozomara A, Griffiths-Jones S. miRBase:annotating high confidence microRNAs using deep sequencing data[J]. Nucl Acids Res, 2014, 42(D1): 68–73. DOI:10.1093/nar/gkt1181 |
| [8] | Budak H, Akpinar B A. Plant miRNAs:biogenesis, organization and origins[J]. Funct Integr Genomics, 2015. DOI:10.1007/s10142-015-0451-2 |
| [9] | 曾奇峰, 曾光尧, 周应军. MicroRNA与药用植物[J]. 中国生物化学与分子生物学报, 2015, 31(4):346–351. |
| [10] | 周芳名, 白志川, 卢善发. 药用植物microRNA[J]. 中草药, 2013, 44(2):232–237. |
| [11] | 易炳学, 余书琦, 张金莲, 等. 黄芪的研究概况[J]. 江西中医药大学学报, 2014, 26(2):98–100. |
| [12] | Bolger A M, Lohse M, Usadel B. Trimmomatic:a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114–2120. DOI:10.1093/bioinformatics/btu170 |
| [13] | Cox M P, Peterson D A, Biggs P J. SolexaQA:At-a-glance quality assessment of Illumina second-generation sequencing data[J]. BMC Bioinformatics, 2010, 11: 485–496. DOI:10.1186/1471-2105-11-485 |
| [14] | Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-seq data without a reference genome[J]. Nat Biotechnol, 2011, 29(7): 644–652. DOI:10.1038/nbt.1883 |
| [15] | 李华云, 王宁, 韦春香, 等. 甘草microRNA及其靶基因的生物信息学预测[J]. 中草药, 2016, 47(14):2506–2514. |
| [16] | Dai X, Zhao P X. psRNATarget:a plant small RNA target analysis server[J]. Nucleic Acids Res, 2011, 39: 155–159. DOI:10.1093/nar/gkq766 |
| [17] | Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C (T)) Method[J]. Methods, 2001, 25(4): 402–408. DOI:10.1006/meth.2001.1262 |
| [18] | Qiu D, Pan X, Wilson I W, et al. High throughput sequencing technology reveals that the taxoid elicitor methyl jasmonate regulates microRNA expression in Chinese yew (Taxus chinensis)[J]. Gene, 2009, 436(1/2): 37–44. |
| [19] | Wu B, Wang M, Ma Y, et al. High-throughput sequencing and characterization of the small RNA transcriptome reveal features of novel and conserved microRNAs in Panax ginseng[J]. PLoS One, 2012, 7(9): e44385. DOI:10.1371/journal.pone.0044385 |
| [20] | Wu B, Li Y, Yan H, et al. Comprehensive transcriptome analysis reveals novel genes involved in cardiac glycoside biosynthesis and mlncRNAs associated with secondary metabolism and stress response in Digitalis purpurea[J]. Bmc Genomics, 2012, 13(13): 15–19. |
| [21] | 梁建萍, 贾小云, 刘亚令, 等. 干旱胁迫对蒙古黄芪生长及根部次生代谢物含量的影响[J]. 生态学报, 2016, 36(14):2–3. |
| [22] | 安明.玉米microRNA159和microRNA168在干旱胁迫下的差异表达[D].雅安:四川农业大学, 2011. |
| [23] | Liu H H, Tian X, Li Y J, et al. Microarray-based analysis of stress-regulated microRNAs in Arabidopsis thaliana[J]. Rna-a Publication of the Rna Society, 2008, 14(5): 836–843. DOI:10.1261/rna.895308 |
| [24] | 罗书芳, 崔浪军, 王健, 等. 干旱胁迫下15种丹参miRNAs差异表达分析[J]. 广东农业科学, 2013, 40(5):134–137. |
| [25] | 罗中钦.大豆逆境胁迫相关microRNA的发掘与验证[D].北京:中国农业科学院, 2012. |
| [26] | Wu B F, Li W F, Xu H Y, et al. Role of cin-miR2118 in drought stress responses in Caragana intermedia and Tobacco[J]. Gene, 2015, 574(1): 34–40. DOI:10.1016/j.gene.2015.07.072 |
| [27] | Wang Z, Jin H, Chen Q, et al. Evolution analysis about soybean MIR166 family[J]. J Northeast Agri Univ, 2015, 22(1): 22–29. |
| [28] | 刘海丽.miR192、miR166及其靶基因HD-Zip对水稻镉胁迫应答的功能研究[D].北京:中国计量学院, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10356-1015638581.htm |
| [29] | Zhang C H, Zhang B B, Ma R J, et al. Isolation and expression analysis of four HD-ZIP Ⅲ family genes targeted by microRNA166 in peach[J]. Genetics & Mol Res Gmr, 2015, 14(4): 14151–14161. |
 2017, Vol. 48
2017, Vol. 48


