2. 河南农业大学农学院, 河南 郑州 450002 ;
3. 福建农林大学中药材GAP研究所, 福建 福州 350002
2. College of Agronomy, Henan Agricultural University, Zhengzhou 450002, China ;
3. Institute of Chinese Crude Drugs GAP, Fujian Agriculture and Forestry University, Fuzhou 350002, China
WRKY是一类植物特有的转录因子家族,包含1~2个约60个氨基酸残基的保守结构域,因其含有一段WRKYGQY的核心序列而得名[1]。而且,WRKY结构域的C端还有一个C2H2或C2HC的锌指结构,根据其锌指结构特征可分为Ⅲ类,即I类含有2个WRKY结构域,锌指序列分别为C-X4-C-X22-H-X1-H和C-X5-C-X23-H-X1-H;II类含1个WRKY结构域,锌指序列为C-X4-5-C-X23-H-X1-H;Ⅲ类也是1个WRKY结构域,锌指序列为C-X7-C-X23-H-X1-C[2]。WRKY结构域能够特异地绑定到下游靶基因启动子上的W-box[(C/T) TGAC(T/C)]序列,调控靶基因的转录[3]。已有的研究表明,WRKY不但参与植物的器官发育[4-5]和逆境胁迫响应[6-7],而且在药用植物药效成分生物合成中发挥调控作用。CjWRKY1 RNA干扰可明显抑制黄连Coptis japonica Makino参与小檗碱生物合成途径多个关键酶基因的表达量[8],过表达CrWRKY1能促进长春花Catharanthus roseus (L.) G. Don蛇根碱的合成[9],过表达AaWRKY1可显著提高黄花蒿Artemisia annua L. 青蒿素的量[10],TcWRKY1以DBAT为靶基因参与中国红豆杉Taxus chinensis (Rehder & E. H. Wilson) Rehder紫杉醇的生物合成[11]。
盾叶薯蓣是薯蓣科(Dioscoreaceae)植物盾叶薯蓣Dioscorea zingiberensis C. H. Wright的根茎,含有丰富的甾体皂苷类成分,是合成多种甾体激素和避孕类药的重要原料[12]。盾叶薯蓣是我国特有种,为世界上最好的甾体激素类药源植物之一[13]。然而,迄今为止关于甾体皂苷类成分生物合成的分子机制研究很少,已有的报道也局限于单个酶基因的同源克隆,限制了盾叶薯蓣的进一步开发利用。本研究利用课题组已有的盾叶薯蓣转录组数据,鉴定含有全长开放阅读框(ORF)的WRKY基因,通过生物信息学方法分析蛋白序列特征及表达特性,为进一步研究其在甾体皂苷类成分生物合成的分子功能提供依据。
1 材料与方法 1.1 材料研究用于鉴定WRKY的cDNA序列来源于课题组构建的转录组数据库。用于转录组测序的实验材料为盾叶薯蓣Dioscorea zingiberensis C. H. Wright 1年生根茎和叶片,采用Illumina Hiseq 2000平台进行de novo测序,序列拼接软件为SOAPdenovo。
1.2 盾叶薯蓣WRKY基因的鉴定以水稻Oryza sativa L.、玉米Zea mays L.、拟南芥Arabidopsis thaliana (L.) Heynh. 等物种的WRKY CDS为查询序列,以本地序列比对软件BLASTn进行同源搜索,E-value设定为0.01,以DNAStar软件去除冗余序列。开放阅读框以ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/orfig.cgi)软件进行预测,以DNAStar软件预测编码的氨基酸序列。最后,为了确认获得的序列是WRKY基因,所有的非冗余氨基酸序列提交到http://pfam.sanger.ac.uk网站进行WRKY结构域预测,仅保留具有DBD结构域的序列。
1.3 盾叶薯蓣WRKY蛋白序列特征分析WRKY一级蛋白的理化特征以在线软件ProtParam(http://web.expasy.org/protparam/)进行分析,二级结构特征以在线软件SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)进行预测。WRKY蛋白的亚细胞定位以在线软件PSORT(http://www.genscript.com/psort.html)进行预测,信号肽以在线软件seqNLS(http://mleg.cse.sc.edu/seqNLS/)进行预测。
应用在线软件MEME version 4.11.1(http://meme-suite.org/tools/meme)对盾叶薯蓣WRKY蛋白进行保守结构域预测,设定MotIfs最大值为3,宽度最小值为6、最大值为100。应用结构域在线分析软件SMART(http://smart.embl.de/)对保守结构域进行验证和功能注释。WRKY保守结构域的氨基酸多序列联配用在线软件Clustal Omega(http://www.ebi.ac.uk/Tools/msa/clustalo/)进行比对,GeneDoc软件进行读取分析。三级结构域以在线软件SWISS-MODEL(http://swissmodel.expasy.org/)进行三维建模。
1.4 进化分析拟南芥WRKY的蛋白序列从NCBI数据库中下载,其完整的氨基酸序列用于进化分析。进化树构建分别利用在线分析软件MAFFT(http://www.ebi.ac.uk/Tools/msa/mafft/)和Clustal Omega进行多序列联配,以离线软件MEGA6.06的Neighbor-Joining算法进行bootstrapping分析(1 000次重复)。
1.5 表达分析盾叶薯蓣WRKY基因在叶和根茎中的表达量以Reads Per Kb per Million reads(RPKM)进行计算。
| $RPKM=\frac{{{10}^{6}}C}{NL/{{10}^{3}}}$ |
RPKM(A)为基因A的表达量,则C为唯一比对到基因A的reads数,N为唯一比对到参考基因的总reads数,L为基因A的碱基数
1.6 同源性分析以27个DzWRKY的氨基酸序列为查询序列,利用NCBI在线软件BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi)在拟南芥蛋白质数据库中进行搜索,分别获得相似性最高的AtWRKY,同时在PubMed中检索文献获取AtWRKY的分子功能。根据已经报道的药用植物药效成分相关的WRKY在NCBI上的注册号,获取其氨基酸序列,应用软件DNAman进行序列相似性比对。
2 结果与分析 2.1 盾叶薯蓣WRKY基因家族序列应用BLASTn进行同源比对,结果从盾叶薯蓣转录组数据库中共筛选出109条Unigene序列,片段大小为204~3 574 bp。通过ORF预测,发现27条序列具有完整的ORF(表 1),片段大小从810 bp(CL5012.Contig2)~3 230 bp(CL2497.Contig8),编码的氨基酸从174 aa(Unigene15016)到724 aa(CL2497.Contig8)。27个基因均包含非编码区,其5’ UTR为39~675 bp,3’ UTR为32~408 bp。
|
|
表 1 盾叶薯蓣WRKY基因家族成员信息 Table 1 Information of WRKY genes in rhizomes of D. zingiberensis |
2.2 盾叶薯蓣WRKY家族蛋白的结构特征
DzWRKY蛋白的理化特性分析结果(表 1)表明,27个蛋白的平均相对分子质量为40 100,最小值为19 600(Unigene15016),最大值为78 100(CL2497.Contig8)。等电点pI平均值为7.18,最小值为CL1835.Contig4的5.18,最大值为CL5012.Contig2的10.17,其中有16个蛋白pI小于7,说明多数蛋白表现偏酸性。27个WRKY蛋白的不稳定系数均大于40,最小的为41.63(CL4098.Contig1),最大的为66.95(Unigene25512和CL697.Contig2),说明盾叶薯蓣WRKY家族蛋白均为不稳定蛋白。WRKY家族蛋白的脂溶性指数(AI)介于47.01~72.79,具有较低的AI,说明其在不同环境中稳定性较差。DzWRKY蛋白的亲水性平均系数GRAVY均小于0,说明27个蛋白均属于亲水蛋白。
DzWRKY蛋白的二级结构预测结果(表 1)表明,27个DzWRKY蛋白主要由α螺旋、延伸链和无规卷曲组成,β转角的比例均较小。无规卷曲所占比例较大,18个蛋白的无规卷曲比例超过50%,仅有1个蛋白(CL7347.Contig1)比例低于40%。22个DzWRKY蛋白表现为无规卷曲>α螺旋>延伸链>β转角,其余5个蛋白CL2497.Contig1、CL2497.Contig2、CL4098.Contig1、CL4098.Contig3、Unigene15016表现为无规则卷曲>延伸链>α螺旋>β转角。
PSORT预测DzWRKY蛋白的亚细胞定位结果(表 1)表明,27个蛋白可能的分布主要有细胞核、细胞质、氧化物酶体、线粒体、细胞骨架等,所有DzWRKY蛋白分布在细胞核中的概率均超过65%,其中Unigene34566和CL4098.Contig1分布在细胞核中的概率超过90%,说明DzWRKY蛋白多数应为核蛋白。
2.3 盾叶薯蓣WRKY家族蛋白的结构域具有WRKY结构域为WRKY蛋白最典型的特征。为了分析盾叶薯蓣WRKY的保守结构域,以MEME 4.9.1在线软件预测了假定的Motif,结果显示,检测发现了3个Motif(图 1-A)。分析3个motif在DzWRKY蛋白上的分布规律发现,26个蛋白具有Motif 1和Motif 2,此外CL4098.Contig1、CL4098.Contig3、CL2497. Contig1、CL2497.Contig8、Unigene1393、Unigene14939等6个蛋白还具有1个Motif 3。只有1个蛋白CL5012.Contig2没有检测到Motif分布。进一步分析保守的motif序列特征发现(图 1-B),Motif 1与Motif 2合在一起为典型WRKY蛋白的保守结构域,含有一段高度保守的核心序列WRKYGQK,C′端还有一个典型的C2H2或C2HC锌指结构。Motif 3为一段保守的WRKY结构域,具有核心序列WRKYGQK和一段C2H2锌指结构。说明CL4098.Contig1、CL4098.Contig3、CL2497.Contig1、CL2497.Contig8、Unigene1393、Unigene14939等6个蛋白具有2个WRKY保守结构域,属于第I类WRKY蛋白[2]。
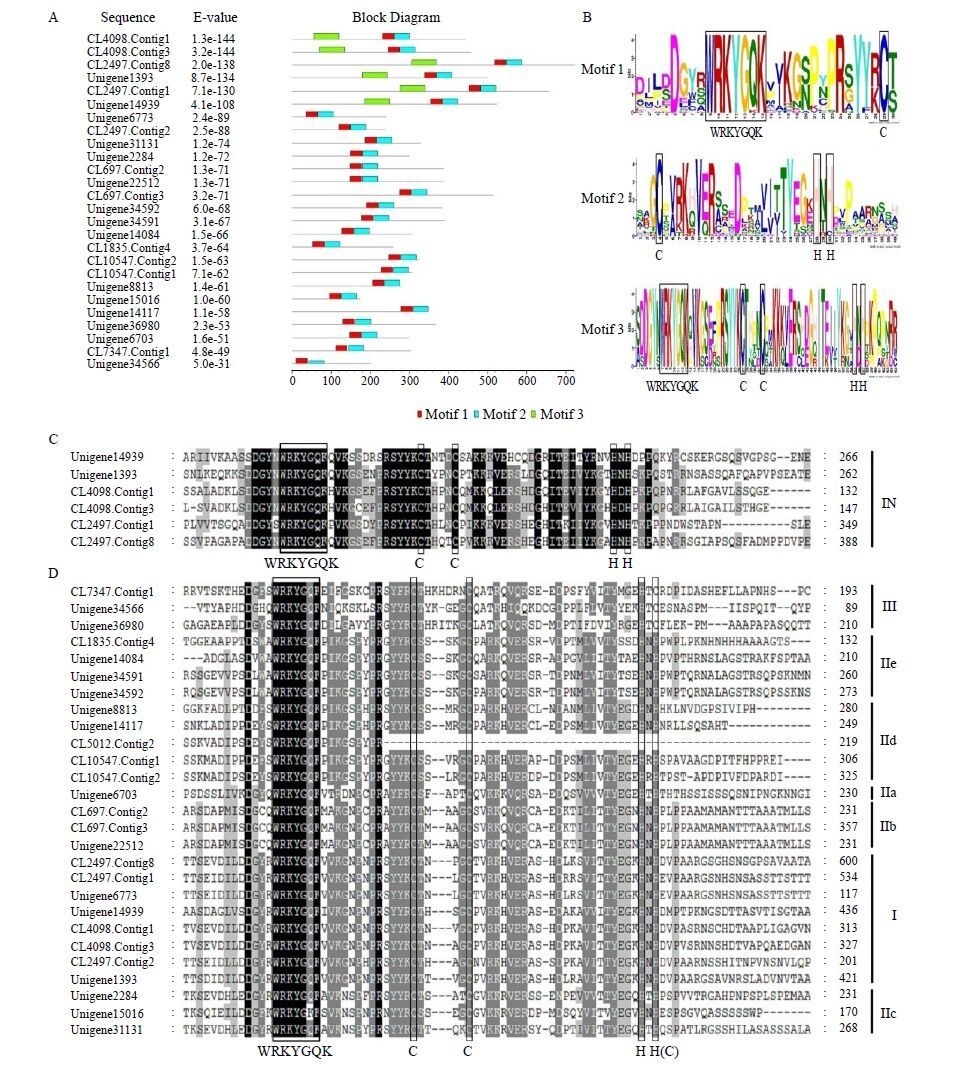
|
A-WRKY蛋白保守结构域分布图 B-保守的结构域基序 C-N′端WRKY结构域氨基酸序列比对 D-C′端WRKY结构域氨基酸序列比对;一致序列显示黑色背景,高度保守的序列显示深灰色背景,类似的序列显示浅灰色背景 图 1 盾叶薯蓣WRKY蛋白结构域分析 Fig.1 Analysis on WRKY domain sequences in rhizomes of D. zingiberensis |
为了分析DzWRKY蛋白N′端WRKY结构域的序列特征,以Clustal Omega对CL4098.Contig1、CL4098.Contig3、CL2497.Contig1、CL2497.Contig8、Unigene1393、Unigene14939等6条氨基酸序列进行多序列联配,结果(图 1-C)表明,6个蛋白N′端WRKY结构域序列保守程度很高,核心序列WRKYGQK,锌指结构为C2H2型,序列为C-X4-C- X22-H-X1-H。进而以Clustal Omega所有DzWRKY蛋白的氨基酸序列进行多序列联配,结果(图 1-D)表明,27个DzWRKY均包含核心序列WRKYGQK,CL5012.Contig2缺失C′末端的锌指结构域。CL7347.Contig1、Unigene34566、Unigene36980的锌指结构为C2HC型,序列为C-X6-7-C-X23-24-H-X1-C,应为第Ⅲ类WRKY蛋白。其余23个DzWRKY的锌指结构为C2H2型,应为第I类和第II类WRKY蛋白。由于第I类C′端WRKY蛋白和第II类c亚类WRKY蛋白的锌指结构序列均为C-X4-C- X23-H-X1-H,第II类a、b、d、e亚类的WRKY蛋白的锌指结构序列均为C-X5-C-X23-H-X1-H,应结合进化分析进一步区分。
2.4 盾叶薯蓣WRKY家族蛋白的三维结构为了进一步了解盾叶薯蓣WRKY蛋白N′端和C′端WRKY结构域的三维结构,通过 Swiss-Model(http://swissmodel.expasy.org)对盾叶薯蓣的27条氨基酸序列进行三维结构同源建模,结果表明它们绝大多数具有十分相似的三维结构。以含有N′端WRKY结构域的CL2497.Contig8 N′端580 aa序列进行同源建模,结果(图 2)发现CL2497.Contig8-N包含4个β折叠,其中β1主要由核心序列WRKYGQK构成,CL2497.Contig8-N与AtWRKY1-C(2ayd.1.A)、AtWRKY4-C(2lex.1.A、1wj2.1.A)的序列一致性分别为59.32%和58.33%。以含有C′端WRKY 结构域的CL2497.Contig8 C′端240 aa序列进行同源建模,结果发现CL2497.Contig8-N包含5个β折叠,其中β2主要由核心序列WRKYGQK构成,CL2497.Contig8-C与AtWRKY1-C、AtWRKY4-C的序列一致性分别为69.86%、85.92%。另外还任选取第Ⅲ类的一个蛋白Unigene36980进行同源建模,发现与CL2497.Contig8-N的三维结构类似,具有4个β折叠,Unigene36980与AtWRKY1-C、AtWRKY4-C的序列一致性分别为50.00%、55.36%。
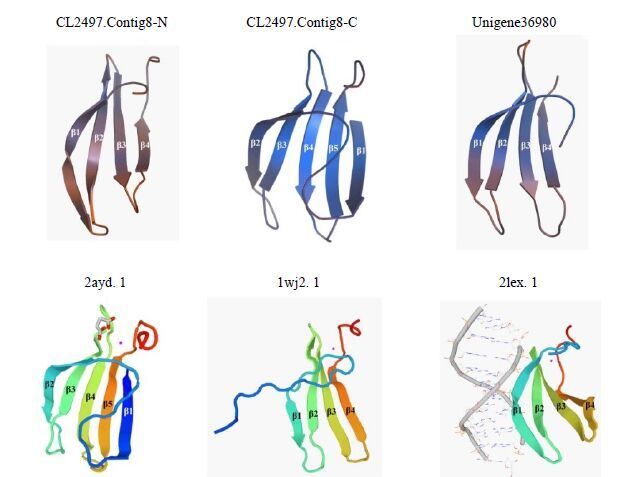
|
图 2 盾叶薯蓣和拟南芥WRKY结构域的三维结构图 Fig.2 Ribbon representation of rhizomes of D. zingiberensis and A. thaliana WRKY domain |
2.5 盾叶薯蓣WRKY蛋白的进化分析
为进一步分析盾叶薯蓣WRKY蛋白之间的进化关系,分别以MAFFT和Clustal Omega软件对27个DzWRKY及与之序列相似性较高的19个拟南芥AtWRKY蛋白的氨基酸序列进行多序列联配,再以MEGA 6.06软件邻接法构建系统进化树,结果表明,2种联配方式获得的进化树类似,都能把46个蛋白分为3个大类群(图 3)。第I类群除包括CL2497.Contig1等含有2个WRKY结构域的6个盾叶薯蓣WRKY蛋白,此外此类群还包括CL2497.Contig2和Unigene6773,与6个I类AtWRKY蛋白聚在一起,锌指结构序列为C-X4-C-X23-H-X1-H。第II类群包括18个DzWRKY蛋白,其中1个IIa、3个IIb、3个IIc、5个IId、4个IIe DzWRKY蛋白,分别与拟南芥同亚类群WRKY蛋白聚在一起,锌指结构序列为C-X4-5-C-X23-H-X1-H。第Ⅲ类群包括CL7347.Contig1、Unigene34566、Unigene36980 3个DzWRKY和3个Ⅲ类AtWRKY蛋白,锌指结构序列为C-X6-7-C-X23-24-H-X1-C。2种联配方式获得的进化树也有一些差异,如Unigene14939在图 3-A中单独为1分支,在图 3-B中与AtWRKY3、AtWRKY4、CL2497.Contig2为同一分支。CL1385.Contig4与AtWRKY69序列相似程度最高,在图 3B中聚在同一个小分支,在图 3-A中单独为1分支。相对而言,以MAFFT进行多序列联配构建的系统进化树较好。
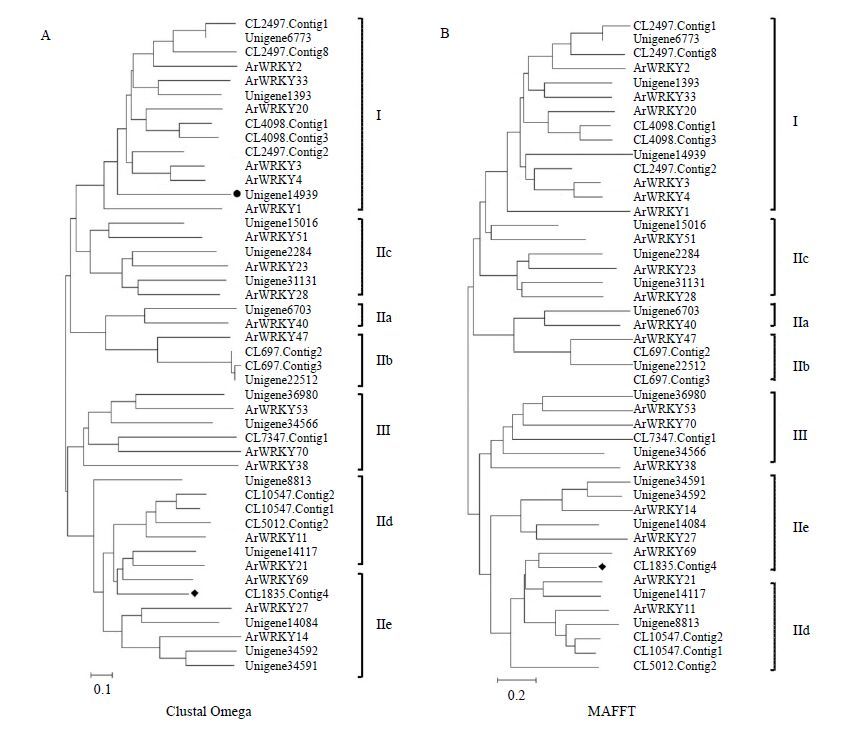
|
A-Clustal Omega联配 B- MAFFT联配 进化树由MEGA 6.06软件构建,在每个节点的数字为1 000次自引导值中该节点存在的百分数 The full-length sequences of the WRKY proteins were aligned using Clustal Omega (A) and MAFFT (B),and the phylogenetic tree was constructed using the neighbor-joining method in the MEGA6 software. Bootstrap values (shown at the corresponding nodes) were obtained from 1 000 replicates and are reported as percentages. The black arcs indicate different groups (or subgroups) of WRKY domains 图 3 盾叶薯蓣和拟南芥WRKY蛋白的系统进化树 Fig.3 Phylogenetic tree of WRKY proteins from rhizomes of rhizomes of D. zingiberensis and A. thaliana |
2.6 盾叶薯蓣WRKY基因的表达特性
薯蓣皂苷(元)主要在根茎中积累,约莫皂苷(元)则主要在叶片中分布[14]。应用盾叶薯蓣根茎、叶的转录组数据,分析了27个DzWRKY基因在根茎和叶中的表达量,结果发现不同DzWRKY基因的表达量有较大差异,而且所有基因在叶片中的表达量均较根茎低(图 4)。在叶中,只有I类的Unigene14939、CL4098.Contig3和IId亚类的Unigene8813、Unigene14117的FPKM值大于10,IIb亚类的3个基因FPKM值均小于0.2。在根茎中有13个基因的FPKM值大于10,其中Unigene14117的FPKM值高达114.81,IIb亚类的3个基因FPKM值均小于8。说明在叶和根茎中高表达的基因主要属于I类WRKY基因和IId亚类WRKY基因。
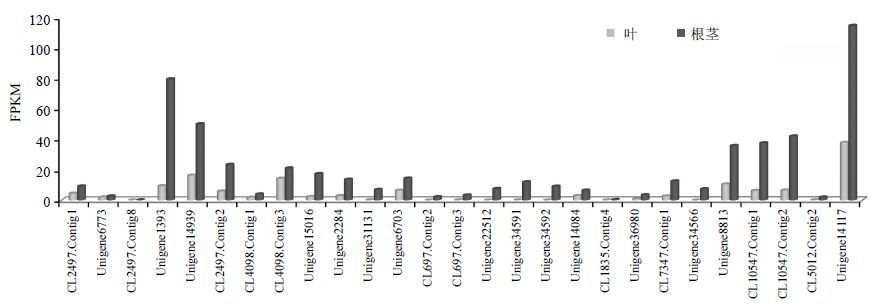
|
图 4 DzWRKY基因在盾叶薯蓣叶和根茎中的表达 Fig.4 Expression level of DzWRKY genes in leaves and rhizome of D. zingiberensis |
2.7 盾叶薯蓣WRKY的功能注释
同源性较高的蛋白往往具有类似的功能[15-16]。为了对获得的27个盾叶薯蓣WRKY基因进行功能注释,在NCBI上通过同源比对,检索获取与它们同源性最高的拟南芥WRKY序列和已知功能的药用植物WRKY,根据已经发表的文章信息对DzWRKY的功能进行预测。结果(表 2)表明,27个DzWRKY分别对应18个同源性较高的AtWRKY,序列相似性从39%到61%,序列一致性较低。除AtWRKY2参与花粉和种子发育外,其他AtWRKY多为植物抗病性调控因子,参与水杨酸、茉莉酸等介导的植物抗性响应。Unigene2284、Unigene31131与黄连CjWRKY1的相似性分别为52%、46%,CL7347.Contig1与长春花CrWRKY1的相似性为48%,均高于与拟南芥WRKY的同源性。Unigene36980与黄花蒿AaWRKY1的相似性为37%,同源性较低。
|
|
表 2 拟南芥与部分药用植物WRKY的分子功能 Table 2 Function of WRKY transcription factors in A. thaliana and several medicinal plants |
3 讨论
本研究鉴定出27个具有完整ORF的盾叶薯蓣WRKY基因,通过对预测的蛋白序列特征分析,发现所有DzWRKY均具有WRKY蛋白特有的结构域核心序列WRKYGQK,6个蛋白具有2个WRKY结构域,21个只有1个WRKY结构域。三维建模表明预测的蛋白与拟南芥AtDWKY具有类似的3D表达分析发现在盾叶薯蓣叶和根茎中高表达的基因主要属于I类WRKY基因和IId亚类WRKY基因,而IIb亚类基因均为低表达基因,说明同类WRKY基因可能存在功能冗余,这与短柄草Brachypodium distachyon (L.) P. Beauv. WRKY家族基因的表达特征类似[32]。研究表明,同一家族基因的表达模式及其编码的蛋白结构特征的相似性可能预示着功能的类似[33],2个结构类似的拟南芥Ⅲ类WRKY转录因子AtWRKY38和AtWRKY62均与组蛋白去乙酰化酶HDA19互作,是拟南芥病害防御的负向调控因子[16]。不同物种WRKY同源蛋白也可能具有类似的分子功能,小麦TaWRKY78与同源的拟南芥AtWRKY20可交叉调控它们的PR4同源基因wPR4e和AtHEL[20]。已经解析的WRKY分子功能主要集中在调控植物的生物、非生物胁迫,个别参与植物的器官发育。植物的抗性响应主要通过激活次生代谢来实现,涉及多个次生代谢途径,其中就包括一些药效成分的合成代谢。这为解析分子生物学基础较薄弱的植物(特别是多数药用植物)基因分子功能提供了参考。
盾叶薯蓣作为世界上最好的甾体激素类药源植物之一,生产上现主要用于提取薯蓣皂苷。由于过度采挖,野生盾叶薯蓣资源不断减少,供需矛盾日益突出[14]。栽培盾叶薯蓣产量不稳定,薯蓣皂苷量偏低,均限制了进一步的开发利用。阐明薯蓣皂苷生物合成的分子途径,克隆参与薯蓣皂苷生物合成的关键酶和转录因子,应用分子修饰方法提高薯蓣皂苷的量,实现盾叶薯蓣的异源生产,能够在一定程度上缓解盾叶薯蓣的供需矛盾。本研究鉴定了27个盾叶薯蓣WRKY转录因子的全长ORF,从推导的蛋白结构、结构域特征、进化分析和功能注释等多个方面深化对DzWRKY的认识,为进一步解析DzWRKY在薯蓣皂苷等药效成分合成中的分子功能奠定基础。
| [1] | 田云, 卢向阳, 彭丽莎, 等. 植物WRKY转录因子结构特点及其生物学功能[J]. 遗传 , 2006, 28 (12) :1607–1612. |
| [2] | Rushton P J, Somssich I E, Ringler P, et al. WRKY transcription factors[J]. Trends Plant Sci , 2010, 15 (5) :247–258. DOI:10.1016/j.tplants.2010.02.006 |
| [3] | Eulgem T, Rushton P J, Robatzek S, et al. The WRKY superfamily of plant transcription factors[J]. Trends Plant Sci , 2000, 5 (5) :199–206. DOI:10.1016/S1360-1385(00)01600-9 |
| [4] | Guo D, Zhang J, Wang X, et al. The WRKY transcription factor WRKY71/EXB1 controls shoot branching by transcriptionally regulating RAX genes in Arabidopsis[J]. Plant Cell , 2015, 27 (11) :3112–3127. DOI:10.1105/tpc.15.00829 |
| [5] | Yang L, Zhao X, Yang F, et al. PtrWRKY19, a novel WRKY transcription factor, contributes to the regulation of pith secondary wall formation in Populus trichocarpa[J]. Sci Rep , 2016, 6 :1234–1240. |
| [6] | Li J, Brader G, Palva E T. The WRKY70 transcription factor:a node of convergence for jasmonate-mediated and salicylate-mediated signals in plant defense[J]. Plant Cell , 2004, 16 (2) :319–331. DOI:10.1105/tpc.016980 |
| [7] | Jimmy J L, Babu S. Role of OsWRKY transcription factors in rice disease resistance[J]. Trop Plant Pathol , 2015, 40 (6) :355–361. DOI:10.1007/s40858-015-0058-0 |
| [8] | Kato N, Dubouzet E, Kokabu Y, et al. Identification of a WRKY protein as a transcriptional regulator of benzylisoquinoline alkaloid biosynthesis in Coptis japonica[J]. Plant Cell Physiol , 2007, 48 (1) :8–18. |
| [9] | Suttipanta N, Pattanaik S, Kulshrestha M, et al. The transcription factor CrWRKY1 positively regulates the terpenoid indole alkaloid biosynthesis in Catharanthus roseus[J]. Plant Physiol , 2011, 157 (4) :2081–2093. DOI:10.1104/pp.111.181834 |
| [10] | Han J, Wang H, Lundgren A, et al. Effects of overexpression of AaWRKY1 on artemisinin biosynthesis in transgenic Artemisia annua plants[J]. Phytochemistry , 2014, 102 :89–96. DOI:10.1016/j.phytochem.2014.02.011 |
| [11] | Li S, Zhang P, Zhang M, et al. Functional analysis of a WRKY transcription factor involved in transcriptional activation of the DBAT gene in Taxus chinensis[J]. Plant Biol , 2013, 15 (1) :19–26. DOI:10.1111/plb.2012.15.issue-1 |
| [12] | 杨如同, 唐世蓉, 潘福生, 等. 药源植物盾叶薯蓣甾体皂苷及皂苷元的研究进展[J]. 中国野生植物资源 , 2007, 26 (4) :1–5. |
| [13] | 谢彩侠, 高山林, 秦惠贞. 不同产地来源的盾叶薯蓣中薯蓣皂苷元含量分析[J]. 河南中医学院学报 , 2009, 24 (1) :58–60. |
| [14] | 李朝阳, 杨朝霞, 陈功锡, 等. 不同居群盾叶薯蓣生长、叶形态及皂苷元含量的研究[J]. 生命科学研究 , 2003, 7 (1) :58–64. |
| [15] | Lai Z, Vinod K M, Zheng Z, et al. Roles of Arabidopsis WRKY3 and WRKY4 transcription factors in plant responses to pathogens[J]. BMC Plant Biol , 2008, 8 (1) :68–75. DOI:10.1186/1471-2229-8-68 |
| [16] | Kim K C, Lai Z, Fan B, et al. Arabidopsis WRKY38 and WRKY62 transcription factors interact with histone deacetylase 19 in basal defense[J]. Plant Cell , 2008, 20 (9) :2357–2371. DOI:10.1105/tpc.107.055566 |
| [17] | Journot-Catalino N, Somssich I E, Roby D, et al. The transcription factors WRKY11 and WRKY17 act as negative regulators of basal resistance in Arabidopsis thaliana[J]. Plant Cell , 2006, 18 (11) :3289–3302. DOI:10.1105/tpc.106.044149 |
| [18] | Guan Y, Meng X, Khanna R, et al. Phosphorylation of a WRKY transcription factor by MAPKs is required for pollen development and function in Arabidopsis[J]. PLoS Genet , 2014, 10 (5) :e1004384. DOI:10.1371/journal.pgen.1004384 |
| [19] | Jiang W, Yu D. Arabidopsis WRKY2 transcription factor mediates seed germination and postgermination arrest of development by abscisic acid[J]. BMC Plant Biol , 2009, 9 (1) :1. DOI:10.1186/1471-2229-9-1 |
| [20] | Proietti S, Bertini L, Van der Ent S, et al. Cross activity of orthologous WRKY transcription factors in wheat and Arabidopsis[J]. J Exp Bot , 2011, 62 (6) :1975–1990. DOI:10.1093/jxb/erq396 |
| [21] | Li J, Brader G, Kariola T, et al. WRKY70 modulates the selection of signaling pathways in plant defense[J]. Plant J , 2006, 46 (3) :477–491. DOI:10.1111/tpj.2006.46.issue-3 |
| [22] | Zheng Z, Qamar S A, Chen Z, et al. Arabidopsis WRKY33 transcription factor is required for resistance to necrotrophic fungal pathogens[J]. Plant J , 2006, 48 (4) :592–605. DOI:10.1111/tpj.2006.48.issue-4 |
| [23] | Mukhtar M S, Deslandes L, Auriac M C, et al. The Arabidopsis transcription factor WRKY27 influences wilt disease symptom development caused by Ralstonia solanacearum[J]. Plant J , 2008, 56 (6) :935–947. DOI:10.1111/tpj.2008.56.issue-6 |
| [24] | Gao Q M, Venugopal S, Navarre D, et al. Low oleic acid-derived repressIon of jasmonic acid-inducible defense responses requires the WRKY50 and WRKY51 proteins[J]. Plant Physiol , 2011, 155 (1) :464–476. DOI:10.1104/pp.110.166876 |
| [25] | Grunewald W, De Smet I, Lewis D R, et al. Transcription factor WRKY23 assists auxin distribution patterns during Arabidopsis root development through local control on flavonol biosynthesis[J]. Proc Natl Acad Sci USA , 2012, 109 (5) :1554–1559. DOI:10.1073/pnas.1121134109 |
| [26] | Chen X, Liu J, Lin G, et al. Overexpression of AtWRKY28 and AtWRKY75 in Arabidopsis enhances resistance to oxalic acid and Sclerotinia sclerotiorum[J]. Plant Cell Rep , 2013, 32 (10) :1589–1599. DOI:10.1007/s00299-013-1469-3 |
| [27] | Xie Y, Huhn K, Brandt R, et al. REVOLUTA and WRKY53 connect early and late leaf development in Arabidopsis[J]. Development , 2014, 141 (24) :4772–4783. DOI:10.1242/dev.117689 |
| [28] | Sun Y, Yu D. Activated expression of AtWRKY53 negatively regulates drought tolerance by mediating stomatal movement[J]. Plant Cell Rep , 2015, 34 (8) :1295–1306. DOI:10.1007/s00299-015-1787-8 |
| [29] | Van Aken O, Zhang B, Law S, et al. AtWRKY40 and AtWRKY63 modulate the expression of stress-responsive nuclear genes encoding mitochondrial and chloroplast proteins[J]. Plant Physiol , 2013, 162 (1) :254–271. DOI:10.1104/pp.113.215996 |
| [30] | Guo C, Guo R, Xu X, et al. Evolution and expression analysis of the grape (Vitis vinifera L.) WRKY gene family[J]. J Exp Bot , 2014, 65 (6) :1513–1528. DOI:10.1093/jxb/eru007 |
| [31] | 杨致荣, 王兴春, 薛金爱, 等. 药用植物长春花WRKY转录因子的鉴定及表达谱分析[J]. 生物工程学报 , 2013, 29 (6) :785–802. |
| [32] | Wen F, Zhu H, Li P, et al. Genome-wide evolutionary characterization and expression analyses of WRKY family genes in Brachypodium distachyon[J]. DNA Res , 2014, 21 (3) :327–339. DOI:10.1093/dnares/dst060 |
| [33] | Wang F, Suo Y, Wei H, et al. Identification and characterization of 40 isolated Rehmannia glutinosa MYB family genes and their expression profiles in response to shading and continuous cropping[J]. Int J Mol Sci , 2015, 16 (7) :15009–15030. DOI:10.3390/ijms160715009 |
 2016, Vol. 47
2016, Vol. 47


