刺五加Eleutherococcus senticosus (Rupr. et Maxim.) Maxim. 是我国传统的珍贵药用植物,具有多种生理活性及药理作用,三萜皂苷类化合物是其主要的活性成分之一[1-3]。目前利用回流浸提法、超声法、微波法等[4]分离、提纯出刺五加皂苷,本实验室又针对三萜皂苷合成途径中的关键酶基因进行克隆研究,成功克隆出β-香树酯醇合成酶(β-amyrin synthase,bAS)、3-羟基-3甲基戊二酰辅酶A还原酶(3-hydroxy-3-methylglutaryl coenzyme A reductase,HMGR)、法尼基焦磷酸合酶(farnesyl diphosphate synthase,FPS)、鲨烯合酶(squatene synthase,SS)等[5]基因,而对于整个基因组的表达规律研究不多,其基因数据库的资源也较少。
近几年来,转录组学技术在揭示细胞生理活动规律和生物代谢机制的研究中起着越来越重要的作用,因此对刺五加转录组的研究势在必行。转录组是指特定生物体在某种状态下所有基因转录产物的总和,转录组研究属于功能基因组学研究的范畴,是连接基因组与蛋白质组的纽带[6]。通过对同一物种不同样品进行转录组分析,在能够得到大量功能基因的基础上,还可获得样品间的表达差异,这对于揭示基因表达与植物表型的关系具有重要意义[7]。在植物分子学研究中,最新的RNA测序技术已经取代基因芯片技术,成为目前从全基因组水平研究基因表达的主流方法。对此,本实验采用转录组测序技术,对皂苷量不同的刺五加进行高通量测序、拼接组装,建立刺五加的转录组数据库,并针对差异性表达基因进行分析。
1 材料与方法 1.1 材料以皂苷高含量(质量分数为3.6%)和低含量(质量分数为2.1%)组的刺五加叶片 [经华北理工大学生命科学学院邢朝斌教授鉴定为五加科植物刺五加Eleutherococcus senticosus (Rupr. et Maxim.) Maxim.]为材料提取总RNA,再逆转录为cDNA,构建转录组数据库。植物总RNA提取试剂盒和逆转录试剂盒购自北京天根生化科技(北京)有限公司。
1.2 转录组数据的组装与分析 1.2.1 转录组数据的组装采用Illumina HiSeq 4000测序技术平台的PE150技术进行测序,将得到的reads进行除杂和冗余处理,获得clean reads,利用段序列组装程序SOAPdenove[8]进行拼接,得到contig,再经过双末端测序法组装得到Unigene,并通过序列聚类软件TGICL[9]做进一步的拼接和去冗余,得到最终的Unigene。
1.2.2 转录组功能预测将最终得到的Unigene进行功能注释,将Unigene序列与蛋白数据库COG、GO、KEGG、Swiss-Prot和nr比对,得到具有同源信息的Unigene。COG是对蛋白质直接进行同源性分类并储存蛋白序列的一个数据库,通过与之比对,可预测刺五加可能存在的功能并进行分类统计。
Geneontology(简称GO),是一个国际标准化的基因功能分类体系,可以对基因和蛋白属性进行全面的描述和限定。为从宏观上认识刺五加的基因功能分布特征,需根据NCBI中的注释信息,利用Blast2GO软件[10]对nr注释信息进行分类,获得Unigene的GO注释,并利用WEGO软件[11]对所有 Unigene做GO功能分类统计。
KEGG是系统分析基因产物在细胞中的代谢途径和基因产物功能的数据库,将刺五加的Unigene比对到KEGG数据库中,获得Unigene的注释信息,而后进行Pathway分析。
1.3 基因差异性表达分析 1.3.1 差异性表达基因的筛选利用华大基因研究开发的两个样本间差异性表达基因筛选算法,算出错误发现率(FDR值),并通过基因的表达量(FPKM值),计算出该基因在不同样本间的差异表达倍数。FDR值与差异表达倍数呈反比关系,FDR值越小,差异表达倍数越大,结果的可信度越高。分析时,将FDR≤0.001和差异倍数≥2作为筛选差异表达基因的标准。
1.3.2 差异性分析对筛选出的基因进行GO功能分析和KEGG Pathway分析,得出2个样本间差异表达基因的GO功能分类注释和显著性富集分析,以及Pathway富集分析,可以更好地定量这些基因在刺五加生命活动中行使的主要生物学功能,显示它们参与的生化代谢途径及信号传导途径。
2 结果与分析 2.1 组装结果总共得到8.34 Gb数据,组装后得到contig 3 387 282条,总长度2.08×108 nt,平均长度为62 nt,N50长度为65 nt。进一步去冗余、拼接,得到最终的Unigene 77 087条,总长度为52 940 804 nt,平均长度为687 nt,大于500 nt 的有26 969条,占总数的34.99%,N50长度为1 207 nt。
2.2 功能预测将最终的Unigene通过Blastx和Blastn与已知的公共数据库COG、GO、KEGG、Swiss-port、nr 5个数据库进行比对,统计获得同源比对信息的Unigene分别有8 084、15 835、6 197、19 450、28 937条,分别占总量的10.47%、20.54%、8.04%、25.23%、37.54%,总共获得同源比对信息的Unigene有29 348条,占38.07%,没有获得同源比对信息的有47 739,占61.93%,获得同源信息的Unigene长度大都在300 nt以上,见表 1。
|
|
表 1 Unigene注释数目统计表 Table 1 Number of annotated Unigene |
Unigene与COG比对的结果显示,刺五加数据库中有8 084个Unigene 分布于25个COG分类中,其中聚类到一般功能预测,复制、重组和修复,转录,碳水化合物转运和代谢,翻译、核糖体结构和生物合成等功能基因分布较多。通过与GO数据库进行比对,共得到71 883个GO功能注释并归类于52个GO分类中,其中生物学过程注释最多,共有34 002条Unigene,占全部的47.30%,其次是分子功能有24 212条,占33.68%,最少的是细胞组分,有13 669条,占19.02%。这3大类又可细分为52个功能亚类,生物学过程20个亚类,分子功能和细胞组分各16个亚类。在生物学过程中,涉及代谢过程、细胞过程和单一有机体过程的基因较多;在分子功能中,涉及催化活性和锚定的基因较多;在细胞组分中,涉及细胞组分、细胞、细胞器和细胞膜的基因较多。具体种类与数量见图 1。
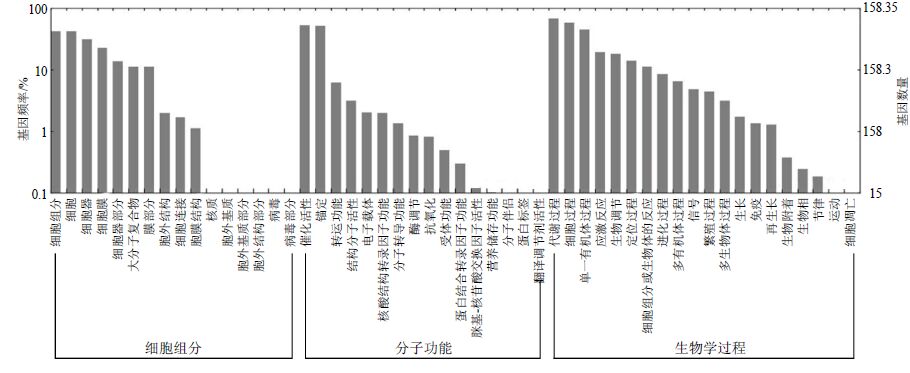
|
图 1 Unigene GO功能分类图 Fig.1 Unigene GO classification |
将测序得到的Unigene与KEGG数据库进行比对,获得Pathway注释信息,比对结果显示,有6 197条Unigene获得6 755个注释(在这之中有多条Unigene涉及2个及以上的通路),共涉及116个KEGG标准代谢通路。其中获得注释量较多的前5个通路分别是:核糖体途径,共有341条,占全部的5.05%;内质网蛋白加工途径,共有230条,占3.39%;植物激素信号转导途径,有228条,占3.37%;氧化磷酸化途径,有226条,占3.34%;RNA转运途径,有212条,占3.13%。其余的主要富集于糖酵解、剪接体、嘌呤代谢等代谢途径。
2.3 基因差异性表达分析利用差异表达基因的筛选方法,筛选出2个样本间的差异性表达基因,共530条,其中高含量组相对于低含量组上调基因有223条,占差异表达基因的42.08%,下调基因307条,占57.92%(图 2)。根据结果可知,上调和下调的基因相差较大,这些上调和下调的基因,为揭示刺五加关键调控基因,以及这些关键调控基因的代谢途径分析提供了可靠的数据。将530条差异性表达基因与数据库COG、GO、KEGG、Swiss-prot和nr进行比对,结果显示有434条基因获得注释。其中获得GO注释的差异表达基因有220条,共获得408个注释,分为生物学过程、细胞组分和分子功能3类,其中生物学过程得到211个功能注释,占51.72%,细胞组分得到55个功能注释,占13.48%,分子功能得到142个功能注释,占34.80%。
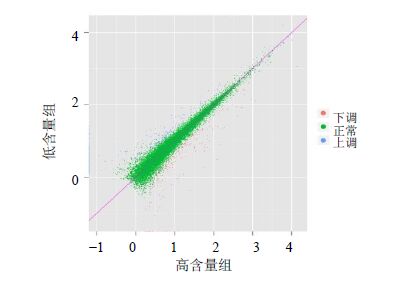
|
图 2 差异性表达基因 Fig.2 Differentially expressed genes |
与KEGG数据库进行比对,共有82条基因获得注释(在这之中有多条基因涉及两个及以上的通路),共涉及40个代谢通路,其中大部分基因归类于遗传信息转导和新陈代谢途径。分类情况见图 3。
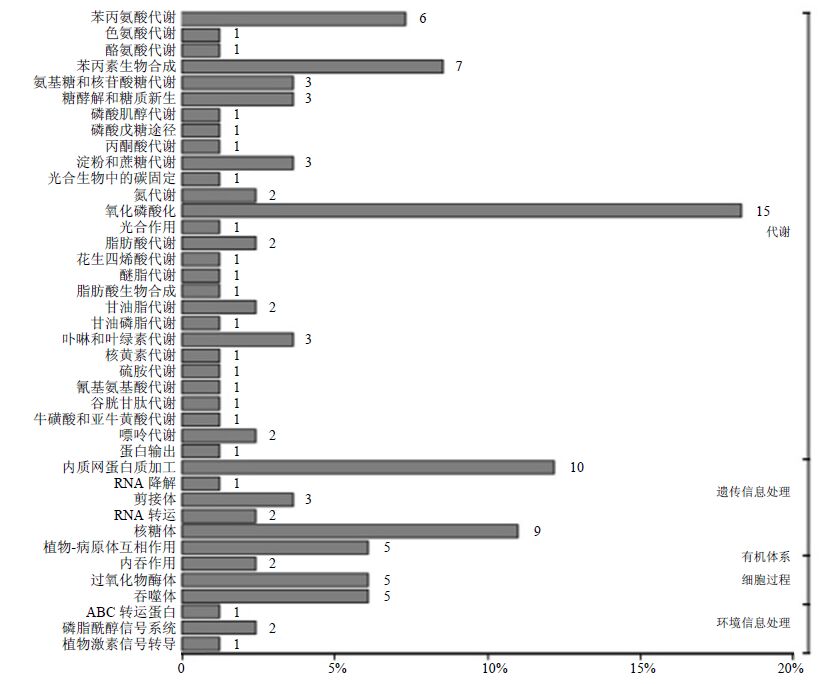
|
图 3 差异性表达基因KEGG分类图 Fig.3 KEGG classification of differentially expressed genes |
显著富集的通路有3条,最为富集的是氧化磷酸化途径,有15条基因,其次是内质网蛋白加工和核糖体途径,分别有10条和9条基因。根据皂苷的合成途径[12]可知,与三萜皂苷合成相关的途径有磷酸戊糖途径、糖酵解和糖质新生、甘油磷脂代谢等途径(表 2),差异性基因共18条。
在皂苷合成途径中还需要一些关键酶的参与,这些酶基因的表达量在2个样本间存在一定差异,但对皂苷合成量具有重大的影响。法尼基焦磷酸合酶(c44724)催化2分子异戊烯基焦磷酸和1分子二甲基烯丙基焦磷酸生成法尼基二焦磷酸,为三萜
|
|
表 2 刺五加三萜皂苷合成相关基因 Table 2 Saponin biosynthesis related genes in E. senticosus |
皂苷合成提供C15骨架。其表达量分别为高含量组:263.308 3(相对值);低含量组:212.644 2(相对值),是典型上调基因。鲨烯环氧酶(c49398)、细胞色素P450酶(c18630)以及β-香树酯醇合成酶(c52011)等关键酶也表达量较高,都属于上调基因。
3 讨论利用RNA-Seq技术,本研究首次获得了刺五加的转录组数据,得到Unigene 77 087条,平均长度为687 nt,组装结果较好。根据GOC、GO和KEGG等的生物信息学结果,揭示了刺五加转录组的整体表达特征,并表明本次转录组测序获得较多可用的基因序列。但未获得功能注释的Unigene 有47 739条,占61.93%。造成这种现象的原因可能有2种:(1)序列太短,难以获得同源信息,本转录组组装得到的Unigene长度在500 nt以下的占65.01%,增加了功能注释和分类的难度;(2)数据库资源的暂时缺乏,注释信息不够全面,使一些表达不够丰富的基因无法得到相应的注释。此现象在已经公布的芝麻[13]、茶树[14]和水稻[15]等5种植物的转录组分析中已经出现过。两个样本间的530条差异性表达基因,通过GO注释和Pathway富集分析,证实皂苷高含量组和低含量组的代谢过程转录水平的差异较大。与皂苷合成相关的表达差异较大的基因有18条,且皂苷合成关键酶基因的表达量存在一定差异,说明基因表达水平的细微差别即可对生物体产生巨大的影响。
| [1] | 邢朝斌, 劳凤云, 田春迎, 等. 刺五加叶柄的体细胞胚胎发生研究[J]. 中草药 , 2009, 40 (8) :1302–1305. |
| [2] | 王子灿, 乔善义, 马安德, 等. 高效液相色谱-质谱联用技术分析刺五加抗疲劳化学成分[J]. 第一军医大学学报 , 2003, 23 (4) :355–357. |
| [3] | 涂正伟, 周渭渭, 单淇, 等. 刺五加的研究进展[J]. 药物评价研究 , 2011, 34 (3) :213–216. |
| [4] | 赵萍, 田晶, 费旭, 等. 刺五加皂苷提取工艺[J]. 大连工业大学学报 , 2013, 32 (5) :316–319. |
| [5] | 邢朝斌, 吴鹏, 修乐山, 等. 刺五加功能基因密码子偏好性分析[J]. 中草药 , 2013, 44 (5) :598–603. |
| [6] | 王东, 曹玲亚, 高建平. 党参转录组中SSR位点信息分析[J]. 中草药 , 2014, 45 (16) :2390–2394. |
| [7] | Jewett M C, Oliveira A P, Patil K R, et al. The role of high-throughput transcriptome analysis in metabolic engineering[J]. Biotechnol Bioprocess Eng , 2005, 10 (5) :385–399. DOI:10.1007/BF02989821 |
| [8] | Li R Q, Zhu H M, Ruan J, et al. De novo assembly of human genomes with massively parallel short read sequencing[J]. Genome Res , 2010, 20 (2) :265–272. DOI:10.1101/gr.097261.109 |
| [9] | Pertea G, Huang X Q, Liang F, et al. TIGR gene indices clustering tools (TGICL):a software system for fast clustering of large EST datasets[J]. Bioinformatics , 2003, 19 (5) :651–652. DOI:10.1093/bioinformatics/btg034 |
| [10] | Conesa A, Gotz S, Garca-gmez J M, et al. Blast2GO:a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics , 2005, 21 (18) :3674–3676. DOI:10.1093/bioinformatics/bti610 |
| [11] | Ye J, Fang L, Zheng H, et al. WEGO:a web tool for plotting GO annotations[J]. Nucl Acids Res , 2006, 34 (Web Server issue) :W293–W297. |
| [12] | 邢朝斌, 王一曼, 陈正恒, 等. 三萜皂苷的生物合成[J]. 生命化学 , 2005, 25 (5) :420–422. |
| [13] | 魏利斌, 苗红梅, 张海洋. 芝麻发育转录组分析[J]. 中国农业科学 , 2012, 45 (7) :1246–1256. |
| [14] | Shi C Y, Yang H, Wei C L, et al. Deep sequencing of the Camellia sinensis transcriptome revealed candidate genes for major metabolic pathways of tea-specific compounds[J]. BMC Genomics , 2011, 12 :131–149. DOI:10.1186/1471-2164-12-131 |
| [15] | Lu T T, Lu G J, Fan D L, et al. Function annotation of the rice transcriptome at single-nucleotide resolution by RNA-seq[J]. Genome Res , 2010, 20 (9) :1238–1249. DOI:10.1101/gr.106120.110 |
 2016, Vol. 47
2016, Vol. 47


