2. 山西大学化学化工学院, 山西 太原 030006
2. College of Chemistry and Chemical Engineering, Shanxi University, Taiyuan 030006, China
尿苷二磷酸葡萄糖焦磷酸化酶(UDP-glucose pyrophosphorylase,UGPase)在所有的真核生物碳水化合物代谢中起重要作用,主要催化反应Glc-1-P+UTP⇋UDPG+PPi[1]。其产物UDPG(尿苷二磷酸葡萄糖)作为葡萄糖基供体主要参与蔗糖、纤维素、胼胝质、糖原等寡糖与多糖的合成,同时也是尿苷二磷酸葡萄糖酸、尿苷二磷酸木糖、尿苷二磷酸半乳糖等核苷二磷酸单糖的前体[2]。在植物的不同组织中UGPase所催化的反应方向不同。在源(source)组织中,UGPase主要参与蔗糖的合成,为蔗糖磷酸合成酶(sucrose phosphate,SPS)提供UDP-葡萄糖,同时UDP-葡萄糖可参与下游多糖、糖脂、糖蛋白等的合成。在库(sink)组织中,UGPase则参与蔗糖的降解,将UDP-葡萄糖转化成葡萄糖-1-磷酸,葡萄糖-1-磷酸则可为淀粉合成提供碳骨架[3]。
目前,研究者已经从黄芪[4]、水稻[5]、棉花[6]等植物中克隆得到UGPase。吴晓俊等[7]发现,不同生长时期黄芪毛状根中总多糖量与UGPase活性具有较强的相关性,较高的酶活性对应较高的多糖量;转化水稻OsUgp2基因的紫芝菌株胞内多糖与胞外多糖较之野生型对照菌株均有较明显的提高[8];在拟南芥中过表达棉花GhUGPase基因的转基因植株中可溶性糖量显著增加[6];转化紫穗槐AfUGPase基因的烟草中,细胞壁合成所需的基质多糖量显著增加[9];过表达兴安落叶松LgUGPase基因的拟南芥植株中葡萄糖、果糖蔗糖量均显著高于野生型[10]。这些研究表明:植物体内UGPase基因过表达,具有促进糖代谢及多糖合成与积累的作用。
党参CodonopsisRadix为桔梗科(Campanulaceae)植物党参Codonopsis pilosula (Franch.) Nannf、素花党参Codonopsis pilosula Nannf.var.modesta (Nannf.) L.T.Shen或川党参Codonopsis tangshen Oliv.的干燥根[11],为传统补益类中药,含有多糖、党参炔苷、党参苷、生物碱、萜类、挥发油等活性成分,且多糖量远高于其他组分[12]。党参多糖是目前研究最多的成分,具有增强机体免疫、调节血液循环、抗溃疡、抗衰老等生物学活性,亦可作为党参的质量评价标准[13]。开展党参多糖代谢研究、阐明党参多糖合成代谢途径,对党参多糖代谢调控、指导良种繁育具有重要的意义。党参的多糖代谢过程复杂[14],关于党参UGPase基因的克隆等研究至今未见有报道。为此,本实验针对党参多糖代谢途径中的关键酶基因尿苷二磷酸葡萄糖焦磷酸化酶CpUGPase基因开展研究。根据党参Codonopsis pilosula (Franch.) Nannf转录组文库CpUGPase基因序列[14],设计特异性引物,通过RT-PCR从党参中扩增得到CpUGPase基因,构建得原核表达载体并成功诱导其表达,以期为CpUGPase蛋白的功能研究奠定基础。
1 材料和试剂2013年10月,于党参采收期,自山西省晋城市陵川县党参GAP基地采集党参鲜根,无菌水洗净后灭菌刀片迅速切碎投入液氮速冻,之后存于-80 ℃用于总RNA提取。原植物由山西医科大学生药学教研室高建平教授鉴定为党参Codonopsis pilosula (Franch.) Nannf。
大肠杆菌菌株DH5α购自生工生物工程股份有限公司,克隆载体pMDTM18-T购自宝生物工程(大连)有限公司。表达宿主菌BL21(DE3)受赠于普健生物(武汉)科技有限公司,表达载体PET-28a购于武汉淼灵生物科技有限公司。
Trizol总RNA提取试剂购自康为世纪公司;反转录试剂盒PrimeScriptTM RT reagent Kit with gDNA Eraser、Ex taq酶、限制性内切酶BamH I和XhoI、T4 DNA连接酶购于宝生物工程(大连)有限公司;琼脂糖凝胶回收试剂盒购自Omega公司;X-Gal和IPTG购于生工生物工程(上海)股份有限公司;其他试剂均为国产分析纯。
2 方法 2.1 总RNA提取及CpUGPase基因全长cDNA的扩增按照试剂盒说明书,提取党参根总RNA并反转录合成cDNA。根据转录组中CpUGPase序列,利用primer premier 5.0和Oligo 7.37设计一对特异引物CpUGPaseF:5’-AATACAACACTTCCCCGT-3’,CpUGPaseR:5’-AGTAAAACTCTCCCTCCG-3’。将cDNA稀释10倍后作为模板进行PCR扩增,反应体系包括:TaKaRa Ex Taq 0.25 μL;10×Ex Taq Buffer 5 μL;dNTP Mixture 4 μL;Template 2 μL;CpUGPase-F 2.0 μL;CpUGPase-R 2.0 μL;灭菌ddH2O 34.75 μL。反应条件为95 ℃、5 min;94 ℃、1 min,55 ℃、1 min,72 ℃、2 min,35个循环;72 ℃延伸10 min。
2.2 CpUGPase基因克隆和测序PCR产物经1.2%琼脂糖凝胶电泳分离,切胶后,采用胶回收试剂盒对目的片段进行回收,并连接至pMD18-T载体上,转化至大肠杆菌DH5α感受态细胞,蓝白斑筛选,重组质粒pMD18-CpUGPase委托北京六合华大基因科技股份有限公司双向测序,确认核苷酸序列。
2.3 CpUGPase的生物信息学分析利用NCBI在线工具ORF finder http//www.ncbi.nlm.nih.gov/gorf/gorf.html查找CpUGPase的开放阅读框(open reading frame,ORF)。编码蛋白的理化性质预测采用ExPASy Proteomics Server提供的在线工具Protparam(http://www.expasy.ch/tools/protparam.html)进行分析。利用NCBI在线工具“blastp”(http://blast.ncbi.nlm.nih.gov/)对编码蛋白保守结构域进行分析。利用糖基化位点在线预测软件(http://www.cbs.dtu.dk/services/YinOYang/,http://www.cbs.dtu.dk/services/NetNGlyc/)对CpUGPase编码的氨基酸糖基化位点进行预测分析。利用在线软件TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测蛋白的跨膜区。利用在线工具SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)预测信号肽。应用在线工具WoLFPSORT进行亚细胞定位预测。氨基酸序列相似性通过NCBI(http://www.ncbi.nlm.nih.gov/)的蛋白质序列数据库进行blast,并利用DNAMAN6.0软件进行氨基酸多重序列比对。通过MEGA5.1重建Neighbor-joining系统进化树,设置bootstrap=1 000。
2.4 CpUGPase基因原核表达载体的构建在CpUGPase基因ORF两侧添加酶切位点,设计带有酶切位点的引物CpUGPase(BamH I)F:5’-CGCGGATCCATGGCTGCTGCCGATA-3’,CpUGPase(XhoI)R:5’-CCGCTCGAGTTAGATATCCTCGGG-ACCA-3’,以测序正确的、含有目的基因的重组质粒为模板进行PCR扩增。将扩增得到的目的基因切胶回收后与PET-28a (+)分别进行BamHI和XhoI双酶切,琼脂糖电泳回收。按1:4物质的量比(载体:目的基因)过夜连接,转化至DH5α感受态细胞,涂布于含100 mg/L卡那霉素的LB固体培养基上,挑取阳性克隆,摇菌提取质粒并酶切鉴定,获得重组质粒PET-28a-CpUGPase。
2.5 PET-28a-CpUGPase在大肠杆菌中的表达及表达条件的优化分别转化PET-28a-CpUGPase和PET-28a至大肠杆菌BL21(DE3)感受态细胞,挑取单菌落接种于3 mL含100 mg/L卡那霉素的LB液体培养基中,37 ℃、180 r/min振荡培养12 h。然后以1:100的比例接种到含100 mg/L卡那霉素的LB液体培养基中,37 ℃、180 r/min振荡培养,待A600为0.6时,加IPTG至终浓度为0.2 mmol/L,30 ℃、140 r/min诱导培养5 h。诱导完成后取1 mL菌液4 000 r/min离心,弃上清液,加入100 μL SDS上样缓冲液,充分悬浮,煮沸10 min,自然冷却至室温后进行SDS-PAGE(5%浓缩胶,12%分离胶)分析,以确定重组蛋白是否表达。
在此基础上,通过添加不同浓度的IPTG(终浓度分别为0、0.1、0.2、0.4、0.6、0.8、1.0 mmol/L)、诱导处理不同时间(0、1、2、3、5、7、9 h)、在不同的温度(25、30、37 ℃)下诱导,进行诱导条件的优化。
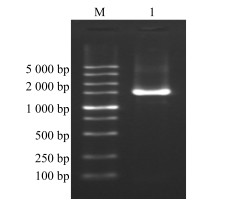
3 结果与分析 3.1 党参CpUGPase cDNA序列的克隆以党参cDNA为模板,扩增得到1 600 bp左右的片段(图 1),通过TA克隆获得重组质粒pMD18-CpUGPase,测序结果表明所扩增序列与前期课题组已提交NCBI的登录号为AJA91184.1的cDNA序列一致。
|
M-Marker 1-PCR扩增结果 M-Marker 1-PCR result 图 1 CpUGPase基因PCR结果 Fig.1 PCR result of CpUGPase gene |
3.2 CpUGPase基因的生物信息学分析
克隆得CpUGPase基因cDNA全长为1 612 bp,含完整的开放阅读框,长度为1 413 bp,编码470个氨基酸。相对分子质量为51 402 a;理论等电点(pI)为5.99,带正电残基数(Arg+Lys)为50,负电残基数(Asp+Glu)为55。该蛋白的不稳定系数为25.94,表明该蛋白质稳定。
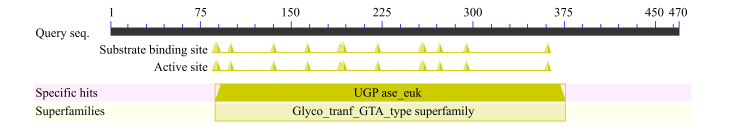
NCBI blastp分析结果显示,CpUGPase蛋白具有保守的UGPase_euk的催化位点,属于A型糖基转移酶家族(图 2),进一步证实所克隆序列属于UGPase基因家族。
|
图 2 CpUGPase蛋白保守区域分析 Fig.2 Prediction of conserved domains of CpUGPase protein |
对CpUGPase糖基化位点预测分析结果显示,该蛋白在162~164位氨基酸(N-Q-S)处存在N连接糖基化位点,在6位(Tys)、35位(Ser)、169位(Tyr)、371位(Ser)、377位(Ser)、426位(Ser)处存在O连接糖基化位点。包含植物UGPase中保守的第356~361位氨基酸残基(RFLPVK)与保守氨基酸位点257位(Lys)、323位(Lys)、361位(Lys)、402位(Lys)、403位(Lys)。同时在93~96位氨基酸(T-M-G-C)处存在核苷酸结合环(图 3)。

|
画直线部分为N连接的糖基化位点,画框线部分为O连接的糖基化位点,画双直线部为保守氨基酸残基,阴影为保守的Lys残基,画波浪线部分为核苷酸结合环 N linked glycosylation site was marked with a straight line, O-linked glycosylation sites was painted with red frame, the double line part was conservative amino acid residues; yellow shade was conservative Lys residues, wavy line part was nucleotide binding loop 图 3 CpUGPase蛋白的氨基酸序列 Fig.3 Amino acid sequence of CpUGPase protein |
在线工具WoLFPSORT亚细胞定位分析结果显示,细胞质定位系数为9,叶绿体定位系数为3,细胞核定位系数为1,说明CpUGPase蛋白最有可能位于细胞质中。TMHMM Server v.2.0跨膜结构预测结果表明,CpUGPase的整个氨基酸序列中,未发现跨膜结构或膜结合区域,推测其属于膜外蛋白。SignalP 4.1 Server信号肽预测结果显示,CpUGPase平均S值0.110、C值0.115、Y值0.111、最大S值0.110,均小于0.45,表明CpUGPase蛋白序列中不存在已知的信号肽,预示该蛋白不可能在细胞中发生移动,与跨膜区分析结果相一致。
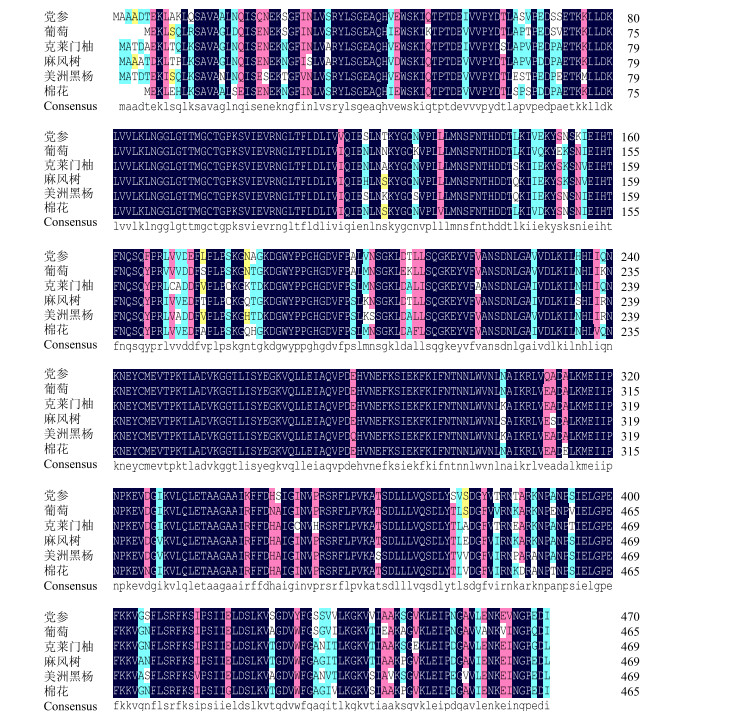
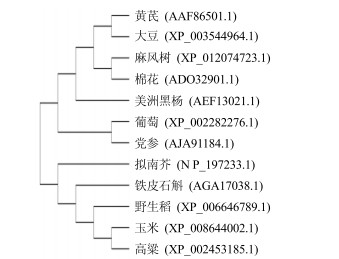
利用DNAMAN 6.0软件对CpUGPase和其他物种的UGPase进行氨基酸序列的多重比对分析,结果表明CpUGPase与其他物种的UGPase均有较高的相似度,其中与葡萄UGPase的相似度最高,达88%(图 4)。从NCBI上搜索到其他11个物种的UGPase蛋白序列,利用MEGA 5.1软件重建系统发育树(图 5)。结果显示党参CpUGPase与葡萄的亲缘关系相对较近,可聚为一支,而与同为药用植物的黄芪亲缘关系相对较远。
|
图 4 CpUGPase与其他植物UGPase序列比对结果 Fig.4 Multiple sequence alignment of CpUGPase amino acid sequence with UGPase sequence in other plants |
|
图 5 CpUGPase蛋白与其他植物UGPase蛋白的系统进化分析 Fig.5 Phylogentic relationship of CpUGPase and UGPase proteins in other plants |
3.3 CpUGPase基因原核表达载体的构建
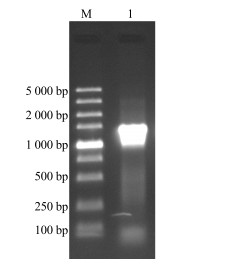
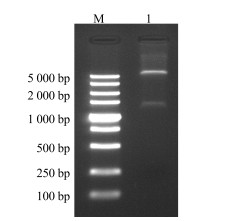
用带有酶切位点的引物进行PCR扩增,扩增产物经电泳检测表明与预期大小一致(图 6)。将目的片段切胶回收纯化后用BamH I和XhoI双酶切,连接到相同酶切回收后的PET-28a上,获得PET-28a-CpUGPase原核表达载体,转化大肠杆菌DH5α。重组质粒酶切鉴定正确后送测序(图 7),结果表明目的基因与原序列一致,说明重组质粒PET-28a-CpUGPase构建成功。
|
M-Marker 1-CpUGPase ORF PCR扩增结果 M-Marker 1-PCR result of CpUGPase ORF 图 6 CpUGPase ORF PCR扩增结果 Fig.6 PCR amplification of CpUGPase ORF |
|
M-Marker 1-BamH I和Xho I双酶切结果 M-Marker 1-double digestion result by BamH I and Xho I 图 7 PET-28a-CpUGPase质粒的双酶切结果 Fig.7 Double digestion of PET-28a-CpUGPase plasmid |
3.4 PET-28a-CpUGPase在大肠杆菌中的诱导表达及条件优化
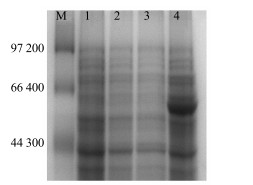
将PET-28a-CpUGPase转化大肠杆菌BL21(DE3)后进行IPTG诱导表达。结果表明,与对照相比,PET-28a-CpUGPase工程菌经IPTG诱导后,在相对分子质量54 900(含融合蛋白)左右有一条蛋白条带(图 8),表明重组质粒PET-28a-CpUGPase在大肠杆菌BL21(DE3)中诱导表达了CpUGPase蛋白。
|
M-Marker 1-含PET-28a的菌未诱导2-含PET-28a的菌诱导3-含PET-28a-CpUGPase的菌未诱导4-含PET-28a-CpUGPase的菌诱导 M-Marker 1-PET-28a-carrying host cells non induced 2-PET-28a-carrying host cells induced 3-PET-28a-CpUGPase carrying host cells non induced 4-PET-28a-CpUGPase carrying host cells induced 图 8 PET-28a-CpUGPase在E.coli BL21 (DE3)中表达的SDS-PAGE分析 Fig.8 SDS-PAGE analysis of expression products of E.coliBL21 (DE3)/PET-28a-CpUGPase |
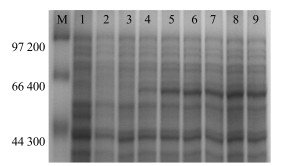
设置的所有IPTG浓度均能有效诱导CpUGPase重组蛋白的表达,IPTG终浓度为0.4 mmol/L时重组蛋白表达量已达到最大,从节约成本的角度选择诱导剂IPTG的终浓度为0.4 mmol/L,见图 9。
|
M-Marker 1-含PET-28a的菌未诱导2-含PET-28a的菌诱导(IPTG浓度为0.2 mmol/L)3~9-含PET-28a-CpUGPase的菌IPTG诱导浓度分别为0、0.1、0.2、0.4、0.6、0.8、1.0 mmol·L-1 M-Marker 1-PET-28a-carrying host cells non induced 2-PET-28a-carrying host cells induced with 0.2 mmol/L IPTG 3—9-PET-28a-CpUGPase carrying host cells induced with 0, 0.1, 0.2, 0.4, 0.6, 0.8, 1.0 mmol·L-1 IPTG 图 9 不同诱导浓度IPTG下PET-28a-CpUGPase基因表达的SDS-PAGE分析 Fig.9 SDS-PAGE analysis on expression products of E.coli BL21 (DE3)/PET-28a-CpUGPase at different concentration |
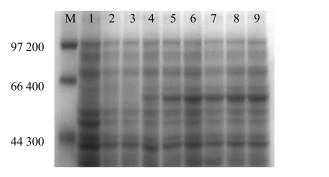
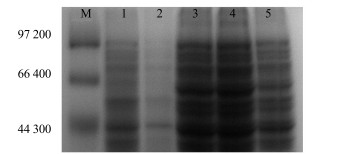
加入IPTG 1 h后CpUGPase重组蛋白即有表达,在3 h后表达量达到最大,之后其表达量占宿主菌总蛋白的比例基本保持一致,因此可选择其最佳诱导时间为3 h,见图 10。诱导温度对CpUGPase重组蛋白的表达量有一定的影响,在30 ℃时该蛋白表达量最大,见图 11。
|
M-Marker 1-含PET-28a的菌未诱导2-含PET-28a的菌诱导5 h 3~9-含PET-28a-CpUGPase的菌分别诱导0~3、5、7、9 h M-Marker 1-PET-28a-carrying host cells non induced 2-PET-28a-carrying host cells induced for 5 h 3—9-PET-28a-CpUGPase carrying host cells induced for 0—3, 5, 7, 9 h 图 10 不同诱导时间下PET-28a-CpUGPase表达的SDS-PAGE分析 Fig.10 SDS-PAGE analysis of expression products of E.coli BL21 (DE3)/PET-28a-CpUGPase at different time |
|
M-Marker 1-含PET-28a的菌未诱导2-含PET-28a的菌37 ℃下诱导3~5-含PET-28a-CpUGPase的菌IPTG诱导温度分别为25、30、37 ℃ M-Marker 1-PET-28a-carrying host cells non induced 2-PET-28a-carrying host cells induced at 30 ℃ 3—5-PET-28a-CpUGPase carrying host cells induced at 25, 30, 37 ℃ 图 11 不同诱导温度下PET-28a-CpUGPase表达的SDS-PAGE分析 Fig.11 SDS-PAGE analysis of expression products of E.coli BL21 (DE3)/PET-28a-CpUGPase at different temperatures |
4 讨论
已从党参中分离出多个党参多糖组分,这些党参多糖主要由单糖和单糖衍生物组成,其中单糖主要是戊糖和己糖,如戊糖的阿拉伯糖、木糖,己糖的葡萄糖、果糖、半乳糖等;单糖衍生物主要是糖醇如甘露醇,糖酸如半乳糖醛酸等[15-17]。Feingold等[18]报道UGPase催化生成的UDP-葡萄糖是UDP-阿拉伯糖、UDP-木糖、UDP-半乳糖、UDP-葡萄糖醛酸等次生活化单糖的前体,而这些活化单糖是合成党参多糖的重要底物,因此CpUGPase是党参多糖生物合成过程中重要的酶,克隆和鉴定CpUGPase基因对党参多糖代谢途径研究具有重要意义。
早期的研究者认为植物中只有1种UGPase基因,但后来的研究发现在水稻[5]、马铃薯[19-20]、拟南芥[21]等中存在两种相似性很高的UGPase。水稻基因组中2个同源UGPase基因UGP1和UGP2核苷酸序列相似性为81%,氨基酸序列相似性为88%,这两种基因在根、茎、叶各组织均有表达,而UGP2则比UGP1表达量低很多。马铃薯中的2种UGPase基因分别为UgpA和UgpB,这2种序列中有27个碱基不同,并导致了5个氨基酸的差异。拟南芥基因组中的2个UGPase基因UGP1和UGP2分别由469和470个氨基酸编码,它们的主要区别是N端、C端的结构略有不同。综上所述,植物中可能存在2种UGPase基因,但至今还没有对UGPase基因家族作专门研究或对基因序列进行系统分析及归类的文献报道。
在党参转录组中,仅检测到1个CpUGPase[14]。本研究从党参中成功扩增出CpUGPase基因的cDNA序列,并对其编码的氨基酸序列的结构和功能进行了生物信息学分析。已有的研究表明,植物之间的UGPase有超过80%的相似度[22],本实验获得的CpUGPase蛋白与已知植物的UGPase氨基酸序列均有较高的相似度,其中与葡萄UGPase的相似度达88%。CpUGPase含有植物组织中UGPase所具有的糖基化位点:162~164位氨基酸(N-Q-S)处的N连接糖基化位点和6位(Tys)、35位(Ser)、169位(Tyr)、371位(Ser)、377位(Ser)、426位(Ser)处的O连接糖基化位点,可能与膜结合活性有关[2, 23]。在93~96位氨基酸(T-M-G-C)处存在核苷酸结合环,可能与核苷酸结合活性相关,96位处的Cys为关键氨基酸,可能与PPi结合有关[6, 24]。另外含有活性中心的保守Lys残基:257位(Lys)、323位(Lys)、361位(Lys)、402位(Lys)、403位(Lys),这些氨基酸对于维持CpUGPase蛋白质的空间结构,使其能够与底物结合并发挥催化活性起到了关键作用[25]。
本实验还成功构建了PET-28a-CpUGPase原核表达载体,实现了CpUGPase基因的异源表达,同时对表达条件进行了优化,确定了PET-28a-CpUGPase重组质粒的最佳表达条件为30 ℃下0.4 mmol/L IPTG诱导3 h。CpUGPase的成功表达为进一步研究CpUGPase蛋白结构和功能奠定了实验基础,也为阐明党参多糖代谢途径积累了资料。
党参多糖的研究虽然起步较早,但这些研究主要集中在含量测定及药理作用上,而极少涉及党参多糖的合成代谢机制。因此,有关党参多糖的研究还有待更深入的开展工作,在今后的研究中,可以将化学与分子生物学技术相结合,研究在党参中过表达UGPase对党参多糖的影响,为党参多糖的生物合成及调控机制研究提供更多的信息。
| [1] | |
| [2] | Kleczkowski L A. Glucose activation and metabolism through UDP-glucose pyrophosphorylase in plants[J]. Phytochemistry , 1994, 37 (6) :1507–1515. DOI:10.1016/S0031-9422(00)89568-0 |
| [3] | Ciereszko I. Sucrose and light activation of a cold-inducible UDP-glucose pyrophosphorylase gene via a hexokinase-independent and abscisic acid-insensitive pathway in Arabidopsis[J]. Biochem J , 2001, 354 (1) :67–72. DOI:10.1042/bj3540067 |
| [4] | Wu X J, Du M, Weng Y. UGPase of Astragalus membranaceus: cDNA cloning, analyzing and expressing in Escherichia coli[J]. Acta Bot Sin , 2002, 44 (6) :689–693. |
| [5] | Abe T, Niiyama H, Sasahara T. Cloning of cDNA for UDP-glucose pyrophosphorylase and the expression of mRNA in rice endosperm[J]. Theoret Appl Genet , 2002, 105 (2/3) :216–221. |
| [6] | Wang Q, Zhang X, Li F. Identification of a UDP-glucose pyrophosphorylase from cotton (Gossypium hirsutum L.) involved in cellulose biosynthesis in Arabidopsis thaliana[J]. Plant Cell Rep , 2011, 30 (7) :1303–1312. DOI:10.1007/s00299-011-1042-x |
| [7] | 吴晓俊, 刘涤, 胡之璧. 黄芪毛状根中尿苷二磷酸葡萄糖焦磷酸化酶活性与多糖含量关系的研究[J]. 上海中医药大学学报 , 2000, 14 (2) :53–55. |
| [8] | 张帆, 钟威, 穆虹, 等. 过量表达OsUgp2基因提高紫芝多糖含量[J]. 菌物学报 , 2011, 30 (3) :442–452. |
| [9] | 刘文哲.紫穗槐UGPase和4CL基因的克隆及在植物中的转化与表达[D].大连:大连理工大学, 2002. http://cdmd.cnki.com.cn/article/cdmd-10141-2003110411.htm |
| [10] | 李宁宁.兴安落叶松UDP-葡萄糖焦磷酸化酶和UDP-葡萄糖脱氢酶基因的克隆及特性分析[D].呼和浩特:内蒙古大学, 2014. http://cdmd.cnki.com.cn/article/cdmd-10126-1014265586.htm |
| [11] | 中国药典[S].一部. 2015. |
| [12] | 马红燕, 高宏伟, 刘素波. 潞党参多糖的提取与精制工艺研究[J]. 长治医学院学报 , 2012, 26 (5) :330–332. |
| [13] | 李黎星, 康杰芳. 中药党参的研究进展[J]. 现代生物医学进展 , 2009, 9 (12) :2371–2373. |
| [14] | Gao J P. Transcriptome sequencing of Codonopsis pilosula and identification of candidate genes involved in polysaccharide biosynthesis[J]. PLoS One , 2015, 10 (2) :3833. |
| [15] | Yang C X. Structural characterization and antitumor activity of a pectic polysaccharide from Codonopsis pilosula[J]. Carbohydr Polym , 2013, 98 (1) :886–895. DOI:10.1016/j.carbpol.2013.06.079 |
| [16] | Zhang Y J, Liang Z Y, Zhao W. Separation, purification and compositional analysis of water soluble polysaccharide CPPS_3 from Codonpsis pilosula[J]. J Chin Pharm Sci , 2005, 40 (14) :1107–1109. |
| [17] | 杨丰榕, 李卓敏, 高建平. 党参多糖分离鉴定及体外抗肿瘤活性的研究[J]. 时珍国医国药 , 2011, 22 (12) :2876–2878. |
| [18] | Feingold D S, Barber G A. Methods in Plant Biochemistry[M].(Vol. 2). London: Academic Press, 1990 . |
| [19] | Sowokinos J R C, Thomas M M, Burrell. Pyrophosphorylases in potato. V. Allelic polymorphism of UDP-glucose pyrophosphorylase in potato cultivars and its association with tuber resistance to sweetening in the cold[J]. Plant Physiol , 1997, 113 (2) :511–517. DOI:10.1104/pp.113.2.511 |
| [20] | Sowokinos, Vigdorovich V, Abrahamsen M. Molecular cloning and sequence variation of UDP-glucose pyrophosphorylase cDNAs from potatoes sensitive and resistant to cold sweetening[J]. J Plant Physiol , 2004, 161 (8) :947–955. DOI:10.1016/j.jplph.2004.04.006 |
| [21] | Meng M, Wilczynska M, Kleczkowsk L A i. Molecular and kinetic characterization of two UDP-glucose pyrophosphorylases, products of distinct genes, from Arabidopsis[J]. Biochim Biophys Acta , 2008, 1784 (6) :967–972. DOI:10.1016/j.bbapap.2008.02.021 |
| [22] | Meng M. Domain-specific determinants of catalysis/ substrate binding and the oligomerization status of barley UDP-glucose pyrophosphorylase[J]. Biochim Biophys Acta , 2009, 1794 (12) :1734–1742. DOI:10.1016/j.bbapap.2009.08.009 |
| [23] | Lance W, Whelan S A, Hart G W. O-GlcNAc: a regulatory post-translational modification[J]. Biochem Biophysical Res Commun , 2003, 302 (3) :435–441. DOI:10.1016/S0006-291X(03)00175-X |
| [24] | Martz F, Wilczynska M, Kleczkowski L A. Oligomerization status, with the monomer as active species, defines catalytic efficiency of UDP-glucose pyrophosphorylase[J]. Biochem J , 2002, 367 (1) :295–300. DOI:10.1042/bj20020772 |
| [25] | Katsube T, Kazuta Y, Tanizawa K. Expression in Escherichia coli of UDP-glucose pyrophosphorylase cDNA from potato tuber and functional assessment of the five lysyl residues located at the substrate-binding site[J]. Biochemistry , 1991, 30 (35) :8546–8551. DOI:10.1021/bi00099a008 |
 2016, Vol. 47
2016, Vol. 47


