川芎Ligusticum chuanxiong Hort. 为四川省著名的道地药材,其主要活性成分为阿魏酸[1]。目前已知阿魏酸的生源途径起源于苯丙氨酸,经肉桂酸、对香豆酸、咖啡酸,最后由咖啡酸-3-O-甲基转移酶(caffeic acid-3-O-methyltransferase,COMT)以咖啡酸和以S-腺苷甲硫氨酸为底物催化合成阿魏酸。COMT可能是阿魏酸生物合成途径中的一个关键酶[2-4]。本实验室在前期研究中已获得川芎根茎与叶的转录组数据,从中发现并克隆了川芎COMT(LCCOMT)基因(GenBank登录号AKZ17612.1)[5]。进一步将LCCOMT基因转化大肠杆菌,并获得有生物活性的重组蛋白[6]。本实验利用同源建模方法构建LCCOMT的三维模型,并结合定点突变技术鉴定其可能的关键氨基酸残基,这为LCCOMT的结构功能研究及应用奠定了理论基础。
1 材料LCCOMT-pET28重组大肠杆菌表达载体由本实验室构建并保存。大肠杆菌BL21由本实验室保存。限制性内切酶、Prime star DNA聚合酶、T4 DNA连接酶、均购自TaKaRa公司。RNA提取试剂盒Omega Plant RNA Kit、质粒小提试剂盒Omega Plasmid Mini Kit I、胶回收试剂盒Gel Extraction Kit为OMEGA公司产品。异丙基硫代半乳糖苷(IPTG)购自MEKER公司,卡那霉素、S-腺苷甲硫氨酸(SAM)、咖啡酸对照品(批号201317)均购自生工生物工程有限公司,二硫苏糖醇(DTT)购自BIOSHARP公司,阿魏酸对照品(批号110773-201313)购自成都市药品检验所,质量分数均大于98%。
2 方法 2.1 序列比对和同源建模使用NCBI的Protein Blast工具,搜寻Protein Database Bank(PDB)数据库与LCCOMT同源性和一致性较高的蛋白质为模板。利用Discovery Studio(DS)2.5软件平台完成二级结构预测和模板序列的序列比对。通过DS的MODELER模块进行同源建模,获得LCCOMT的初始模型。利用DS的Proteins Superimposing模块将LCCOMT的始模型和模板的三维结构进行叠合比较,根据均方根偏差(root mean square deviation,RMSD)分析建模与模板的相似度。
2.2 初始模型的结构优化利用DS 2.5软件的能量最小化和分子动力学模拟模块对初始模型进行结构优化。步骤:(1)Minimization:将模型溶解于生理盐水环境,首先采用1 000步最陡下降法对体系进行优化;随后,采用5 000步共轭梯度法对体系进行再次优化;(2)Heating:设置温度梯度为50 K到300 K,进行200 ps的动态模拟;(3)Equilibration:在T=300 K条件下进行200 ps的分子动力学模拟;(5)Production:同样在热力学温度条件下进行200 ps的分子动力学计算,每隔500 fs收集1个分子构象保存,选取能量最低的构象为最终的模型结构。COMT的立体化学可靠性通过PROCHECK计算的Psi/Phi Ramachandran图进行评估[7-8];蛋白质侧链氨基酸残基的兼容性通过Verify-3D进行检测[9]。
2.3 分子对接咖啡酸(Compound ID:689043;MF:C9H8O4)的三维结构来源于NCBI的Pubchem数据库。利用DS软件的CDOKER模块对接LCCOMT与咖啡酸分子,最后对咖啡酸-LCCOMT复合物进行动力学优化。
2.4 LCCOMT基因的定点突变LCCOMT基因的定点突变采用一步法[7],以pET28/LCCOMT野生型重组载体为模板,根据His268所对应的核苷酸设计突变引物(表 1)。
|
|
表 1 突变引物及其序列 Table 1 Mutation primers and their sequences |
以含有LCCOMT基因的重组质粒DNA为模板,用引物1和引物2进行PCR扩增,获得含有突变碱基的LCCOMT基因。PCR反应体系总量为50 μL,在美国Applied Biosystems公司Veriti® 96孔快速PCR仪上进行。PCR反应条件:95 ℃,4 min;95 ℃,1 min;Tm,1 min;68 ℃,6 min;18 个循环;72 ℃,10 min;4 ℃保存。其中Asn突变的Tm为59 ℃,Gln突变的Tm为61 ℃。
DNA片段的回收、酶切、克隆、转化方法参见文献报道[6]及试剂盒说明;扩增获得H268N与H268Q 2种突变型基因,构建突变型原核表达载体,导入BL21菌株中,转化完成后取100 μL菌液涂布于含有卡那霉素(Kan)的LB平板上进行筛选,分别获得突变型菌株。每个突变型菌株挑取3个菌落提取质粒,分别进行酶切和PCR鉴定,由成都擎科梓熙生物技术有限公司测序,验证突变的正确性。
2.5 突变酶的表达、纯化及活性检测突变酶的诱导表达、纯化参见文献方法[6]。重组大肠杆菌BL21在含Kan的LB培养基培养至A600值大于0.6时,加入IPTG使终浓度为1 mmol/L,过夜诱导表达(37 ℃,200 r/min)。菌液在超声破碎和冻融后经过NI2+亲和柱后得到纯化蛋白质。
突变酶的活性检测方法参见文献方法[6]。检测反应体系包括100 mmol/L磷酸钾缓冲液(pH 7.0),500 μmol/L咖啡酸,1 mmol/L SAM,1.5 mmol/L MgCl2,1 mmol/L DTT与1 000 μg纯化蛋白,总体积为10 mL,37 ℃下反应2 h后加1 mL浓盐酸终止反应。用5 mL醋酸乙酯萃取3次得到反应萃取液并浓缩至1 mL左右。浓缩萃取液经过滤后HPLC检测,根据咖啡酸和阿魏酸积分后的峰面积来计算酶活性。
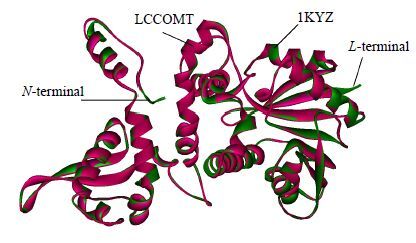
3 结果和分析 3.1 序列比对和同源建模BLAST的结果显示,LCCOMT的氨基酸序列与来源于紫花苜蓿的COMT(PDB ID:1KYZ_E)具有最高的同源性,其序列一致性(identity)为72.7%,相似度(similarity)为85.9%。因此,本实验选择1KYZ作为LCCOMT同源建模的模板。二级结构预测结果表明,LCCOMT含有17个α螺旋(48.9%),9个β折叠(11.3%)及27个无规卷曲(39.8%)(图 1)。Proteins Superimposing显示LCCOMT初始模型与1KYZ的RMSD值为0.042 5 nm,表明两者具有十分相似的三维框架(图 2)。

|
α-α-螺旋 β-β-折叠 c-无规卷曲 α-α-helix β-β-sheet c-randon coil 图 1 LCCOMT与1KYZ的序列比对 Fig.1 Sequence alignment of LCCOMT and 1KYZ |
|
绿色为模板1KYZ,红色为LCCOMT的三维模型 Green part is the structure of 1KYZ, red part is the structure of LCCOMT 图 2 LCCOMT初始模型与1KYZ的三维结构叠合 Fig.2 3D superimposition structure of LCCOMT initial model and 1KYZ |
3.2 同源模型的验证
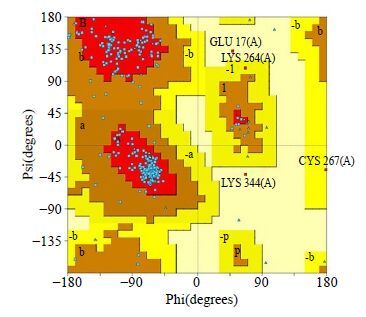
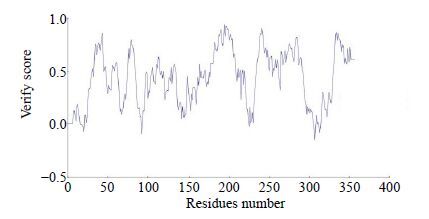
LCCOMT的初始模型经过动力学优化后使用Psi/Phi Ramachandran图分析模型主链和侧链的立体化学构型的合理性(图 3)。结果表明LCCOMT优化模型的绝大多数φ、ψ二面角均在合理范围内,其中核心区域占89.5%,允许区域占8.6%,仅有0.6%在不允许区域内。Verify-3D分析结果显示LCCOMT优化模型中有98.2%的侧链氨基酸残基得分在0.2以上(图 4),表明模型的侧链环境是合理的。以上结果均表明LCCOMT优化模型能够用于后续的对接研究。
|
图 3 LCCOMT优化模型的Psi/Phi Ramachandran图 Fig.3 Psi/Phi Ramachandran plot of LCCOMT optimized model |
|
图 4 LCCOMT优化模型的Verify-3D图 Fig.4 Verify-3D score of LCCOMT optimized model |
3.3 对接研究与活性位点分析及催化机制推测
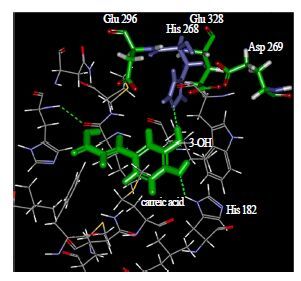
LCCOMT主要含有2个结构域,即蛋白二聚化结构域与依赖SAM的催化活性结构域[10-11]。蛋白二聚化结构域位于LCCOMT的N末端,包括了33~84位等多个氨基酸残基,该结构域在多个植物的甲基转移酶中均有出现,其主要功能是促进COMT单体二聚化[12]。SAM依赖的催化活性结构域主要由Leu126、Met129、Asn130、Lue135、Phe175、Met179、His182、Trp265、His268、Asp269、Glu296、Ile318、Met319、His322与Asn323等氨基酸残基组成,其主要功能是将SAM上的甲基转移到底物分子中。其中的部分氨基酸残基能够通过疏水作用与氢键来稳定咖啡酸分子在催化中心的立体位置,比如His182与咖啡酸的-OH之间的氢键。序列比对发现,LCCOMT含有氧甲基转移酶家族中高度保守的His268残基,该残基作为质子受体,在底物甲基化的过程中可能发挥了关键作用。分子对接结果发现,His268位于Glu296、Glu328与Asp269等3个酸性氨基酸残基组成的“酸性口袋”中,并能够与咖啡酸的3-OH之间形成氢键。推测咖啡酸的3-OH在酸性环境中容易发生断裂,释放质子,导致咖啡酸的3-O-离子(亲核基团),后者即与SAM的甲基发生甲基转移反应,形成阿魏酸(图 5)。
|
绿色小分子为咖啡酸;蓝色氨基酸残基为His268;绿色氨基酸残基分别Asp269、Glu296和Glu328 Green molecule is caffeic acid; blue amino acid residues is His268; green amino acid residues are Asp269, Glu296, and Glu328 respectively 图 5 咖啡酸与LCCOMT的结合位点 Fig.5 Binding sites of caffeic acid with LCCOMT |
3.4 突变酶的活性检测
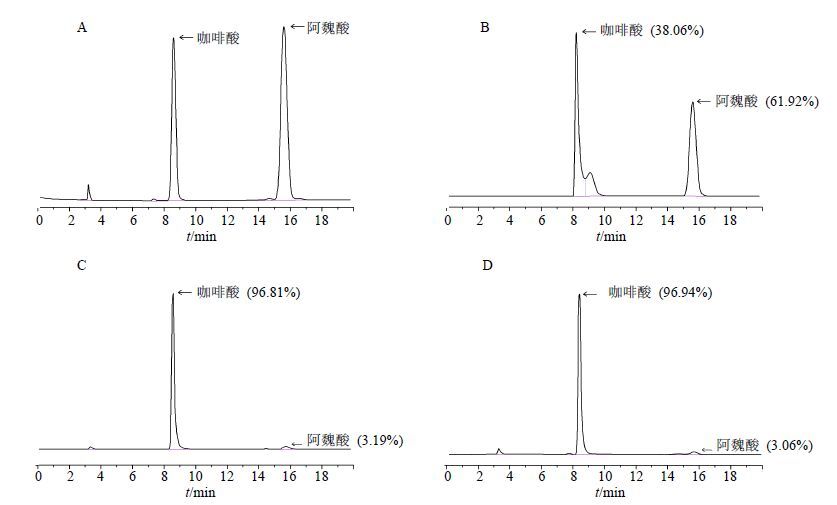
根据同源建模的结果,对该His268残基展开了定点突变的研究。活性检测结果如图 6所示,反应2 h后,野生型LCCOMT能够高效地转化咖啡酸形成阿魏酸,其催化活力为30.07 U/mg(图 6-B)。当His268分别突变为Asn与Gln后,催化活力仅为1.55 U/mg和1.484 U/mg(图 6-C与图 6-D)。相对于野生型LCCOMT,活性分别下降了94.85%和95.28%,这表明His268确为LCCOMT的关键氨基酸残基,其作为质子受体参与了LCCOMT的催化过程。
|
A为咖啡酸和阿魏酸对照品,B为野生型蛋白,C为H268N突变蛋白,D为H268Q突变蛋白;括号内为咖啡酸与阿魏酸的峰面积占比< A: standard caffeic acid and ferulic acid; B: wild-type protein; C: H268N mutant protein; D: H268Q mutant protein; The peak area ratio of caffeic acid and ferulic acid was in parentheses 图 6 LCCOMT的活性检测 Fig.6 Enzyme activity assay of LCCOMT |
4 讨论
药用植物的次生代谢产物是众多中药的天然来源。发掘次生代谢产物合成基因并研究其功能,将为阐明次生代谢产物生物合成的分子机制,进而开展代谢调控奠定基础。阿魏酸具有较强的抗氧化与去除氧自由基的作用[13],其作为治疗和预防神经退行性疾病、糖尿病和心血管疾病等药物的应用前景受到了广泛的关注。COMT为阿魏酸合成途径的末端酶,对其编码基因展开代谢调控极有可能提高植物中阿魏酸的量[14]。
本实验首先通过同源建模获得了LCCOMT的三维模型。LCCOMT的三维模型中含有2个重要的结构域,即蛋白二聚体结构域与催化结构域。分析苜蓿COMT的蛋白二聚化结构域发现,该结构域能够促进COMT单体的二聚化,提高COMT的稳定性[12]。分析不同植物来源氧甲基转移酶(OMT)的催化结构域,发现该区域中结合SAM的氨基酸残基保守性较强,结合底物的氨基酸残基保守性较差,分析这与不同氧甲基转移酶具有不同的底物特异性有关[14]。根据LCCOMT与咖啡酸分子对接的结果,推测His268是LCCOMT的关键氨基酸残基,该残基作为质子受体,能够诱导咖啡酸3-OH发生脱质子,形成亲核性较强的3-O-。定点突变实验进一步证实了His268确实在LCCOMT的催化过程中发挥了关键作用。Zubieta等也发现査尔酮-O-甲基转移酶和异黄酮-O-甲基转移酶的His突变为Leu、Asn、Gln等氨基酸残基后,其活性则彻底丧失,这表明His在氧甲基转移酶分子结构中发挥了重要作用[15]。本实验所获得的LCCOMT的三维模型及定点突变结果将为认识LCCOMT的结构与功能的相互关系,为固定化LCCOMT体外合成阿魏酸、LCCOMT的转基因奠定了理论基础。
| [1] | Ran X, Ma L, Peng C, et al. Ligusticum chuanxiong Hotr:a review of chemistry and pharmacology[J]. Pharm Biology , 2011, 49 (11) :1180–1189. DOI:10.3109/13880209.2011.576346 |
| [2] | Komél N, Lucas A, Karine R, et al. Identification of cholesteryl ester of ferulic acid in human plasma by mass spectrometry[J]. J Chromatogr A , 2013, 1301 :162–168. DOI:10.1016/j.chroma.2013.05.078 |
| [3] | Justin E L, Tomas V, Bettina H, et al. Methyl jasmonate induces an O-methyltransferase in barley[J]. Plant Cell Physiol , 1997, 389 (7) :851–862. |
| [4] | Noriko Y, Masako F M, Hiroaki O, et al. Molecular cloning and characterization of O-methyltransferases from the flower buds of Iris hollandica[J]. J Plant Physiol , 2008, 165 (4) :415–422. DOI:10.1016/j.jplph.2006.12.002 |
| [5] | Li J J, Zhang G, Yu J H, et al. Molecular cloning and characterization of caffeic acid 3-O-methyltransferase from the rhizome of Ligusticum chuanxiong[J]. Biotechnol Lett , 2015, 37 (11) :2295–2302. DOI:10.1007/s10529-015-1917-y |
| [6] | 廖海, 周嘉裕, 张赶, 等. 一种酶法合成阿魏酸的方法:中国, CN 104818300 A[P]. 2015-08-05. |
| [7] | Liu Z, Zhu Q, Li J, et al. Isolation, structure modeling and function characterization of a trypsin inhibitor from Cassia obtusifolia[J]. Biotechnol Lett , 2015, 37 (4) :863–869. DOI:10.1007/s10529-014-1744-6 |
| [8] | Laskowski R A, MacArthur M W, Moss D S, et al. PROCHECK-a program to check the stereochemical quality of protein structures[J]. J App Cryst , 1993, 26 (2) :283–291. DOI:10.1107/S0021889892009944 |
| [9] | Bowie J U, Lüthy R, Eisenberg D, et al. A method to identify protein sequences that fold into a known three-dimensional structure[J]. Science , 1991, 253 (5016) :164–170. DOI:10.1126/science.1853201 |
| [10] | Zubieta C, He X Z, Dixon R A, et al. Structures of two natural product methyltransferases reveal the basis for substrate specificity in plant O-methyltransferases[J]. Nat Struct Biol , 2001, 8 (3) :271–279. DOI:10.1038/85029 |
| [11] | Keller N P, Dischinger H C J, Bhatnagar D, et al. Purification of a 40-kilodalton methyltransferase active in the aflatoxin biosynthetic pathway[J]. Appl Environ Microbiol , 1993, 59 (2) :479–484. |
| [12] | Zubieta C, Kota P, Ferrer J L, et al. Structural basis for the modulation of lignin monomer methylation by caffeic acid/5-hydroxyferulic acid 3/5-O-methyltransferase[J]. Plant Cell , 2002, 14 (6) :1265–1277. DOI:10.1105/tpc.001412 |
| [13] | Kanski J, Aksenova M, Stoyanova A, et al. Ferulic acid antioxidant protection against hydroxyl and peroxyl radical oxidation in synaptosomal and neuronal cell culture systems in vitro:structure-activity studies[J]. J Nutr Biochem , 2002, 13 (5) :273–281. DOI:10.1016/S0955-2863(01)00215-7 |
| [14] | 李潞滨, 刘蕾, 何聪芬, 等. 木质素生物合成关键酶基因的研究进展[J]. 分子植物育种 , 2008, 5 (11) :45–51. |
| [15] | Kaganr M, Clarke S. Widespread occurrence of three sequence motifs in diverse S-adenosylmethionine-dependent methyltransferases suggests a common structure for these enzymes[J]. Arch Biochem Biophys , 1994, 310 (2) :417–427. DOI:10.1006/abbi.1994.1187 |
 2016, Vol. 47
2016, Vol. 47


