2. 呼吸疾病诊疗与新药研发河南省协同创新中心, 河南 郑州 450046
2. Collaborative Innovation Center for Respiratory Disease Diagnosis and Treatment & Chinese Medicine Development of Henan Province, Zhengzhou 450046, China
独行菜Lepidium apetalum Willd.为十字花科(Cruciferae)独行菜属Lepidium L.植物,其成熟种子为传统中药北葶苈子,具有泻肺平喘、利水消肿、强心之功效,含有强心苷、黄酮、三萜、甾醇、香豆素、生物碱、酚酸、挥发油等多种成分,对心血管、高血脂等疾病具有显著功效,具有降压、抗癌、细胞毒、抗氧化等药理活性[1]。独行菜强心苷类化学成分伊夫单苷[2]等,对其进行结构分析可知属于植物甾醇(五环内酯),目前,对植物甾醇和三萜类皂苷的生物合成途径已有初步认识,从生物合成来源分析,植物甾醇与三萜类似,在生物体内也是由鲨烯以不同方式环化而成,即通过甲羟戊酸途径(MVA)衍生而来[3]。
MVA途径存在于细胞质中,是植物萜类物质合成的主要途径。磷酸甲羟戊酸激酶(phosphomevalonate kinase,PMK)是GHMP激酶超家族中的一员[4],是萜类化合物生物合成MVA途径中的一个重要限速酶,在这个途径中有3个连续依赖ATP的限速反应,其中PMK催化第2个限速反应,其作用是将ATP上的γ磷酸基团转移到甲羟戊酸-5-磷酸(MVAP)上生成甲羟戊酸-5-二磷酸(MVAPP),是控制整个代谢途径的关键酶之一[5-6]。目前,关于植物中PMK基因的研究较少,杨恩泽等[7]首次从药用植物阳春砂中克隆了AvPMK基因,并使用生物信息学的方法初步分析了酶结构特点和功能。对于哺乳类动物的PMK基因研究的较多,如果PMK基因突变将可能致使哺乳动物产生甲羟戊酸缺乏症(导致动物夭折),或产生半乳糖激酶缺乏症(导致动物产生白内障)[8]。
在对本实验室前期获得的大量独行菜转录组数据的分析中,发现多个参与MVA途径的基因,本研究首次从独行菜中克隆了PMK基因(LaPMK)的cDNA序列,进行生物信息学分析和原核表达,为进一步研究PMK基因在独行菜萜类合成途径中的作用奠定了基础。
1 材料与方法 1.1 材料样品采自河南省伏牛山区,由河南中医学院董诚明教授和谢小龙博士鉴定独行菜Lepidium apetalum Willd.种子,种子采集后,种植于人工气候培养箱中,光照强度为3 000 lx,在16 h、23 ℃光照,8 h、20 ℃黑暗,70%相对湿度条件下培养。种子萌发,长出4片真叶后,移至培养罐,每个培养罐放置4颗幼苗,每隔3 d更换1次Hoagland营养液,培养60 d的独行菜幼苗,取根、茎、叶,采集后样品经液氮速冻,保存于−80 ℃超低温冰箱中备用。本研究所用的实验材料为独行菜幼苗叶片。
1.2 试剂植物总RNA提取试剂盒、质粒小提试剂盒、PCR产物纯化试剂盒购自北京天根生化科技公司;TransScript Ⅱ Reverse Transcriptase、大肠杆菌Escherichia coli DH5α感受态细胞和BL21感受态细胞购自北京全式金生物技术公司;Ex Taq酶、限制性核酸内切酶、T4 DNA连接酶、pMD19-T vector购自Takara公司;引物由北京三博远志生物技术公司合成。一般试剂均参照《分子克隆实验指南》配制,所使用化学试剂均为分析纯试剂。
1.3 RNA提取及cDNA合成取100 mg独行菜幼苗的叶片,在液氮中研磨,根据天根公司植物总RNA提取试剂盒说明书提取独行菜叶片的总RNA,用NanoDrop 2000和1%琼脂糖凝胶电泳检测RNA的量和完整性。用全式金公司的TransScript Ⅱ Reverse Transcriptase试剂盒反转录合成cDNA。
1.4 LaPMK基因克隆根据已获得的独行菜转录组数据,用Primer 5设计LaPMK基因的特异性引物,正向引物:LaPMK-Exp-F(5’-CGGAATTCATGGCTGTAGTTG-CT-3’,下划线部分为EcoRI酶切位点);反向引物:LaPMK-Exp-R(5’-CCCAAGCTTTTACTCGAGAC-GAAT3’,下划线部分为Hind Ⅲ酶切位点)。PCR反应体系为cDNA模板1 μL,LaPMK-Exp-F 1 μL,LaPMK-Exp-R 1 μL,10×Ex Taq buffer 5 μL,2.5 mmol/L dNTP 4 μL,Ex Taq酶0.25 μL,加水至50 μL。反应程序为95 ℃预变性2 min,然后进行30个循环:95 ℃变性10 s,55 ℃退火15 s,72 ℃延伸1 min 40 s,最后72 ℃延伸8 min。PCR产物用1%琼脂糖凝胶电泳检测分析,用PCR产物纯化试剂盒纯化后与pMD19-T vector连接,转化大肠杆菌DH5α感受态细胞,在氨苄抗性平板上进行筛选,经菌液PCR和电泳检测后,挑选阳性克隆送北京三博公司测序。
1.5 LaPMK基因的生物信息学分析LaPMK基因编码的蛋白质使用ExPASy Protemics Server提供的在线工具ProtParam(http://web.expasy.org/protparam/)预测其相对分子质量与理论等电点(PI),使用InterPro Scan(http://www.ebi.ac.uk/interpro/)进行蛋白的保守结构域预测,使用SWISS-MODEL(http://swissmodel.expasy.org/)进行三维同源建模,使用TargetP 1.1Server(http://www.cbs.dtu.dk/services/TargetP/)分析蛋白质亚细胞定位,SignalP 4.1 server( http://www.cbs.dtu.dk/services/SignalP/)分析蛋白质信号肽,使用TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)进行跨膜区预测。
将LaPMK基因编码蛋白质的氨基酸序列提交到美国国立生物技术信息中心NCBI的蛋白质序列数据库进行BLASTP搜索相似序列(http://blast.ncbi.nlm.nih.gov/Blast.cgi),从搜索结果中选取与LaPMK基因编码的蛋白质相似性较高的其他植物的蛋白质序列作为参考(包括模式植物拟南芥、水稻、玉米,药用植物丹参、三七、长春花等植物),使用MEGA4软件的相邻连接法(neighbor-joining)构建系统进化树[9]。
1.6 LaPMK原核表达载体的构建与诱导表达用限制性内切酶EcoRI和HindIII分别对原核表达载体pET-32a(+)和测序正确的pMD19-T-LaPMK质粒进行双酶切,琼脂糖凝胶电泳检测后,切胶回收载体片段和目的基因片段,T4 DNA连接酶16 ℃连接过夜。将连接产物转化E.coli BL21(DE3)感受态细胞,在氨苄抗性平板上进行筛选,挑取单克隆,经菌液PCR和双酶切鉴定后送北京三博公司测序。将测序正确的单克隆,接种于含氨苄青霉素的LB液体培养基中,37 ℃培养过夜后,按照1∶100比例稀释到含氨苄青霉素的LB液体培养基中,220 r/min振荡培养至对数生长期[(600 nm吸光度(A600)至0.6],在0.4 mmol/L IPTG,28 ℃,150 r/min条件下培养8 h,诱导独行菜LaPMK重组蛋白的表达。诱导完成后,用SDS-PAGE检测重组蛋白的表达。
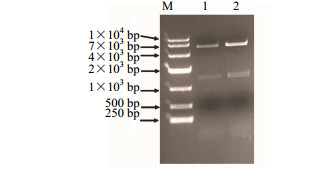
2 结果与分析 2.1 LaPMK的基因克隆通过分析本实验室前期获得的独行菜转录组数据,发现多个参与MVA途径的基因,其中有一个注释为PMK的转录本,长度为1 925 bp,利用NCBI ORF Finder分析其含有一个完整的ORF,根据该基因开放阅读框(ORF)序列设计特异性引物LaPMK-Exp-F和LaPMK-Exp-R,以独行菜叶片总RNA反转录得到的cDNA为模板,PCR扩增得到1条1 500 bp左右的片段,电泳结果如图 1所示,与预期大小相符,进行测序后得到独行菜LaPMK基因的编码序列,大小为1 518 bp,编码505个氨基酸(图 2),序列信息已经提交到NCBI Genbank,登录号为KT004541。
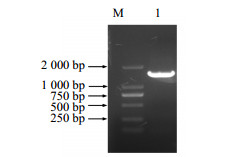
|
M-Marker 1-LaPMK基因PCR扩增产物 M-Marker 1-PCR product of LaPMK gene 图 1 PCR扩增LaPMK基因 Fig.1 PCR amplification of LaPMK gene |

|
黑色下划线:ATP结合位点 ATP binding sites are underlined 图 2 LaPMK基因cDNA编码区序列及氨基酸序列 Fig.2 Coding sequence and amino acid sequence of LaPMK gene cDNA |
2.2 LaPMK基因编码蛋白的生物信息学分析
LaPMK基因预测编码505个氨基酸(图 2),利用ExPASy Proteomics Server的在线工具Protparam对其编码蛋白的理化性质进行预测分析。推测分子式为C2411H3840N650O742S20,相对分子质量54 450,pⅠ:5.49;正电残基(Arg+Lys):44,负电残基(Asp+Glu):56。不稳定指数为33.13,说明LaPMK蛋白较稳定,亲水性总平均系数为−0.073,说明LaPMK蛋白为亲水性蛋白。
利用InterPro Protein进行LaPMK蛋白的保守结构域预测(图 3),结果显示LaPMK蛋白含有5种保守结构域,分别在氨基酸序列的1~495位是磷酸甲羟戊酸激酶(IPR016005)、2~259位是核糖体蛋白质-5S区域(2类型折叠,IPR020568)、2~109和157~257位是核糖体蛋白质-5S区域(2类型折叠小组)(IPR014721)、179~250位是GHMP激酶N端区域(IPR006204)、397~467、295~350与391~484位是GHMP激酶C端区域(IPR003750),含有GHMP激酶家族特异的N末端和C末端的保守结构域。
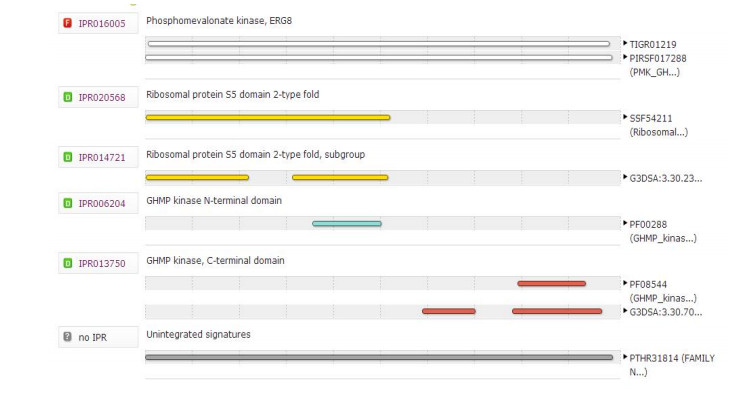
|
图 3 LaPMK基因的编码蛋白质保守结构域预测 Fig.3 Prediction of conserved domain of LaPMK protein encoding by LaPMK gene |
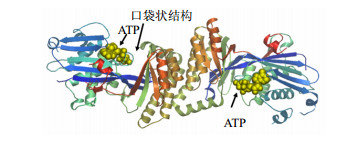
SignalP 4.1 server预测(图 4),LaPMK蛋白不含信号肽,为非分泌蛋白,TargetP 1.1 server预测,显示LaPMK蛋白可能定位于细胞质,TMHMM server预测该蛋白不含跨膜域(图 5)。用PredictProtein对LaPMK蛋白的二级结构进行预测,结果显示在该蛋白中α-螺旋结构占37.82%,β-折叠占14.65%,无规则卷曲占47.52%。用SWISS-MODEL以哺乳动物甲羟戊酸激酶蛋白(PDB ID:1kvk)为模板,预测LaPMK蛋白的三维结构,序列相似度为20.4%,在第3~495位氨基酸建模,模型覆盖率72%。从预测的三维模型中可以看到LaPMK可能以二聚体形式发挥作用,ATP结合位点位于LaPMK蛋白三维结构所形成的一个口袋状结构中(图 6),推测LaPMK催化的底物磷酸化反应可能在这个口袋状结构中进行[7]。
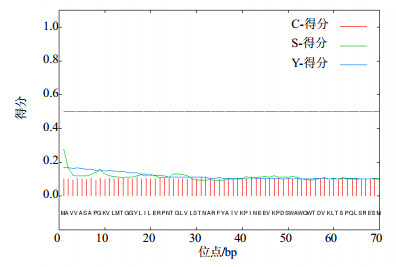
|
图 4 LaPMK基因的编码蛋白质信号肽预测分析(SignalP预测) Fig.4 Prediction of signal peptide of LaPMK protein encoding by LaPMK gene (SignalP prediction) |
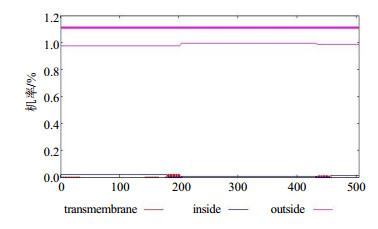
|
图 5 LaPMK基因的编码蛋白质的跨膜区预测结果(TMHMM预测) Fig.5 Prediction of transmembrane domains of LaPMK protein encoding by LaPMK gene (TMHMM prediction) |
|
图 6 LaPMK蛋白三维结构预测 Fig.6 Prediction of 3D structure of LaPMK protein |
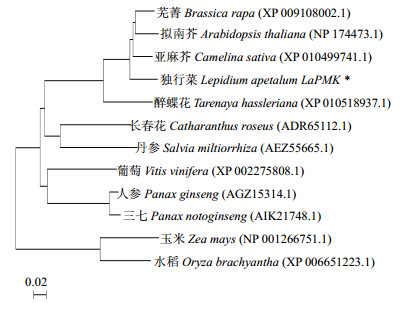
将LaPMK基因编码蛋白质的氨基酸序列提交到NCBI的蛋白质序列数据库进行BLASTP搜索,结果表明:LaPMK与芜菁的PMK蛋白序列相似性最高,达92%。从BLASTP搜索结果中选取与LaPMK基因编码的蛋白质相似性较高的13条蛋白质序列,绘制系统进化树(图 7)。可以看出LaPMK蛋白与芜菁Brassica rapa Linn.、拟南芥Arabidopsis thaliana (Linn.)Heynh.和亚麻芥Camelina sativa (Linn.)Crantz等十字花科植物的PMK蛋白处于同一分支上,亲缘关系较近,都属于双子叶植物。
|
图 7 LaPMK蛋白与其他物种PMK蛋白的系统进化树分析 Fig.7 Phylogenetic tree of LaPMK protein and PMK proteins from other species |
2.3 LaPMK基因原核表达载体的构建
用限制性内切酶EcoRI和HindⅢ分别对原核表达载体pET-32a(+)和测序正确的pMD19-T-LaPMK质粒进行双酶切,连接得到重组质粒pET32a-LaPMK,然后转化E.coli BL21(DE3),挑取单克隆,进行双酶切鉴定,结果显示有1 500 bp左右的目的条带(图 8)。将酶切正确的单克隆送测序公司测序,测序结果显示与目的基因LaPMK序列一致,未发生移码突变,表明已成功将LaPMK基因克隆到原核表达载体pET-32a上。
|
M-Marker 1、2-EcoRⅠ和HindⅢ双酶切 M-Marker 1, 2-double digestion results by EcoRⅠ and HindⅢ 图 8 原核表达载体pET32a-LaPMK的双酶切鉴定 Fig.8 Identification of prokaryotic expression vector pET32a-LaPMK with double digestion |
2.4 LaPMK蛋白的表达
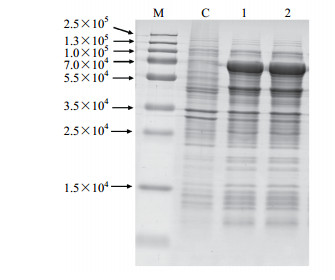
将测序正确的重组质粒pET32a-LaPMK转化E.coli BL21(DE3)后,用IPTG诱导LaPMK蛋白表达。在28 ℃、IPTG终浓度为0.4 mmol/L、150 r/min的条件下,诱导表达8 h后,提取总蛋白进行SDS-PAGE分析(图 9),蛋白相对分子质量与预测结果相一致,结果表明LaPMK基因编码的蛋白质原核表达成功。下一步研究需要检测原核表达LaPMK蛋白的可溶性,通过优化表达条件(温度、诱导时间、IPTG浓度等),提高原核表达LaPMK蛋白的可溶性,为纯化LaPMK蛋白和检测目的蛋白活性奠定基础,避免因诱导时间过长(24 h以上)、温度过高(35~37 ℃)等因素降低目的蛋白的可溶性,形成包涵体[10]。
|
M-Marker C-含pET32a空载体的E. coli菌株1、2-IPTG诱导的含pET32a-LaPMK质粒的E. coli菌株 M-Marker C-E. coli containing pET32a used as control 1, 2-E. coli containing pET32a-LaPMK under IPTG induction 图 9 重组LaPMK蛋白SDS-PAGE电泳 Fig.9 SDS-PAGE analysis on recombinant LaPMK protein |
3 讨论
丹参酮、紫杉醇、人参皂苷等萜类化合物具有重要的药用和经济价值,而关于独行菜中萜类物质生物合成途径的报道较少。独行菜以干燥成熟的种子入药,本研究从独行菜种子萌发后,长成幼苗的叶片中克隆得到了PMK基因,该基因编码蛋白的氨基酸序列与其他植物中MVA途径PMK蛋白的序列同源性高,表明LaPMK为MVA途径的第2个限速酶PMK,也是控制整个MVA代谢途径的关键酶之一[11],独行菜PMK蛋白作为其萜类物质生物合成途径的关键酶,具有重要的研究价值。有研究表明在植物体内的MVA途径中的MK和PMK基因表达量较低,提高它们的表达量可以提高萜类在植物中的产量,同时若PMK在植物体内过量表达,可以增加类异戊二烯衍生物的量[12-13]。目前PMK基因在植物研究中的报道较少,仅在药用植物阳春砂中克隆了PMK基因和检测不同组织中PMK基因表达差异,结果显示在叶中PMK基因表达量较高,茎、根中表达量相对较低,推测叶片可能是阳春砂中萜类的最主要合成部位[7]。
萜类化合物是植物次生代谢产物中最大的一个家族,在生物体内有着重要的生理作用,而且广泛应用于工农业生产和医药卫生中,但是萜类化合物在生物体内的量很低,在植物体内的量往往只达到百万分之一的水平,如何提高植物萜类产量一直是个热门研究方向[14]。近年来随着萜类化合物生物合成途径及其关键酶研究的深入,以分子生物学技术为基础的合成生物学和代谢工程,成为提高植物体内萜类化合物产量最有潜力的途径之一。所以,通过构建PMK基因的植物表达载体并通过农杆菌介导的遗传转化体系转化独行菜,对其进行遗传改良,有希望提高转基因独行菜中萜类物质的量,进一步培育高品质的独行菜新品种,具有重要的商业价值和现实意义。本实验首次从独行菜中克隆了PMK基因,利用pET32a原核表达载体和大肠杆菌BL21(DE3)进行原核表达,大肠杆菌BL21(DE3)菌株可以增加细胞质中蛋白质二硫键的形成,使得蛋白的可溶性更好,同时,利用pET32a载体表达的重组蛋白N端带有6个His Tag标签,方便后续重组蛋白的纯化[15]。通过构建原核表达载体pET32a-LaPMK,转入大肠杆菌BL21(DE3),成功诱导表达出LaPMK蛋白,并且目的蛋白表达量较高,为下一步纯化LaPMK蛋白进行蛋白质结构和生物学功能研究奠定了基础。该研究将有助于深入认识独行菜萜类化合物生物合成途径及其调控机制,也为独行菜萜类代谢工程提供候选基因。
| [1] | 李红伟, 郑晓珂, 弓建红, 等. 独行菜和播娘蒿化学成分及药理作用研究进展[J]. 药物评价研究 , 2013, 36 (3) :235–240. |
| [2] | Hyun J W, Shin J E, Lim K H, et al. Evomonoside:the cytotoxic cardiac glycoside from Lepidium apetalum[J]. Planta Med , 1995, 61 (3) :294–295. DOI:10.1055/s-2006-958084 |
| [3] | 刘强, 丛丽娜, 张宗申. 植物甾醇与三萜类皂苷生物合成基因调控的研究进展[J]. 安徽农业科学 , 2006, 34 (19) :4844–4846. |
| [4] | 常卿, 刘俊峰, 梁栋材.磷酸甲羟戊酸激酶PMK的重组表达和晶体生长[A]//第十次中国生物物理学术大会论文摘要集[C].北京:中国生物物理学大会, 2006. |
| [5] | Garcia D E, Keasling J D. Kinetics of phosphomevalonate kinase from Saccharomyces cerevisiae[J]. PLoS One , 2014, 9 (1) :e87112. DOI:10.1371/journal.pone.0087112 |
| [6] | 王宝莲, 樊庆琦, 李永波, 等. 甲羟戊酸激酶基因研究进展[J]. 中国农业科技导报 , 2011, 13 (3) :17–25. |
| [7] | 杨恩泽, 陆颖锶, 吴秋红, 等. 阳春砂磷酸甲羟戊酸激酶的基因克隆与表达谱分析[J]. 暨南大学学报:自然科学与医学版 , 2015, 36 (2) :1–7. |
| [8] | Andreassi J L, Leyh T S. Molecular functions of conserved aspects of the GHMP kinase family[J]. Biochemistry , 2004, 43 (46) :14594–14601. DOI:10.1021/bi048963o |
| [9] | Tamura K, Dudley J, Nei M, et al. MEGA4:Molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evol , 2007, 24 (8) :1596–1599. DOI:10.1093/molbev/msm092 |
| [10] | Galloway C A, Sowden M P, Smith H C. Increasing the yield of soluble recombinant protein expressed in E.coli by induction during late log phase[J]. Biotechniques , 2003, 34 (3) :524–526. |
| [11] | 张长波, 孙红霞, 巩中军, 等. 植物萜类化合物的天然合成途径及其相关合酶[J]. 植物生理学通讯 , 2007, 43 (4) :779–786. |
| [12] | Redding-Johanson A M, Batth T S, Chan R, et al. Targeted proteomics for metabolic pathway optimization:Application to terpene production[J]. Metab Eng , 2011, 13 (2) :194–203. DOI:10.1016/j.ymben.2010.12.005 |
| [13] | Singh P, Batth T S, Juminaga D, et al. Application of targeted proteomics to metabolically engineered Escherichia coli[J]. Proteomics , 2012, 12 (8) :1289–1299. DOI:10.1002/pmic.201100482 |
| [14] | Ajikumar P K, Tyo K, Carlsen S, et al. Terpenoids:opportunities for biosynthesis of natural product drugs using engineered microorganisms[J]. Mol Pharm , 2008, 5 (2) :167–190. DOI:10.1021/mp700151b |
| [15] | Rosano G L, Ceccarelli E A. Recombinant protein expression in Escherichia coli:advances and challenges[J]. Front Microbiol , 2014, 5 :1–17. |
 2016, Vol. 47
2016, Vol. 47


