2. 北京中医药大学中药学院, 北京 100102
2. School of Chinese Materia Medica, Beijing University of Chinese Medicine, Beijing 100102, China
雪莲Saussureae cum Flore Herba为菊科(Compositea)凤毛菊属Saussurea DC.植物,全世界共有40余种及3个变种,药用雪莲共有12种和1个变种[1],其中天山雪莲Saussurea involucrata (Kar. et Kir.) Sch.-Bip.是我国珍稀的名贵中药[2],主要生长在我国西南和西北高寒地区,有着长期的药用历史以及丰富的民间用药经验[3],在抗风湿、镇痛、调节心血管系统、抗癌、抗缺氧、延缓衰老、终止妊娠、防辐射等多方面具有很大的开发利用价值[4-8]。随着雪莲药用价值的发现,其用量增大,资源大大减少,目前雪莲已濒危,资源贮量下降,乱采滥挖野生雪莲现象十分严重,加之人工栽培困难,已被列为国家三级濒危物种而受到保护[9]。因此利用雪莲细胞生产活性成分是缓解野生雪莲资源短缺的有效途径之一。
鲨烯合酶(squalene synthase,SQS)是三萜类生物合成途径中的关键酶之一,使法呢基焦磷酸(gerany lpyrophosphate,FPP)缩合生成鲨烯(squalene,SQ)[10-11],再经过化学结构修饰成为三萜类化合物。目前已经从拟南芥[12]、烟草[13]、水稻[14]、青蒿[15]、人参[16]、三七[17]、乌拉尔甘草[18]、东北红豆杉[19]、丹参[20]、大豆[21]、灵芝[22]、酵母[23]、刺五加[24]等中克隆出不同的SQS基因,通过对这些SQS基因进行研究,得出如下结论:(1)不同的物种含有SQS基因的数目不同;(2)不同的SQS基因存在着不同的拷贝形式;(3)并不是所有的SQS基因都具有鲨烯合酶活性;(4)SQS基因表达的最适条件各不相同。由于SQS基因存在特性与多态性[25-26],因而对不同SQS的研究就显得十分必要。
雪莲中所含的甾醇类化合物主要为β-谷甾醇[27]等,由于其本身无毒性,且具有乳化性与稳定性,还有降低胆固醇、消炎解热、抗氧化、抗肿瘤和抗癌等药理作用,因此在医药、化妆品、饲料、化工等领域都得到广泛应用[28-33]。根据甾醇类与三萜类化合物的生物合成途径可知,其都以SQS为关键酶,催化生成前体物质鲨烯,再经过不同化学结构修饰而成[34]。因此研究SQS在天山雪莲细胞中的蛋白特性和表达水平对于提高下游β-谷甾醇的量至关重要。
本实验拟利用天山雪莲红色系、绿色系、粉色系细胞,克隆其SQS基因,并初步比较不同细胞系SQS蛋白特性和表达水平,且通过结合β-谷甾醇的化学定量分析结果,研究SQS基因结构和蛋白特性对天山雪莲细胞β-谷甾醇积累的影响,为进一步研究SQS催化β-谷甾醇合成机制提供理论依据。
1 材料与试剂天山雪莲红色系、绿色系、粉色系细胞由大连普瑞康生物技术有限公司提供;Trace 1310 TSQ 8000气相质谱仪(美国Thermo ScientificTM公司);对照品β-谷甾醇(质量分数≥95%)和5α-胆甾烷均购自美国Sigma公司;BSTFA[双三氟乙酰胺,含1% TMCS(三甲基氯硅烷)]购自Supelco公司;二氯甲烷、正己烷、95%乙醇、浓盐酸、氢氧化钾(国药集团化学试剂有限公司,分析纯)。pGEM-T3载体购自Promega公司;原核表达载体pGEX-4T-1为本实验室保存;Trizol试剂购自Invitrogen生物技术有限公司;大肠杆菌DH5α、Transetta(DE3)感受态购自全氏金有限公司;DNA测序由北京睿博生物科技公司完成;寡核苷酸引物由上海生工生物工程公司合成;T4 DNA聚合酶购自上海生工生物工程公司;BamH I和Sma I限制性内切酶购自NEB有限公司;氨苄青霉素、β-巯基乙醇、IPTG等化学试剂购于Sigma公司;反转录试剂盒(Takara Prime ScriptTM 1st Strand cDNA Synthesis Kit)购自TaKaRa公司;其他生化试剂均为国产分析纯。
2 方法 2.1 总RNA提取及cDNA的合成冰冷的PBS液5~10 mL洗涤天山雪莲细胞2次,采用Trizol法提取总RNA,1.5%的琼脂糖凝胶电泳用于评价RNA的质量。ND2000测定总RNA的吸光度(A260、A280)值,选择A260/A280为1.8~2.0的总RNA反转录成cDNA。
2.2 SiSQS1和SiSQS2基因cDNA的克隆天山雪莲SiSQS1和SiSQS2来源于天山雪莲细胞转录组数据。用Primer Premier 5.0软件设计特异性引物。SiSQS-F:5’-ATGGGGAGTTTAAAAGCA-GTGTTGA-3’;SiSQS-R:5’-TTACAACGTAAGCTT-GATTTTATTT-3’。以cDNA为模板进行PCR扩增,取阳性样品与pEASY-T3 Cloning Kit进行连接,并导入大肠杆菌DH5α感受态细胞进行转化,并均匀涂在含氨苄青霉素抗性的LB培养板上,取阳性PCR样品进行测序,将测序结果与天山雪莲细胞转录组数据进行比对,判断目的片段是否与克隆载体pEASY-T3 Cloning Kit连接成功。
2.3 SiSQS1和SiSQS2基因的生物信息学分析将测序获得的序列结果使用ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)查找开放阅读框(ORF)。利用在线工具Protparam(http://www.expasy.ch/tools/protparam.html)预测基因编码蛋白的相对分子质量、氨基酸数目、等电点、不稳定系数、脂肪指数、亲水性/疏水性和编码区全长等理化性质;采用CDD(http://www.ncbi.mlm.nih.gov/Structure/cdd/wrpsb.cgi)进行蛋白质结构域分析;采用CFSSP(http://swissmodel.expasy.org/)进行蛋白质二级结构分析;利用Swiss Model程序(http://swissmodel.expasy.org/),根据基因氨基酸序列进行建模,预测蛋白质的三级结构。用DNAMAN软件对序列进行多重比对以及限制性酶切位点的预测,用ClustalW软件与其他物种的氨基酸序列进行比较(表 1),并用MEGA 6.06软件构建Neighbor-joining系统进化树,bootstrap重复次数为1 000次。
|
|
表 1 系统进化树中不同物种来源的SQS氨基酸序列 Table 1 SQS amino acid sequence in source of different species in phylogenetic tree |
2.4 pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核表达载体的构建
对测序后的序列进行分析,设计带有酶切位点的引物。两条序列酶切位点引物一样,BamHI-F:5’-CGCGGATCCATGGGGAGTTTGGGAGCGAT-3’;Sma I-R:5’-TCCCCCGGGTCATTTAGTGGAGAG-AAATGCAAAC-3’。以重组质粒为模板,进行PCR扩增,取琼脂糖凝胶电泳检测的阳性样品进行切胶回收。并与表达载体pGEX-4T-1分别进行BamH I和Sma I双酶切处理,并进行切胶回收。将切胶回收后的目的片段与表达载体pGEX-4T-1用T4 DNA连接酶在22 ℃连接4 h,将连接产物转化到DH5α感受态细胞。将转化后的沉淀物均匀涂布在含amp抗性的LB培养板上,挑取单克隆进行菌液PCR阳性检验、双酶切检验与测序。挑选阳性菌于含amp抗性的LB培养液中扩大培养并提取质粒,将质粒转化到大肠杆菌Transetta(DE3)表达感受态中,转化表达宿主菌。
2.5 pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核诱导表达及条件优化将转化产物均匀涂布在含amp抗性的LB平板上,挑取单克隆抗体进行菌液PCR阳性检验,取阳性样品于含amp抗性的LB培养液中摇菌10 h后,并于37 ℃ 250 r/min振荡扩大培养至宿主菌的A600值为0.6时,加终浓度为0.4 mmol/L的异丙基-β-D-硫代半乳糖苷(IPTG)于25 ℃,200 r/min摇床中诱导表达,诱导12 h后离心再重悬沉淀。同时对影响蛋白表达的4个关键因素进行条件优化,包括诱导时间(4、8、16、24 h),诱导温度(16、20、25、30、37 ℃),IPTG浓度(0.2、0.4、0.6、0.8、1.0 mmol/L)诱导时宿主菌的A600值(0.2、0.4、0.6、0.8、1.0)。取30 μL菌液加20 μL的2.5×蛋白染色上样,沸水浴10 min后,12 000 r/min离心10 min,取上清进行SDS-PAGE分析,考马斯亮蓝染色2 h后,用洗脱液脱色至条带清晰,扫描图片后并保存。
2.6 RT-PCR分析SiSQS1、SiSQS2基因表达水平利用Primer Primer 5.0设计雪莲SiSQS1与SiSQS2基因实时荧光定量引物,扩增产物长度在80~200 bp,送由生工生物工程(上海)有限公司合成,见表 2。体系按照SYBR Premix Ex TaqTM Kit(Takara公司)说明书配制,cDNA材料来源于雪莲红色系、绿色系和粉色系的悬浮细胞,每个色系包括3个生物样本,每个反应重复3次(包括阴性对照),在LightCycler® 480上进行,结束后进行熔解曲线分析。各基因表达量以内参基因GAPDH作为标准进行相对定量,相对定量方法采用2−ΔΔCt法。
|
|
表 2 RT-PCR引物 Table 2 Primers of RT-PCR |
2.7 雪莲三色系细胞中β-谷甾醇分析 2.7.1 色谱条件
采用TraceGOLDTM TG-5MS气相色谱柱(30 m×0.25 mm,0.25 μm;Thermo ScientificTM,美国),程序升温(初始温度80 ℃,保持2 min;以20 ℃/min升温至280 ℃,保持15 min)。载气为氦气,体积流量1.0 mL/min;进样量1 μL,分流比25:1,进样口温度250 ℃。
2.7.2 质谱条件电离方式为电子轰击离子源(EI离子源),电离电压70 eV,质谱范围:40~550 m/z;离子源温度280 ℃。
2.7.3 对照品溶液的制备精密称取适量β-谷甾醇对照品和5α-胆甾烷内标置50 mL棕色量瓶中,正己烷溶解并定容到刻度,摇匀,即得对照品溶液和内标溶液。
2.7.4 供试品溶液的制备分别称取0.5 g雪莲三色系细胞干燥样品于20 mL具塞试管中,加入1 mL 5α-胆甾烷内标溶液及3 mL 4 mol/L HCl的95%乙醇溶液,于80 ℃水解120 min,水浴冷却至室温。
2.7.5 线性关系考察精密移取适量β-谷甾醇对照品溶液于10 mL棕色量瓶中,配制成1.0、5.0、10.0、20.0、25.0、50.0 μg/mL的对照品溶液,各加入0.10 mg/mL的内标溶液1 mL,内标质量浓度均为10 μg/mL。上述不同质量浓度对照品溶液各取1 mL,氮气吹干后,加入200 μL BSTFA(含1% TMCS)试剂,80 ℃下衍生60 min后,冷却加入0.25 mL正己烷,按“2.7.1和2.7.2”项下条件进行GC-MS分析。以各对照品溶液质量浓度为横坐标(X),相应对照品溶液与内标的峰面积之比为纵坐标(Y)进行回归分析,得到回归方程为Y=0.023 9 X-0.010 8(r=0.999 7),线性范围1.00~50.0 μg/mL。
2.7.6 β-谷甾醇测定取“2.7.3”项溶液,酸水解液中加入6 mL 4.0 mol/L KOH的95%乙醇溶液,于80 ℃皂化120 min,水浴冷却至室温。取2 mL皂化液于20 mL具塞玻璃试管中,加入5 mL正己烷、1 mL去离子水,室温下超声振荡10 min。取2.5 mL正己烷萃取液于色谱瓶中,用氮气吹干;加入200 μL BSTFA(含1% TMCS)试剂80 ℃下衍生60 min后,冷却加入0.25 mL正己烷,进行GC-MS分析,进样量1.0 μL。按“2.7.1和2.7.2”项下条件进行GC-MS分析,并分别计算雪莲三色系细胞中β-谷甾醇的量。
2.8 β-谷甾醇量与SiSQS1、SiSQS2基因表达水平的相关性分析参考Liang等[35]报道的方法,使用SPSS 20.0软件对雪莲细胞中β-谷甾醇量与SiSQS1、SiSQS2基因表达水平做线性相关性分析。
3 结果与分析 3.1 SiSQS1和SiSQS2基因的克隆利用DNAMAN软件结合ORF Finder在线软件对雪莲转录组数据中SiSQS1、SiSQS2基因序列进行分析,通过设计特异性引物克隆雪莲cDNA SiSQS1、SiSQS2,并将其重组入T3载体中,菌液PCR后测序结果与预测结果相一致,表明pGEM-T3载体已经插入SiSQS1、SiSQS2基因。通过blast在线比较,推测SiSQS1、SiSQS2基因为鲨烯合酶基因,分别命名为SiSQS1(GenBanK注册号KU057087)、SiSQS2(GenBanK注册号KU057088)。
3.2 SiSQS1、SiSQS2基因的生物信息学分析利用在线工具Protparam预测SiSQS1和SiSQS2都含1 257 bp完整的开放阅读框,编码的氨基酸数目都为418 aa;NCBI的Conserved domains在线工具表明38~320 aa都为Isoprenoid_ Biosyn_C1 superfamily家族;跨膜域分析结果表明386~408 aa都有跨膜结构域。SiSQS1、SiSQS2编码蛋白的理化性质如等电点都为7.14,脂肪指数都为95.93,但SiSQS1编码蛋白的相对分子质量为47 691,SiSQS2编码蛋白的相对分子质量为47 677。
3.2.1 SiSQS1与SiSQS2基因氨基酸序列比对对雪莲SiSQS1与SiSQS2基因编码的氨基酸序列进行比对,发现一致性为99.76%,氨基酸序列只在242 aa位点处发生了变异,根据NCBI的Conserved domains分析结果可得知该变异位点处于Isoprenoid_Biosyn_ C1 superfamily结构功能域中(图 1)。
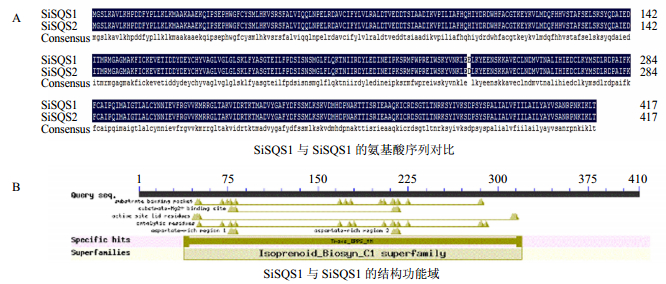
|
图 1 SiSQS1和SiSQS2基因的氨基酸序列比对(A)与结构功能域(B) Fig.1 Comparison on amino acid sequences (A) and structure function domain (B) of SiSQS1 and SiSQS2 |
3.2.2 SiSQS1与SiSQS2系统进化树分析
雪莲SiSQS1、SiSQS2与GenBank中20种不同物种来源的SQS氨基酸序列进行比对,软件MEGA 6.06采用邻接法构建系统进化树进行聚类分析。从进化树物种可以明显地看到,植物、藻类、动物、真菌、细菌分别聚为一支,在进化树上的远近与其所属类别的亲缘关系一致。且雪莲SiSQS1与SiSQS2明显聚为一支,亲缘关系最近,与这2条基因来源于同一植物雪莲的事实相符;与同为菊科植物青蒿的亲缘较近,与植物进化的亲缘关系也相符合(图 2)。
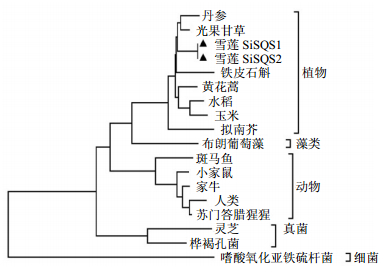
|
图 2 SiSQS1、SiSQS2与其他物种SQS氨基酸序列的系统进化树分析 Fig.2 Phylogenetic tree of SQS amino acid sequences encoded by SiSQS1 and SiSQS2 with other species |
3.3 pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核表达载体构建与原核表达
将pGEX-4T-1-SiSQS1和pGEX-4T-1-SiSQS2转化至大肠杆菌transetta(DE3)中,挑取若干单克隆抗体,进行菌液PCR检验,并挑取阳性单克隆菌液进行测序,重组质粒测序结果表明目的基因成功插入表达载体中,可进行下一步原核表达。同时将构建好的原核表达载体于大肠杆菌transetta(DE3)进行原核表达,SDS-PAGE电泳结果表明与空载pGEX-4T-1相比,pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2在68 000处出现目的蛋白条带(图 3),表明pGEX-4T-1-SiSQS1和pGEX-4T-1-SiSQS2在大肠杆菌中成功表达。
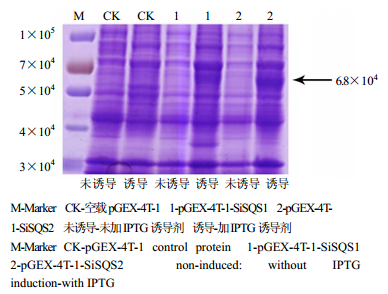
|
M-Marker CK-空载pGEX-4T-1 1-pGEX-4T-1-SiSQS1 2-pGEX-4T-1-SiSQS2 未诱导-未加IPTG诱导剂 诱导-加IPTG诱导剂 M-Marker CK-pGEX-4T-1 control protein 1-pGEX-4T-1-SiSQS1 2-pGEX-4T-1-SiSQS2 non-induced: without IPTG induction-with IPTG 图 3 pGEX-4T-1-SiSQS1和pGEX-4T-1-SiSQS2的原核表达 Fig.3 Prokaryotic expression of pGEX-4T-1-SiSQS1 and pGEX-4T-1-SiSQS2 |
3.4 诱导温度对pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核表达的影响
保持诱导时间16 h,IPTG浓度为0.4 mmol/L,诱导时宿主菌A600值为0.6这3个因素不变,探讨5个诱导温度(16、20、25、30、37 ℃)对pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核表达的影响(图 4),结果表明pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2最适表达温度不同,pGEX-4T-1-SiSQS1蛋白最适表达温度为25 ℃,pGEX-4T-1-SiSQS2蛋白最适表达温度为16 ℃。

|
M-Marker CK-空载pGEX-4T-1 1~5-诱导温度为16、20、25、30、37 ℃ M-Markers CK-pGEX-4T-1 control protein 1—5-induced temperature: 16、20、25、30、37 ℃ 图 4 pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2在不同诱导温度处理下的原核表达 Fig.4 Prokaryotic expression of pGEX-4T-1-SiSQS1 and pGEX-4T-1-SiSQS2 under different temperature |
3.5 IPTG浓度对pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核表达的影响
保持诱导温度20 ℃,诱导时间16 h,宿主菌A600值为0.6不变,考察5个IPTG浓度(0.2、0.4、0.6、0.8、1.0 mmol/L)对pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核表达的影响(图 5)。结果表明IPTG浓度对pGEX-4T-1-SiSQS1表达量几乎没有影响;对于pGEX-4T-1-SiSQS2来说,IPTG浓度为0.6 mmol/L时,表达量最大。

|
M-Marker CK-空载pGEX-4T-1 1~5-IPTG浓度为0.2、0.4、0.6、0.8、1.0 mmol/L M-Marker CK-pGEX-4T-1 control protein 1—5-IPTG concentration 0.2、0.4、0.6、0.8、1.0 mmol/L 图 5 pGEX-4T-1-SiSQS1和pGEX-4T-1-SiSQS2在不同IPTG浓度处理下的原核表达 Fig.5 Prokaryotic expression of pGEX-4T-1-SiSQS1 and pGEX-4T-1-SiSQS2 under different IPTG concentration |
3.6 诱导时间对pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核表达的影响
保持诱导温度20 ℃,IPTG浓度0.6 mmol/L,诱导时宿主菌A600值3个因素不变,考察诱导时间(4、8、16、24 h)对pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核表达的影响(图 6)。实验结果表明pGEX-4T-1-SiSQS1在诱导8 h时表达量最大,pGEX-4T-1-SiSQS2在诱导4 h时表达量最大;同时说明随着诱导时间的延长,并不利于目的蛋白的表达。

|
M-Marker CK-空载pGEX-4T-1 1~4-诱导时间-4、8、16、24 h M-Marker CK-pGEX-4T-1 control protein 1—4-induction time-4、8、16、24 h 图 6 pGEX-4T-1-SiSQS1和pGEX-4T-1-SiSQS2在不同诱导时间处理下的原核表达 Fig.6 Prokaryotic expression of pGEX-4T-1-SiSQS1 and pGEX-4T-1-SiSQS2 under different induction time |
3.7 宿主菌密度对pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核表达的影响
保持诱导温度20 ℃,诱导时间16 h,IPTG浓度0.6 mmol/L不变,考察不同密度宿主菌(A600值为0.2、0.4、0.6、0.8、1.0)对pGEX-4T-1-SiSQS1与pGEX-4T-1-SiSQS2原核表达的影响(图 7)。实验结果表明宿主菌A600值在0.2~0.4,pGEX-4T-1-SiSQS1和pGEX-4T-1-SiSQS2表达量在不断增加,超过0.4后,表达量都下降。说明2条基因在宿主菌A600值为0.4时表达量最大,高密度的宿主菌反而不利于蛋白表达。
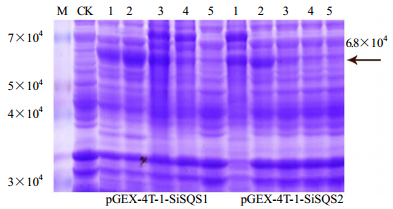
|
M-Marker CK-空载pGEX-4T-1 1~5-宿主菌A600值为0.2、0.4、0.6、0.8、1.0 M-Marker CK-pGEX-4T-1 control protein 1—5-host bacteria A600: 0.2、0.4、0.6、0.8、1.0 图 7 pGEX-4T-1-SiSQS1和pGEX-4T-1-SiSQS2在不同密度宿主菌的原核表达 Fig.7 Prokaryotic expression of pGEX-4T-1-SiSQS1 and pGEX-4T-1-SiSQS2 in different densities of host bacteria |
3.8 雪莲三色系细胞中β-谷甾醇的化学定量分析
GC-MS分析雪莲三色系细胞中β-谷甾醇量,结果表明绿色系细胞中β-谷甾醇量最高,粉色系细胞中β-谷甾醇量最低。绿色系、红色系与粉色系细胞样品中β-谷甾醇量分别为(374.54±2.20)、(279.71±3.74)、(155.19±0.06)μg/g(n=3)。
3.9 SiSQS1与SiSQS2基因表达分析RT-PCR分析SiSQS1与SiSQS2基因在雪莲红色系、绿色系和粉色系细胞中的表达水平,结果表明SiSQS1在雪莲绿色系、红色系与粉色系细胞中表达水平依次是0.88±0.05、0.37±0.06、0.28±0.02(n=3);SiSQS2在雪莲绿色系、红色系与粉色系细胞中表达水平依次是1.15±0.03、0.64±0.04、0.50±0.03(n=3)。可以看出SiSQS1与SiSQS2在雪莲绿色系细胞中的表达水平都是最高的,其次是红色系细胞,再次是粉色系细胞。并对细胞中β-谷甾醇量与SiSQS1、SiSQS2基因的表达水平做相关性分析,发现β-谷甾醇量与SiSQS1基因表达水平相关性系数为0.92,与SiSQS2相关性系数为0.89。
4 讨论一般认为活性成分量差异与其生物合成途径上关键酶基因的表达是有直接关系的。本研究通过比较天山雪莲细胞中2条鲨烯合酶SiSQS1和SiSQS2的位点变异、蛋白原核表达特性与表达水平差异来推测SiSQS1和SiSQS2的表达可能会影响β-谷甾醇前体物质的合成,进而影响β-谷甾醇的积累。因此,本实验对天山雪莲三色系细胞中β-谷甾醇进行定量分析比较,发现β-谷甾醇量由高到低依次是绿色系、红色系与粉色系细胞;同时对β-谷甾醇生物合成途径上关键酶SiSQS1和SiSQS2在天山雪莲三色系细胞中的转录水平进行了比较,结果表明SiSQS1与SiSQS2在天山雪莲绿色系细胞中表达水平最高,其次是红色系细胞,最后是粉色系细胞,这与天山雪莲细胞中β-谷甾醇量呈正相关,相关性系数分别为0.92和0.89。从β-谷甾醇量与基因表达水平相关性分析结果可推测SiSQS1和SiSQS2调控β-谷甾醇积累可能存在功能差异。
对SiSQS1和SiSQS2蛋白特性进行了初步比较,发现二者存在一个氨基酸位点突变,且位于结构功能域中。进一步蛋白原核表达实验结果表明,在温度25 ℃、IPTG浓度为0.6 mmol/L、宿主菌A600值为0.6、诱导时间为16 h时SiSQS1和SiSQS2都有表达,但诱导温度、诱导时间、IPTG浓度和诱导时宿主菌密度对上述两个蛋白表达特性的影响存在差异,暗示242E/D残基变异可能影响SiSQS蛋白的表达,且二者在催化活性以及对β-谷甾醇积累调控作用上也可能存在着一定的差异,都有待于进一步深入研究。因此,SiSQS1和SiSQS2可用来研究调控β-谷甾醇积累的机制,为以β-谷甾醇为目的成分,以SQS为核心,以二者之间相关性分析为桥梁的天山雪莲细胞研究体系提供技术支撑,同时利用天山雪莲细胞生产活性成分也是缓解野生天山雪莲资源短缺的有效途径之一。
| [1] | 王瑛, 张本印, 陶燕铎, 等. 雪莲的化学成分与药理作用研究进展[J]. 光谱实验室 , 2013, 30 (2) :530–535. |
| [2] | Kuo C L, Agrawal D C, Chang H C, et al. In vitro culture and production of syringin and rutin in Saussurea involucrata (Kar.et Kir.)-an endangered medicinal plant[J]. Bot Studies , 2015, 56 (12) :1–8. |
| [3] | Ning H, Liu M, Kang X, et al. Biological characteristics of vegetative growth of cultivated Saussurea involucrata in a low altitude region[J]. Med Plant , 2014, 5 (6) :1–6. |
| [4] | Qiu H, Zhang L, Liu C, et al. Cloning and characterization of a novel dehydrin gene, SiDhn2, from Saussurea involucrata Kar.et Kir[J]. Plant Mol Biol , 2014, 84 (6) :707–718. DOI:10.1007/s11103-013-0164-7 |
| [5] | 翟科峰, 段红, 邢建国, 等. 天山雪莲提取物纯化前后各部位抗炎镇痛作用[J]. 中国医院药学杂志 , 2010, 30 (5) :374–377. |
| [6] | 韦善君, 武运芳, 罗云燕, 等. 濒危药用植物新疆雪莲资源的研究进展[J]. 中央民族大学学报:自然科学版 , 2014, 23 (2) :10–15. |
| [7] | Li M, Li H, Hu X, et al. Genetic transformation and overexpression of a rice Hd3a induces early flowering in Saussurea involucrata Kar.et Kir.ex Maxim[J]. Plant Cell, Tiss Org Cult (PCTOC) , 2011, 106 (3) :263–371. |
| [8] | 范文霞, 张献, 曹坦. 雪莲培养物的总抗氧化能力与抗黑色素能力研究[J]. 生物技术世界 , 2014 (11) :86–87. |
| [9] | 肖皖, 波拉提, 王歆君, 等. 雪莲化学成分和药理活性研究进展[J]. 现代药物与临床 , 2011, 26 (5) :344–348. |
| [10] | 陶晨陈, 马成通, 吴耀生, 等. 红花栝楼鲨烯合酶基因的克隆及其序列分析[J]. 中草药 , 2015, 46 (7) :1034–1041. |
| [11] | Singh A K, Dwivedi V, Rai A, et al. Virus-induced gene silencing of Withania somnifera squalene synthase negatively regulates sterol and defence-related genes resulting in reduced withanolides and biotic stress tolerance[J]. Plant Biotechnol J , 2015, 13 (9) :1287–1299. DOI:10.1111/pbi.12347 |
| [12] | Mirjalili M H, Moyano E, Bonfill M, et al. Overexpression of the Arabidopsis thaliana squalene synthase gene in Withania coagulans hairy root cultures[J]. Biol Planta , 2011, 55 (2) :357–360. DOI:10.1007/s10535-011-0054-2 |
| [13] | 张毅, 刘彦, 王红, 等. 转青蒿反义鲨烯合酶基因对烟草鲨烯合酶基因表达的影响[J]. 农业生物技术学报 , 2005, 13 (4) :416–422. |
| [14] | 彭梅芳, 甘凤, 潘春梅, 等. 鲨烯合酶RNAi载体构建及对水稻的遗传转化研究[J]. 西南农业学报 , 2015, 28 (4) :1413–1418. |
| [15] | Yang R Y, Feng L L, Yang X Q, et al. Quantitative transcript profiling reveals down-regulation of a sterol pathway relevant gene and overexpression of artemisinin biogenetic genes in transgenic Artemisia annua plants[J]. Planta Med , 2008, 74 (12) :1510–1516. DOI:10.1055/s-2008-1081333 |
| [16] | Kim T D, Han J Y, Huh G H, et al. Expression and functional characterization of three squalene synthase genes associated with saponin biosynthesis in Panax ginseng[J]. Plant Cell Physiol , 2011, 52 (1) :125–137. DOI:10.1093/pcp/pcq179 |
| [17] | 孙颖, 赵恒伟, 葛锋, 等. 三七中SS基因超表达载体的构建及其遗传转化[J]. 药学学报 , 2013, 48 (1) :138–143. |
| [18] | Yang R, Yuan B, Ma Y, et al. HMGR, SQS, -AS, and Cytochrome P450 monooxygenase genes in glycyrrhiza uralensis[J]. Chin Herb Med , 2015, 7 (4) :290–295. DOI:10.1016/S1674-6384(15)60054-5 |
| [19] | Huang Z, Jiang K, Pi Y, et al. Molecular cloning and characterization of the yew gene encoding squalene synthase from Taxus cuspidata[J]. J Biochem Mol Biol , 2007, 40 (5) :625–635. DOI:10.5483/BMBRep.2007.40.5.625 |
| [20] | 马艺沔, 袁丽钗, 张林甦, 等. 2个丹参鲨烯合酶基因的克隆和鉴定[J]. 中草药 , 2014, 45 (9) :1307–1312. |
| [21] | Nguyen H T M, Neelakadan A K, Quach T N, et al. Molecular characterization of Glycine max squalene synthase genes in seed phytosterol biosynthesis[J]. Plant Physiol Biochem , 2013, 73 (2) :23–32. |
| [22] | Zhao M, Liang W, Zhang D, et al. Cloning and characterization of squalene synthase (SQS) gene from Ganoderma lucidum[J]. J Micro Biotech , 2007, 17 (7) :1106–1112. |
| [23] | 朱明, 王彩霞, 李春. 工程化酿酒酵母合成植物三萜类化合物[J]. 化工学报 , 2015, 66 (9) :3350–3356. |
| [24] | 邢朝斌, 劳凤云, 龙月红, 等. 刺五加鲨烯合酶和鲨烯环氧酶基因单核苷酸多态性及其与总皂苷量的相关性研究[J]. 中草药 , 2012, 43 (10) :2020–2024. |
| [25] | 刘春生, 刘颖. 药用植物功能基因的研究思路与展望-以甘草为例[J]. 中国实验方剂学杂志 , 2015, 21 (1) :1–6. |
| [26] | 刘颖, 陈宏昊, 文浩, 等. 甘草鲨烯合酶1基因多态性及其与β-香树脂醇合成酶共表达对β-香树脂醇积累的影响研究[J]. 药学学报 , 2014, 49 (5) :734–741. |
| [27] | 景临林, 何蕾, 樊鹏程, 等. 天山雪莲抗缺氧活性成分研究[J]. 中药材 , 2015, 38 (1) :89–92. |
| [28] | 袁金伟, 王帆, 买文鹏, 等. β-谷甾醇的结构修饰研究进展[J]. 河南工业大学学报:自然科学版 , 2015, 36 (2) :107–112. |
| [29] | Ramadan M F. Oxidation of β-sitosterol and campesterol in sunflower oil upon deep-and pan-frying of French fries[J]. J Food Sci Technol , 2015, 52 (10) :6301–6311. DOI:10.1007/s13197-015-1738-y |
| [30] | Wang X, Huang W, Lei L, et al. Blockage of hydroxyl group partially abolishes the cholesterol-lowering activity of β-sitosterol[J]. J Funct Foods , 2015, 12 :199–207. DOI:10.1016/j.jff.2014.11.019 |
| [31] | Bin Sayeed M S, Ameen S S. Beta-Sitosterol:A promising but orphan nutraceutical to fight against cancer[J]. Nutrit Cancer , 2015, 67 (8) :1216–1222. DOI:10.1080/01635581.2015.1087042 |
| [32] | Mahmood T, Bibi Y, Zafar R, et al. Complexation of β-sitosterol with tris (dibenzylideneacetone) dipalladium and its anti-microbial activity[J]. Pakistan J Pharm Sci , 2015, 28 (2) :631–634. |
| [33] | Sriraman S, Ramanujam G M, Ramasamy M K, et al. Identification of beta-sitosterol and stigmasterol in Bambusa bambos (L.) Voss leaf extract using HPLC and its estrogenic effect in vitro[J]. J Pharm Biomed Anal , 2015, 115 :55–61. DOI:10.1016/j.jpba.2015.06.024 |
| [34] | Vishwakarma R K, Patel K, Sonawane P, et al. Squalene synthase gene from medicinal herb bacopa monniera:molecular characterization, differential expression, comparative modeling, and docking studies[J]. Plant Mol Biol Rep , 2015, 33 (6) :1675–1685. DOI:10.1007/s11105-015-0864-z |
| [35] | Liang J, Jiang C, Peng H, et al. Analysis of the age of Panax ginseng based on telomere length and telomerase activity[J]. Sci Rep , 2015, 5 :126–135. |
 2016, Vol. 47
2016, Vol. 47


