细胞色素P450还原酶(EC 1.6.2.4,cytochrome P450 reductase,简称CPR或POR)是一种在许多代谢进程中必需的酶,主要参与细胞色素P450氧化酶系(cytochrome P450 oxidoreductase system,P450s)催化的初级及次级代谢反应,如萜类物质代谢、药物代谢、外源化合物代谢等[1-2]。在此氧化系统中,CPR的功能是从底物NADPH获取电子,通过FMN和FAD辅因子传递给细胞色素P450,氧化底物;CPR与P450s间的电子传递是P450s氧化反应的限速步骤[3]。此外,CPR还能向细胞色素b5、鲨烯单氧化酶、血红素加氧酶提供电子,能直接催化一些前体药物的单原子还原活化[2, 4]。
茯苓Poria cocos (Schw.) Wolf是一种腐生或寄生在松科植物赤松和马尾松根部的高等真菌,隶属于担子菌门(Basidiomycetes)多孔菌科(Polyporaceae)[5]。茯苓菌核是一种传统中药,约10%的中成药(约300种)原料配方中含有茯苓,也被用于食品添加剂、膳食、保健品、化妆品等[6-7]。茯苓具有抗肿瘤、抗氧化、抗惊厥等多种生物活性,其主要化学成分为多糖及三萜类化合物[5]。
P450参与萜类物质代谢已有大量研究报道,如CYP71AV1参与青蒿素的生物合成[8]、CYP88D6与CYP72A154参与甘草酸的生物合成[9-10]、CYP76AH1参与丹参酮的生物合成[11]、在灵芝中大量CYP基因被推测与灵芝三萜合成相关[12]。此外,研究发现担子菌基因组中存在大量CYP基因,且数量较其他真菌多;另有认为棕腐菌比白腐菌拥有丰富的CYP基因,这都让CYP被认为与担子菌的生物学、生态、进化密切相关[13-14]。
本实验室通过对茯苓转录组测序及分析,提出了茯苓中主要活性成分三萜骨架的生物合成途径,并注释出了249个Unigene为CYP,且推测多个参与茯苓酸、土莫酸等三萜化合物的生物合成[15]。本研究在上述研究基础之上,利用茯苓的高通量测序结果,挖掘出茯苓CPR(PcCPR)基因相关序列,通过RACE(rapid-amplification of cDNA ends)技术扩增得到PcCPR基因cDNA全长,PCR得到对应基因组DNA序列。通过生物信息学方法分析PcCPR基因及其编码蛋白,为阐释PcCPR基因功能提供理论依据。从而为研究茯苓CYP基因功能乃至解析茯苓三萜的生物合成途径奠定基础。
1 材料与方法 1.1 材料茯苓Poria cocos (Schw.) Wolf菌核于2012年采自湖北省英山县石头咀镇的栽培田间,并通过分离纯化得到纯培养。28℃下,在马铃薯葡萄糖培养基(PDA)上培养7 d,收集得到菌丝体。
1.2 方法 1.2.1 核酸提取茯苓菌丝总RNA的提取使用RNAiso Plus(Takara,大连)试剂提取,提取方法依据其说明书进行。茯苓DNA的提取采用CTAB法。
1.2.2 PcCPR基因的克隆根据茯苓的转录组数据[15]中注释为CPR基因的Unigene设计RACE引物CPR-31、CPR-32、CPR-51、CPR-52(表 1)。3’RACE方法[16]使用经典3’RACE方法,使用PrimeScript Ⅱ 1st strand cDNA synthesis试剂盒(Takara,大连),以QT引物对提取的茯苓总RNA进行反转录,得到第1链cDNA产物用Tricine-EDTA稀释10倍作为PCR模版。3’RACE第1轮PCR反应体系:0.2μmol/L dNTPs,0.25μmol/L QO,0.25μmol/L CPR-31,1 U Ex Taq酶,1μL cDNA模版,20μL体系。PCR程序为94℃、5 min预变性;94℃、30 s,67℃、30 s,72℃、2 min,每个循环退火温度降1℃,共10个循环;后为25个循环94℃、30 s,57℃、30 s,72℃、2 min扩增;最后加72℃10 min延伸。巢式PCR第2轮使用CPR-32、QI引物,以1μL稀释20倍的第1轮PCR产物为模版,从66℃开始进行降落PCR反应。PCR产物使用胶回收试剂盒(Axygen,杭州)回收,TA克隆连接至pMD18-T载体(Takara,大连),转化Trans1-T1E. coli感受态细胞(全式金,北京)。单克隆进行菌落PCR鉴定,阳性菌株测序。
|
|
表 1 实验中使用的引物 Table 1 Primers used in this study |
5’RACE利用SMARTer RACR 5’/3’试剂盒(Clontech,日本),依据其说明书进行5’RACE的反转录,得到相应的第一链cDNA。根据试剂盒说明书使用引物CPR-51、CPR-52分别进行第1轮PCR和巢式第2轮PCR。PCR产物胶回收,In-Fusion克隆至pRACE载体中,转化。菌落PCR验证单克隆,阳性菌落测序。
根据3’RACE及5’RACE测序拼接结果设计引物CPR-1、CPR-2,PCR并测序验证。利用转录组数据,在茯苓的基因组数据[17]中找到PcCPR基因位置,在其上下游设计引物CPR-g1、CPR-g2。以提取的茯苓DNA为模版,克隆PcCPR基因。PCR体系:在GC buffer I中加入0.4μmol/L dNTPs,0.2μmol/L CPR-g1,0.2μmol/L CPR-g2,1.5 U LA Taq酶,1μL DNA模版,共25μL体系。反应程序:95℃预变性1 min;98℃变性20 s;57℃退火30 s;68℃延伸7 min,且延伸时间每个循环加10 s;共35个循环反应,后加72℃反应15 min。
1.2.3 PcCPR基因的生物信息学分析使用DNAMAN 8软件对3’和5’RACE得到的基因序列进行拼接,得到基因全长mRNA序列。利用NCBI BlastX比对,以确定得到的为PcCPR基因,并得到其开放阅读框(ORF)。利用DNAMAN 8翻译得到PcCPR蛋白质氨基酸序列。从GenBank下载真菌CPR蛋白序列,使用MAFFT v7,在默认参数下进行序列多重比对,比对结果使用MEGA 6进行系统发育分析。使用邻接法(Neighbor-Joining,NJ),Poisson模型构建系统发育树,建树可靠性使用Bootstrap,重复1 000次进行验证。
利用GSDS 2.0(Gene Structure Display Server,http://gsds.cbi.pku.edu.cn/)在线分析PcCPR的基因结构。使用ExPASy-ProtParam tool(http://web.expasy.org/protparam/)分析PcCPR蛋白的基本理化性质,应用ExPASy-ProtScale(http://web.expasy.org/protscale/)分析蛋白质氨基酸的亲疏水性。PcCPR蛋白质信号肽预测采用SignalP 4.1 server(http://www.cbs.dtu.dk/services/SignalP/);跨膜区预测采用TMHMM Server v. 2.0(http://www.cbs.dtu.dk/services/TMHMM/);蛋白亚细胞定位预测则采用ProtComp v. 9.0(http://linux1.softberry.com/berry.phtml?topic=protcompan & group=programs & subgroup=proloc)进行。应用I-TASSER(http://zhanglab.ccmb.med.umich.edu/I-TASSER/)进行蛋白质三级模拟。应用NCBI的CDD(Conserved Domain Database)数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行蛋白质保守结构域分析。
2 结果与分析 2.1 PcCPR基因的克隆利用茯苓转录组数据中注释为cytochrome P450 oxidoreductase的Unigene序列直接设计引物进行3’RACE及5’RACE,结果见图 1。3’RACE得到1 647 bp含有17 bp Poly(A)尾的序列;5’RACE得到1 002 bp序列。使用DNAMAN拼接得到2 514 bp的基因全长mRNA序列,并通过基因全长序列PCR测序验证正确。通过Blast确认所得为CPR基因序列,并将其命名为PcCPR。利用分离自美国弗罗里达州阿拉楚瓦郡的茯苓单孢培养菌株MD-104 SS10的基因组测序结果设计的引物扩增得到5 292 bp的PcCPR基因的基因组DNA序列(图 1-D)。将PcCPR基因的mRNA及基因组DNA序列提交至Genbank,获得Genbank登录号分别为KP768251、KP896487。利用GSDS 2.0在线工具分析PcCPR基因结构,如图 2所示,其含有4个外显子、3个内含子。PcCPR基因含2 199 bp的编码区(CDS),编码732个氨基酸;含有80 bp的5’非编码区(5’UTR)、218 bp的3’非编码区(3’UTR)。
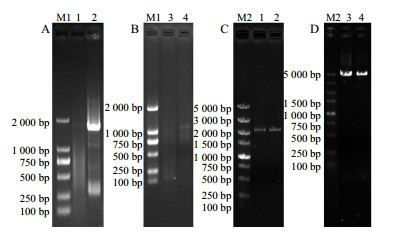
|
图 1 PcCPR基因扩增产物电泳结果 Fig.1 Agarose gel electrophoretic analysis of PCR products of PcCPR gene |

|
图 2 PcCPR基因结构 Fig.2 Gene structure of PcCPR gene |
2.2 PcCPR蛋白基本性质预测
利用克隆得到的PcCPR mRNA序列,翻译得到PcCPR的氨基酸序列。使用ExPASy-ProtParam tool预测PcCPR蛋白的性质,见表 2。预测该蛋白为相对分子质量81 147的蛋白,等电点(pⅠ)为5.39,不稳定系数(Ⅱ)40.02,平均疏水性(grand average of hydropathy,GRAVY)为−0.337。根据工具定义,不稳定系数(Instability index,Ⅱ)值大于40时,蛋白预测为不稳定,小于40时为稳定蛋白;GRAVY值范围为−2~2,正值表明此蛋白为疏水蛋白,负值表明为亲水蛋白。因此,PcCPR为不稳定的亲水蛋白。此外,利用ProtScale工具预测蛋白一级结构上的亲疏水性,使用默认的Hphob./Kyte & Doolittle标度打分,窗口大小(window size)为9,线性加权模型(linear weight variation model)。从预测结果(图 3)可知,PcCPR在一级结构上,多数区域的打分值为负,即表现亲水性,其结果与ExPASy-ProtParam tool预测结果一致。其中,第11位具有最高正值3.122,疏水性最强;第195位具有最低负值−2.967,亲水性最强。
|
|
表 2 PcCPR理化性质预测 Table 2 Predicted physicochemical properties of PcCPR |
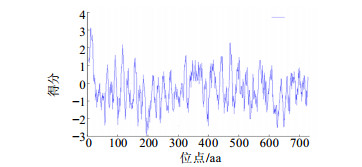
|
图 3 PcCPR氨基酸亲水性/疏水性预测 Fig.3 Predicted hydrophobicity/hydrophilicity of the amino acid sequence of PcCPR |
2.3 PcCPR信号肽预测
使用在线工具SignalP 4.1 server预测PcCPR的N端信号肽,使用默认参数设置,预测结果如图 4所示。预测结果显示,在第18位氨基酸有最高的C分值(原始剪切位点的分值,raw cleavage site score)0.325,即蛋白可能在此位置被切割;而最大S分值(0.627,信号肽分值,signal peptide score)在第1位氨基酸,蛋白第1~17位氨基酸的平均S分值为0.387;几何平均分C值与S分值打分的斜率得到的Y分值(combined cleavage site score)最大在第18位氨基酸,为0.342。对第1~17位氨基酸平均S分值及最大Y分值加权平均后得到的D分值(discrimination score)为0.360,小于设定阈值0.500。因此,预测PcCPR不含有信号肽,说明此蛋白为非分泌蛋白且不具有信号识别功能。
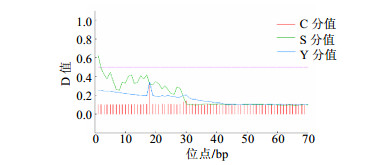
|
图 4 PcCPR信号肽预测 Fig.4 Signal peptide prediction of PcCPR |
2.4 PcCPR跨膜区预测
使用TMHMM Server v. 2.0预测PcCPR的跨膜区,结果(图 5)表明PcCPR有一段跨膜螺旋区,即蛋白的第7~22位氨基酸。蛋白第1~6位氨基酸在膜内侧,第23~732位氨基酸在膜外侧。

|
图 5 PcCPR跨膜区预测 Fig.5 Transmembrane helices prediction of PcCPR |
2.5 PcCPR亚细胞定位预测
ProtComp 9.0是一种结合多种方法对蛋白进行亚细胞定位预测的软件。通过直接对比已知亚细胞定位的同源性蛋白,得出总分为10分的打分结果LocDB;而对比数据库中具有有力理论推测定位的蛋白结果,则得到总分为5分的打分结果(PotLocDB);基于神经网络分析预测的打分则定义为总分3分的Neural Nets分值;基于序列与数据库五聚体分布对比计算打分结果得到总分5分的Pentamers分值;最后,对这4项打分进行加权后,得到总分为10分的最终结果(Integral)。对PcCPR使用ProtComp 9.0进行亚细胞定位预测,结果如表 3所示。预测结果表明,PcCPR应定位于内质网上,其软件综合打分结果最高,为9.90。
|
|
表 3 PcCPR亚细胞定位预测 Table 3 Sub-cellular location prediction of PcCPR |
2.6 PcCPR三级结构模拟
I-TASSER是一款蛋白高级结构及功能预测软件,能利用蛋白序列模拟预测出高质量、高分辨的蛋白3D结构。利用I-TASSER对PcCPR的高级结构进行模拟预测,模型结果利用Swiss-PdbViewer 4.1.0软件查看。模型预测结果如图 6所示,PcCPR蛋白的空间结构以螺旋和无规则卷曲为主。模型预测结果与PDB数据库中酿酒酵母(2bf4A)、褐家鼠(1ja1A、1tllA)CPR蛋白的结构相似度高。
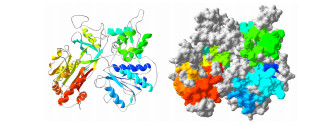
|
图 6 应用I-TASSER模拟PcCPR蛋白三级结构 Fig.6 Predicted three-dimensional structure of PcCPR with I-TASSER |
2.7 PcCPR保守结构域分析
PcCPR的保守结构域分析预测使用NCBI中的CDD数据库(Conserved Domain Database),结果如图 7所示。该蛋白N端第65~208位与NADPH依赖的FMN还原酶超家族(FMN_red superfamily)蛋白保守相似,第264~731位氨基酸的C端则与类似铁氧还蛋白还原酶超家族(FNR_like superfamily)蛋白保守相似;属于CysJ多保守域蛋白。结果显示,PcCPR蛋白的第264~731位极相似于NADPH cytochrome p450 reductase(CYPOR)保守域,且在此区域中预测出多个FAD结合位点、NADP结合位点、催化残基等。
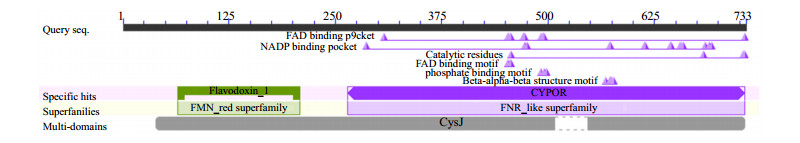
|
图 7 PcCPR保守结构域分析 Fig.7 Conserved domain prediction of PcCPR |
2.8 PcCPR系统进化分析
从Genbank中下载不同分类下的真菌的18条CPR蛋白质序列,以及黏菌盘基网柄菌的序列。使用BioEdit 7对PcCPR蛋白与其他物种蛋白进行同源性分析显示(表 4),PcCPR氨基酸序列与同属于担子菌亚门的黄孢原毛皮革菌、异孔异担子菌、毛韧革菌的序列相似度较高,相似性分别为91%、87%、86%;序列一致性为82%、79%和76%;而与黑曲霉、裂殖酵母等子囊菌的CPR蛋白的相似度相对较低为54%~60%,序列一致性为37%~44%;与分类为黏菌的盘基网柄菌的相似为49%,一致性为32%。对这20条蛋白序列使用MAFFT软件进行多序列比对。使用MEGA软,采用NJ法构建系统发育树。结果显示(图 8),PcCPR蛋白与黄孢原毛皮革菌中该蛋白的亲缘关系最近,同时与另2种担子菌异孔异担子菌、毛韧革菌聚为一支。这与它们的分类地位一致,茯苓与黄孢原毛皮革菌同属于多孔菌目(Polyporales);而后两者则属于红菇目(Russulales)。不仅如此,真菌CPR蛋白的聚类结果与其分类基本一致。
|
|
表 4 PcCPR与不同真菌CPR蛋白序列一致性及相似性 Table 4 Identities and similarities between PcCPR and CPR proteins from different fungi |
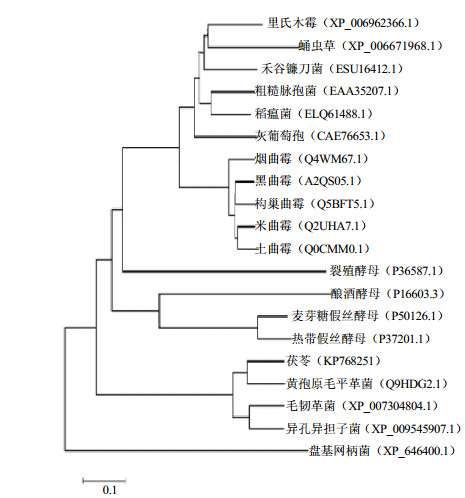
|
图 8 不同真菌CPR蛋白系统发育关系 Fig.8 Phylogenetic relationships among CPR proteins from different fungi |
3 讨论
在真核生物体内,CPR是唯一必需的中间体黄素蛋白,是P450s的重要组成部分[3],而后者因其参与了生物体的许多重要代谢途径,在各物种里均成为了研究热点。CPR基因已在许多动物、植物、酵母中克隆出来,而在丝状真菌仅有黄孢原毛皮革菌、黑曲霉等少量报道[1, 3, 18],PcCPR基因尚未见报道。
本研究从茯苓中克隆出PcCPR基因,基因cDNA全长2 514 bp,其中2 199 bp的编码区编码732个氨基酸,与黄孢原毛皮革菌的CPR同源性最高。同时获得相应的含有上下游序列的基因组基因序列5 292 bp,PcCPR基因的结构与报道的近缘种黄孢原毛皮革菌相似,均有3个内含子[18]。与分离自美国的茯苓菌株MD-104 SS10的高通量测序结果对比分析,基因组基因的序列一致性为93.56%,MD-104 SS10菌株基因序列长度增加115 bp,主要是在基因上游约−1 030 bp位置多出一段78 bp的序列;基因编码区序列一致性为98.59%,在30个位点有31 bp的碱基差异,这些碱基差异造成编码蛋白的共7个氨基酸的变异。
生物信息学分析结果显示,PcCPR是一个相对分子质量为81 147,PI为5.39的不稳定性亲水蛋白。蛋白为非分泌蛋白,且不具有信号识别功能。蛋白的第7~22位氨基酸被预测为跨膜区,这与报道的黄孢原毛平革菌的前22个氨基酸为跨膜区的结果相似[18]。同时,预测出蛋白定位于内质网上,这与一般报道的CPR蛋白的定位结果一致;且研究表明这种定位是P450s功能所必需的,若将N端锚定序列去除,CPR蛋白不能定位在膜上则CPR不能支撑P450反应,只能向细胞色素C、小分子化合物等提供电子[2]。对蛋白的亲疏水性、信号肽及跨膜区的预测也支持了蛋白定位于膜上的预测。
CPR蛋白的活性需要FMN和FAD 2个辅因子才能从底物NADPH获取并传递电子。PcCPR蛋白预测出多个FAD、NADP的结合位点,这些位点在模拟的蛋白三级结构空间上是各自相邻近的。并且预测得到2个黄素结合的保守结构域,分别与FMN还原酶超家族、类似铁氧还蛋白还原酶(ferredoxin NADP oxidoreductase,FNR)超家族保守相似,其中FMN与前者结合,FAD结合至后者上。这一预测结果与学者研究认为的CPR基因是由2个分别编码黄素氧还蛋白(flavodoxin,Fld)和FNR基因融合而成的结果相一致[2, 18]。
综上所述,本研究首次从茯苓中克隆出PcCPR基因,通过各种生物信息学分析方法对蛋白序列进行预测和分析,为深入研究该酶提供基础及参考。同时,CPR在细胞色素P450酶系中具有关键作用,也为研究茯苓细胞色素P450酶系奠定基础。
| [1] | Zhao C Q, Tang T, Feng X Y, et al. Cloning and characterisation of NADPH-dependent cytochrome P450 reductase gene in the cotton bollworm, Helicoverpa armigera[J]. Pest Manage Sci , 2014, 70 (1) :130–139. DOI:10.1002/ps.2013.70.issue-1 |
| [2] | Pandey A V, Fluck C E. NADPH P450 oxidoreductase: structure, function, and pathology of diseases[J]. Pharm Therap , 2013, 138 (2) :229–254. DOI:10.1016/j.pharmthera.2013.01.010 |
| [3] | Liu D, Zhou X J, Li M, et al. Characterization of NADPH-cytochrome P450 reductase gene from the cotton bollworm, Helicoverpa armigera[J]. Gene , 2014, 545 (2) :262–270. DOI:10.1016/j.gene.2014.04.054 |
| [4] | Riddick D S, Ding X X, Wolf C R, et al. NADPH-cytochrome P450 oxidoreductase: roles in physiology, pharmacology, and toxicology[J]. Drug Metab Dispos , 2013, 41 (1) :12–23. DOI:10.1124/dmd.112.048991 |
| [5] | Wang Y Z, Zhang J, Zhao Y L, et al. Mycology, cultivation, traditional uses, phytochemistry and pharmacology of Wolfiporia cocos (Schwein.) Ryvarden et Gilb.: A review[J]. J Ethnopharmacol , 2013, 147 (2) :265–276. DOI:10.1016/j.jep.2013.03.027 |
| [6] | Wang W H, Dong H J, Yan R Y, et al. Comparative study of lanostane-type triterpene acids in different parts of Poria cocos (Schw.) Wolf by UHPLC-Fourier transform MS and UHPLC-triple quadruple MS[J]. J Pharm Biomed Anal , 2015, 102 :203–214. DOI:10.1016/j.jpba.2014.09.014 |
| [7] | Xia B, Zhou Y, Tan H S, et al. Advanced ultra-performance liquid chromatography-photodiode array-quadrupole time-of-flight mass spectrometric methods for simultaneous screening and quantification of triterpenoids in Poria cocos[J]. Food Chem , 2014, 152 :237–244. DOI:10.1016/j.foodchem.2013.11.151 |
| [8] | Paddon C J, Westfall P J, Pitera D J, et al. High-level semi-synthetic production of the potent antimalarial artemisinin[J]. Nature , 2013, 496 (7446) :528–532. DOI:10.1038/nature12051 |
| [9] | Seki H, Ohyama K, Sawai S, et al. Licorice beta-amyrin 11-oxidase, a cytochrome P450 with a key role in the biosynthesis of the triterpene sweetener glycyrrhizin[J]. PNAS , 2008, 105 (37) :14204–14209. DOI:10.1073/pnas.0803876105 |
| [10] | Seki H, Sawai S, Ohyama K, et al. Triterpene functional genomics in licorice for identification of CYP72A154 involved in the biosynthesis of glycyrrhizin[J]. Plant Cell , 2011, 23 (11) :4112–4123. DOI:10.1105/tpc.110.082685 |
| [11] | Guo J, Zhou Y J, Hillwig M L, et al. CYP76AH1 catalyzes turnover of miltiradiene in tanshinones biosynthesis and enables heterologous production of ferruginol in yeasts[J]. PNAS , 2013, 110 (29) :12108–12113. DOI:10.1073/pnas.1218061110 |
| [12] | Chen S L, Xu J, Liu C, et al. Genome sequence of the model medicinal mushroom Ganoderma lucidum[J]. Nat Commun , 2012, 3 :913. DOI:10.1038/ncomms1923 |
| [13] | Syed K, Shale K, Pagadala N S, et al. Systematic identification and evolutionary analysis of catalytically versatile cytochrome p450 monooxygenase families enriched in model basidiomycete fungi[J]. PLoS One , 2014, 9 (1) :e86683. DOI:10.1371/journal.pone.0086683 |
| [14] | Ide M, Ichinose H, Wariishi H. Molecular identification and functional characterization of cytochrome P450 monooxygenases from the brown-rot basidiomycete Postia placenta[J]. Arch Microbiol , 2012, 194 (4) :243–253. DOI:10.1007/s00203-011-0753-2 |
| [15] | Shu S H, Chen B, Zhou M C, et al. De novo sequencing and transcriptome analysis of Wolfiporia cocos to reveal genes related to biosynthesis of triterpenoids[J]. PLoS One , 2013, 8 (8) :e71350. DOI:10.1371/journal.pone.0071350 |
| [16] | Scotto-lavino E, Du G, Frohman M A. 3' end cDNA amplification using classic RACE[J]. Nat Prod , 2006, 1 (6) :2742–2745. |
| [17] | Floudas D, Binder M, Riley R, et al. The Paleozoic origin of enzymatic lignin decomposition reconstructed from 31 fungal genomes[J]. Science , 2012, 336 (6089) :1715–1719. DOI:10.1126/science.1221748 |
| [18] | Yadav J S, Loper J C. Cytochrome P450 oxidoreductase gene and its differentially terminated cDNAs from the white rot fungus Phanerochaete chrysosporium[J]. Curr Genet , 2000, 37 (1) :65–73. DOI:10.1007/s002940050010 |
 2016, Vol. 47
2016, Vol. 47


