2. 天津药物研究院 中药研究部, 天津 300193 ;
3. 天津化学化工协同创新中心合成生物学平台, 天津 300072
2. Traditional Chinese Medicine Research Department, Tianjin Institute of Pharmaceutical Research, Tianjin 300193, China ;
3. SynBio Research Platform, Collaborative Innovation Center of Chemical Science and Engineering (Tianjin), Tianjin 300072, China
木脂素(lignans)是桃儿七、鬼臼、连翘、五味子、牛蒡子、厚朴、细辛等中药的活性成分,具有多种药理活性和重要的应用价值[1-2]。木脂素类成分松脂酚具有高效的抗炎作用,开环异落叶松脂醇和罗汉松脂酚能够在哺乳动物体内转化为肠内脂和肠二醇,具有雌激素作用。五味子甲素、五味子乙素及五味子丙素具有抗氧化、保护肝脏和解毒、增强抗癌药的敏感性以及中枢神经镇静作用,并能改善慢性肝炎患者的肝功能异常。桃儿七、鬼臼、八角莲、山荷叶等鬼臼类植物具有抗肿瘤、抗菌消炎、抗病毒等药理活性[3],而木脂素类成分鬼臼毒素是半合成抗癌药物依托泊苷的原料。因此木脂素在药品、保健品和食品等领域有广阔的应用前景。
目前木脂素主要通过植物提取制备,不仅受限于生长时间和种植地域,而且已经造成鬼臼植物资源枯竭、濒临灭绝。随着系统生物学、代谢工程和合成生物学的发展,天然产物工程取得突破,如在工程大肠杆菌中合成了紫杉醇前体紫杉二烯[4]、丹酚酸前体丹参素[5],在工程酵母中合成了青蒿素前体青蒿酸[6]、人参皂苷元[7]、次丹参酮[8]等。近年来,木脂素生物合成途径解析方面取得了重要进展,使人们越来越重视通过生物工程改造,在模式植物或微生物中合成木脂素。本文综述了木脂素生物合成途径解析及其代谢工程和合成生物学的国内外研究进展,并对未来的发展趋势进行了讨论。
1 木脂素的生物合成途径解析木脂素的生物合成包括3个阶段,首先是由苯丙烷途径合成木脂素的前体松柏醇,然后由松柏醇合成结构多样的木脂素,最后木脂素被糖基化修饰,形成木脂素糖苷,在植物细胞和组织中积累贮存。
1.1 松柏醇的生物合成途径松柏醇(coniferyl alcohol)是合成木脂素的前体,由苯丙氨酸转化而来(图 1)[9]。苯丙氨酸解氨酶(PAL)催化苯丙氨酸脱氨,生成反式肉桂酸,再经肉桂酸-4-羟化酶(C4H)催化生成香豆酸。咖啡酰辅酶A的生物合成有2种机制:(1)4-香豆酰辅酶A连接酶(4CL)催化香豆酸生成香豆酰辅酶A,再由香豆酸-3-羟化酶(C3H)催化C-3位羟基化,生成咖啡酰辅酶A;(2)以香豆酰辅酶A为底物,由莽草酸羟基转移酶(CST)或奎宁酸羟基肉桂酰转移酶(CQT)催化,生成香豆酰莽草酸或香豆酰奎尼酸,继而被C3H催化,分别生成羟基化产物咖啡酰莽草酸和咖啡酰奎尼酸中间体,再由CST或CQT催化转变为咖啡酰辅酶A。
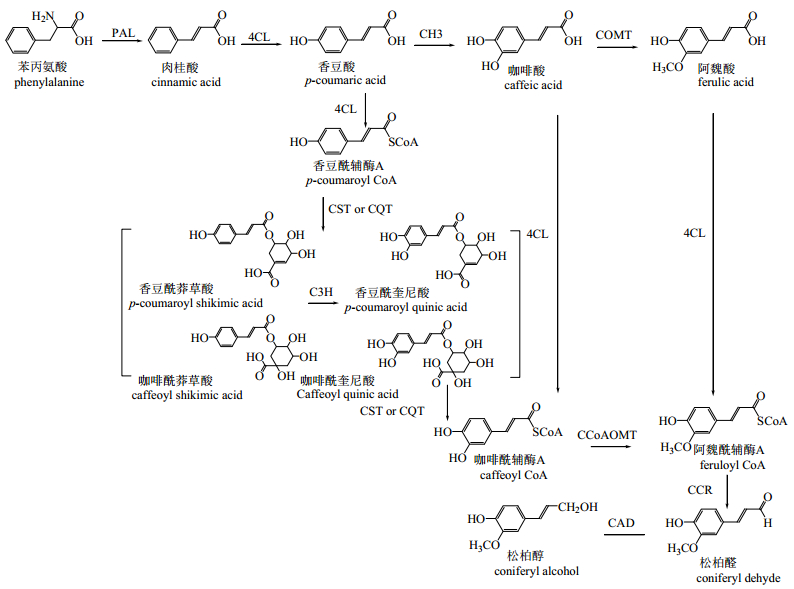
|
图 1 松柏醇的生物合成途径 Fig.1 Biosynthetic pathway of coniferyl alcohol |
咖啡酰辅酶A在咖啡酰辅酶A-O-甲基转移酶(CCoAOMT)催化下发生甲基化反应,生成阿魏酰辅酶A。由于C3H、4CL、甲基转移酶的底物较为宽泛,从香豆酸到阿魏酰辅酶A存在其他机制,即香豆酸先在C3H的催化下生成咖啡酸,咖啡酸由4CL催化生成咖啡酰辅酶A再进行甲基化反应;或者咖啡酸-O-甲基转移酶(COMT)催化咖啡酸,生成甲基化产物阿魏酸,阿魏酸再由4CL催化生成阿魏酰辅酶A。最后,阿魏酰辅酶A经肉桂酰辅酶A还原酶(CCR)还原为松柏醛,再由肉桂醇脱氢酶(CAD)的催化生成松柏醇。
松柏醇也是合成细胞壁中木质素(lignin)的单体之一,其生物合成途径在维管植物中高度保守,已在松、杉、柏、杨、拟南芥等植物中克隆了部分基因。在桃儿七、鬼臼、连翘、亚麻等植物也存在上述途径,但相关基因及其功能研究鲜见报道。鬼臼转录组[10]和亚麻基因组[11]已测序,有望从含木脂素植物中克隆和鉴定出松柏醇生物合成途径的基因。
1.2 松脂酚的生物合成途径松脂酚(pluviatolide)是由2分子松柏醇单体聚合而成,是木脂素生物合成的限速步骤。该反应是自由基加成反应,但酶体系还没有完全鉴定出来。虽然在连翘、盾叶鬼臼、亚麻等多种植物中发现了同化蛋白氧化酶(DIR),其参与了产物的立体选择,决定了产物松脂酚的构型,但松脂酚合成酶及其编码基因至今未知。在芝麻属植物中,细胞色素P450酶(CYPs)CYP81Q1催化松脂酚生成芝麻素。在连翘和鬼臼等植物中,松脂酚经落叶松脂醇还原酶(PLR)催化,先后形成落叶松脂醇和开环异落叶松脂醇,后者再经开环异落叶松脂醇脱氢酶(SDH)催化生成罗汉松脂酚(图 2)。DIR、PLR、SDH 3个基因的转录表达被茉莉酮酸甲酯、脱落酸所诱导,这与鬼臼毒素的生物合成的诱导模式相一致。
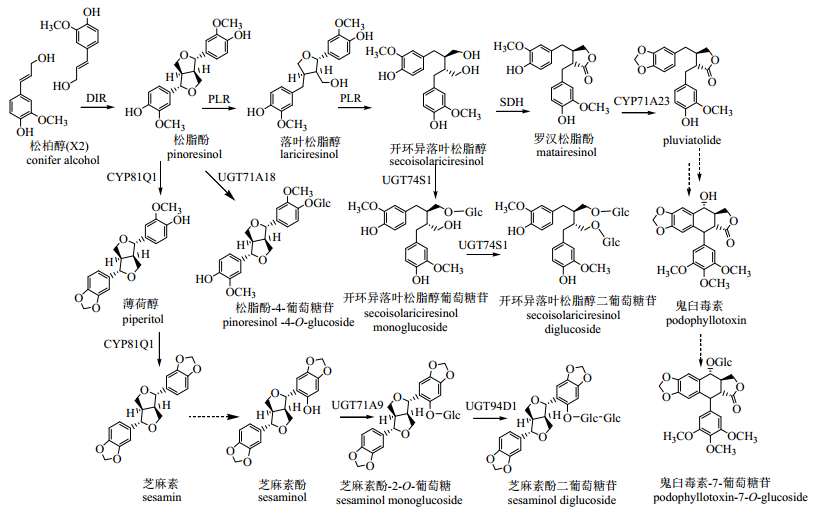
|
图 2 木脂素及其糖苷的生物合成途径 Fig.2 Biosynthetic pathways of lignans and their glycosides |
PLR基因已经在连翘、鬼臼、板蓝根中先后被克隆,它们是同源基因[12-15]。盾叶鬼臼PLR与连翘和板蓝根PLR氨基酸序列的同源性分别为84%和72%,在分子进化上是高度保守的,归为同一类。酶学分析表明,盾叶鬼臼PLR(Km=19.8μmol/L)和连翘PLR(Km=23μmol/L)对松脂酚的底物亲和性接近,而板蓝根PLR1对于松脂酚亲和性较低(Km=65.4μmol/L);但对落叶松脂醇而言,板蓝根PLR1的亲和性(Km=2.5μmol/L)远远高于盾叶鬼臼PLR(Km=37.3μmol/L)和连翘PLR(Km=121μmol/L)[15],这可能有利于盾叶鬼臼和连翘合成更多的落叶松脂醇。尽管在2001年从连翘和盾叶鬼臼中发现了SDH酶,但在2014年才从盾叶鬼臼P. pleianthum Hance中克隆了SDH基因[14],盾叶鬼臼的SDH底物亲和性(Km=231μmol/L)比美洲鬼臼P. peltatum L.的SDH低(Km=160μmol/L)。
基于二代测序的转录组分析,Marques等[16]从桃儿七中鉴定了CYP719A23,从盾叶鬼臼中鉴定了CYP719A24,二者是同源酶,参与了罗汉松脂酚到松脂酚的合成。但这2个酶是催化生物碱类的亚甲基二氧桥的形成,而芝麻种属的CYP81Q1是催化双四氢呋喃类木脂素的亚甲基二氧桥的形成,它们不属于同一个家族。
1.3 依托泊苷元的生物合成途径鬼臼毒素是半合成依托泊苷和替尼泊苷的原料,虽然受到高度关注和广泛研究,由于受研究手段和方法的限制,长期以来鬼臼毒素的生物合成途径没有实质性进展。2015年,Lau等[17]采用前体饲喂和候选基因共表达策略,以烟草叶片为转基因表达系统,把液质联用分析代谢物组和二代转录组测序相结合,完成了依托泊苷元合成途径的编码基因的鉴定。首先对盾叶鬼臼的叶、根、茎进行转录组测序和基因表达的差异分析,并且与NCBI数据库已知鬼臼毒素生物合成基因和拟南芥蛋白质组数据库进行BLAST比对,初步筛选出4个O-甲基转移酶(OMT1-4)、12个CYPs和1个2-酮戊二酸/Fe(II)依赖的双加氧酶(2-ODD),它们可能参与了依托泊苷元的生物合成。在4个候选OMT基因中,发现只有OMT3和CYP719A23共表达时,才能消耗底物松脂酚,生成5′-去甲氧基-亚太因,由此确定OMT3是催化松脂酚的甲基转移酶。类似地,鉴定出CYP71CU1催化5′-去甲氧基亚太因生成5′-去甲氧基亚太因,由OMT1进一步催化生成亚太因,由2-ODD催化亚太因生成去氧鬼臼毒素,CYP71BE54催化生成4′-去甲基去氧鬼臼毒素,CYP82D61催化生成4′-去甲基表鬼臼毒素(图 3)。
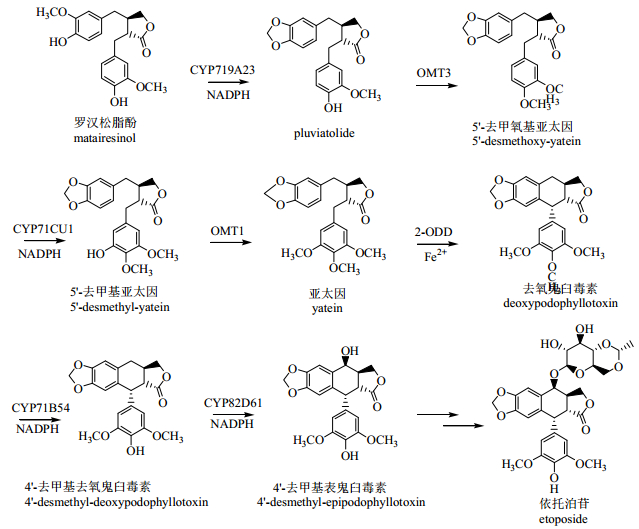
|
图 3 4′-去甲基表鬼臼毒素的生物合成途径 Fig.3 Biosynthetic pathway of 4′-desmethyl-epipodophyllotoxin |
该研究鉴定的鬼臼毒素生物合成途径,与在盾叶鬼臼中推测的途径不完全相同。在以前的研究中,认为盾叶鬼臼中去氧鬼臼毒素7-羟化酶催化去氧鬼臼毒素,生成鬼臼毒素;而去氧鬼臼毒素6-羟化酶催化生成β-盾叶鬼臼毒素。但在烟草叶片的实验中,没有检测到鬼臼毒素,而是检测到去甲基-去氧鬼臼毒素和4′-去甲基表鬼臼毒素。去氧鬼臼毒素7-羟化酶基因和6-羟化酶基因仍然有待挖掘和鉴定,才能确定鬼臼毒素的生物合成途径。
1.4 木脂素的糖基化途径大部分木脂素是以糖基化形式存在于植物中,由UDP-依赖的糖基转移酶(the uridine diphosphate (UDP)-sugar dependent glycosyltransferases,UGT)催化生成木脂素糖苷。直到2008年以后,才从芝麻、亚麻等植物中克隆并研究了木脂素糖基化基因和编码酶(图 2)。在芝麻中,UGT71A9利用UDP-葡萄糖作为糖基供体,把1分子葡萄糖转移到芝麻素酚的2-OH处,生成芝麻素酚-2-O-葡萄糖;而UGT94D1则特异性参与芝麻素酚二葡萄糖苷的合成[18]。在连翘中,UGT71A18可促进落叶松脂醇糖苷的积累量增加[19]。亚麻的UGT74S1利用UDP-葡萄糖,催化生成开环异落叶松脂醇葡萄糖苷和开环异落叶松脂醇二葡萄糖苷[20]。亚麻中鬼臼毒素的2种主要产物是6-甲基鬼臼毒素和鬼臼毒素7-葡萄糖苷,在亚麻细胞悬浮培养中检测到鬼臼毒素7-O-糖基化和6-O-甲基化活性的酶[21-22],但其编码基因还未鉴定出来。
2 代谢工程合成木脂素随着木脂素生物合成途径的解析,在连翘、亚麻、板蓝根等植物中进行代谢工程研究,建立了木脂素合成的转基因植物,以期提高木脂素的量。
2.1 转基因连翘连翘的茎、叶、花、果等组织可合成和积累丰富的木脂素,如松脂醇、连翘脂素、开环异落叶松脂醇、罗汉松脂酚和牛蒡苷元,是理想的木脂素合成平台。转基因连翘的研究主要是由日本Satake课题组进行。他们通过代谢工程,建立了合成木脂素的连翘悬浮细胞系。2009年,他们采用反义RNA
技术干扰PLR基因表达,构建了单转基因的连翘细胞系(U18i),阻断了罗汉松脂酚的合成,使松脂酚产量提高了2倍,并使松柏醇糖苷产量提高20倍[23]。随后再过表达芝麻的CYP81Q1,构建了双转基因的连翘细胞系(CPi-Fk,即抑制表达PLR基因和过表达芝麻的CYP81Q1),首次在连翘中合成了芝麻素[23]。2015年,该课题组[24]进一步干扰抑制松脂醇糖基转移酶基因(UGT71A18)表达,建立稳定的三转基因连翘细胞系(U18i-CPi-Fk,即抑制表达松脂醇糖基酶基因和PLR基因、过表达芝麻CYP81Q1),使松脂醇和芝麻素的量分别提高5倍和1.4倍。
2.2 转基因亚麻亚麻中含有多种木脂素,同时能合成鬼臼毒素及其衍生物,从而备受关注。在亚麻细胞培养体系中,鬼臼毒素可占细胞干质量的0.6%~1.7%。Lorenc-Kukula等[25]把马铃薯的糖基转移酶基因(SsGT1)稳定转化到亚麻中,使开环异落叶松脂醇的量提高2倍多。同时,该转基因亚麻种子的提取物具有治疗伤口和促进愈合,抑制大肠杆菌和金黄色葡萄球菌的作用[26],显示出良好的医药应用前景。Żuk等[27]将矮牵牛的3个黄酮合成酶基因(查尔酮合成酶基因、查尔酮异构酶基因和二氢黄酮还原酶基因)转化到到亚麻中,构建了稳定的转基因亚麻株系,开环异落叶松脂醇二葡萄糖苷的量增加2倍多,黄酮类化合物的量增加50%以上,抗氧化活性增加了4~6倍,亚麻的品质得到明显改善。
2.3 转基因板蓝根板蓝根是常用的治疗感冒、发热和流感的中药,其中落叶松脂醇是其重要的活性成分。2015年,Xiao等[15]采用RNA干扰沉默策略,从板蓝根的3个PLR基因中鉴定出PLR1(负责落叶松脂醇的生物合成)。为了提高板蓝根中落叶松脂醇的量,他们用2个强的CaMV35S启动子驱动板蓝根PLR1基因的转录,转化到板蓝根的毛根中后,落叶松脂醇的量是野生型的6.3倍。该研究为板蓝根药材的品种改良和通过毛根培养系统生产木脂素探索了新途径。
3 合成生物学策略合成木脂素 3.1 工程大肠杆菌合成罗汉松脂酚大肠杆菌是生物制药领域应用最广泛的合成天然产物的宿主微生物,已经合成了紫杉醇、青蒿素等的前体。2014年,Kuo等[14]将盾叶鬼臼的PLR和SDH基因以3种不同方式组合,以松脂酚为前体,构建了合成罗汉松脂酚的工程菌。当PLR和SDH单独表达时,转化率仅为17.7%;PLR-SDH融合表达时,转化率为49.8%;SDH-PLR融合表达时,只形成了中间产物开环异落叶松脂醇,没有终产物罗汉松脂酚生成。可见,PLR-SDH的融合是这2个基因在大肠杆菌中的最佳表达方式,松脂酚的转化效率最高。
3.2 转基因烟草合成依托泊苷元前体依托泊苷是临床应用的重要抗肿瘤药物,鬼臼毒素是其半合成前体。目前,从鬼臼植物的根茎部提取是鬼臼毒素的主要制备方式,由于过度开采而导致鬼臼类植物濒临灭绝,对其资源造成极大破坏。Lau等[17]在解析了鬼臼毒素的生物合成途径的基础上,首次在烟草中实现了从松柏醇到依托泊苷元合成途径的创建和表达。采用Gibson组装技术,构建了多基因(PLR、SDH、CYP719A23、OMT3、CYP71CU1、2-ODD、OMT1、CYP82D61、CYP71BE54)串联的共表达载体。添加松柏醇前体,转基因烟草合成了4′-去甲基表鬼臼毒素。如果不表达CYP71BE54,则合成去氧鬼臼毒素,其量高于鬼臼叶愈伤组织培养。
用烟草表达鬼臼毒素代谢途径的基因,直接合成非天然木脂素4′-去甲基表鬼臼毒素,绕过了盾叶鬼臼的种植和鬼臼毒素的后续差向异构化、脱甲基化反应,减少了半合成反应步骤,提高了依托泊苷合成的经济性,展示出工业化应用的良好前景。
4 结语木脂素存在于多种药用植物中,目前已经确定了从松柏醇到4′-去甲基表鬼臼毒素的生物合成途径的主要基因,但途径不完整。克隆和鉴定木脂素途径中的未知基因、发现新的合成途径是未来的一个重要研究方向。例如,木脂素合成的第一步涉及的松脂酚合成酶基因、鬼臼毒素合成的最后几步基因以及木脂素生物合成的调控基因的研究都还处于空白;且仅有几个木脂素糖基化基因被克隆,糖基化途径几乎未知。除了鬼臼类结构的木脂素外,连翘脂素、牛蒡子素、五味子素、厚朴酚、细辛脂素等木脂素的生物合成研究鲜见报道。目前基因组测序已经发展到第3代测序技术,具有快速、高通量、数据产出量大、成本更低的优点,将加速植物基因组和转录组测序,与代谢物组学和蛋白质组学技术的结合,可为木脂素合成基因的挖掘和鉴定提供有力的技术支持。可以预见,将有更多种药用植物的木脂素生物合成途径被解析,为中药材鉴别和品质改良奠定基础。在转基因连翘、亚麻和板蓝根以及烟草中首次实现了异源合成依托泊苷的前体,展示出代谢工程和合成生物学在木脂素及其衍生物合成中的应用潜力。未来研究工作应主要集中在:(1)改良木脂素类中药材品质。在木脂素合成基因鉴定的基础上,通过代谢途径设计和工程组装[28],可重构木脂素合成途径。采用CRISPR-Cas技术,对基因组进行精细编辑[29],定点删除木脂素合成的支路途径,定点插入关键基因、主路途径或组装的新途径,从而培育无标记的环境安全的高木脂素产量的药用植物品种和细胞系,为中药现代化研究提供优质药材。(2)构建木脂素的人工细胞工厂。大肠杆菌和酿酒酵母等微生物、烟草和胡萝卜细胞等模式植物,具有生长快、易培养、基因组操作成熟等优点,已成为植物次级代谢产物合成的重要宿主[30]。按宿主生物的遗传密码偏向性,设计和优化木脂素合成途径,构建出高效合成木脂素的生物细胞工厂,通过微生物发酵或植物细胞培养,生产制备单一活性成分的木脂素。可望解决木脂素药原短缺的问题,从而保护中药资源,促进木脂素天然药物创新研究与开发。
| [1] | 张国良, 李娜, 林黎琳, 等. 木脂素类化合物生物活性研究进展[J]. 中国中药杂志 , 2007, 32 (20) :2089–2094. |
| [2] | 孙彦君, 王雪, 陈辉, 等. 四氢呋喃型木脂素类化合物研究进展[J]. 中草药 , 2013, 44 (21) :3067–3079. |
| [3] | 孙彦君, 李占林, 陈虹, 等. 鬼臼类植物化学成分和生物活性研究进展[J]. 中草药 , 2012, 43 (8) :1626–1633. |
| [4] | Ajikumar P K, Xiao W H, Tyo K E J, et al. Isoprenoid pathway optimization for taxol precursor overproduction in Escherichia coli[J]. Science , 2010, 330 (6000) :70–74. DOI:10.1126/science.1191652 |
| [5] | Yao Y F, Wang C S, Qiao J J, et al. Metabolic engineering of Escherichia coli for production of salvianic acid A via an artificial biosynthetic pathway[J]. Metab Eng , 2013, 19 (5) :79–87. |
| [6] | Paddon C J, Westfall P J, Pitera D J, et al. High-level semi-synthetic production of the potent antimalarial artemisinin[J]. Nature , 2013, 496 (7446) :528–532. DOI:10.1038/nature12051 |
| [7] | Yan X, Fan Y, Wei W, et al. Production of bioactive ginsenoside compound K in metabolically engineered yeast[J]. Cell Res , 2014, 24 (6) :770–773. DOI:10.1038/cr.2014.28 |
| [8] | Guo J, Zhou Y J J, Hillwigc M L, et al. CYP76AH1 catalyzes turnover of miltiradiene in tanshinones biosynthesis and enables heterologous production of ferruginol in yeasts[J]. Proc Natl Acad Sci USA , 2013, 110 (29) :12108–12113. DOI:10.1073/pnas.1218061110 |
| [9] | Vanholme R, Demedts B, Morreel K, et al. Lignin biosynthesis and structure[J]. Plant Physiol , 2010, 153 (3) :895–905. DOI:10.1104/pp.110.155119 |
| [10] | Bhattacharyya D, Sinha R, Hazra S, et al. De novo transcriptome analysis using 454 pyrosequencing of the himalayan mayapple, Podophyllum hexandrum[J]. BMC Genomics , 2013, 14 (1) :1594–1598. |
| [11] | Wang Z, Hobson N, Galindo L, et al. The genome of flax (Linum usitatissimum) assembled de novo from short shotgun sequence reads[J]. Plant J , 2012, 72 (3) :461–473. DOI:10.1111/tpj.2012.72.issue-3 |
| [12] | Dinkova-Kostova A T, Gang D R, Davin L B. (+)-Pinoresinol/(+)-lariciresinol reductase from Forsythia intermedia. Protein purification, cDNA cloning, heterologous expression and comparison to isoflavone reductase[J]. J Biol Chem , 1996, 271 (46) :29473–29482. DOI:10.1074/jbc.271.46.29473 |
| [13] | Wankhede D P, Biswas D K, Rajkumar S, et al. Expressed sequence tags and molecular cloning and characterization of gene encoding pinoresinol/lariciresinol reductase from Podophyllum hexandrum[J]. Protoplasma , 2013, 250 (6) :1239–1249. DOI:10.1007/s00709-013-0505-z |
| [14] | Kuo H J, Wei Z Y, Lu P C, et al. Bioconversion of pinoresinol into matairesinol by use of recombinant Escherichia coli[J]. Appl Environ Microbiol , 2014, 80 (9) :2687–2692. DOI:10.1128/AEM.03397-13 |
| [15] | Xiao Y, Ji Q, Gao S, et al. Combined transcriptome and metabolite profiling reveals that IiPLR1 plays an important role in lariciresinol accumulation in Isatis indigotica[J]. J Exp Bot , 2015, 66 (20) :6259–6271. DOI:10.1093/jxb/erv333 |
| [16] | Marques J V, Kim K W, Lee C, et al. Next generation sequencing in predicting gene function in podophyllotoxin biosynthesis[J]. J Biol Chem , 2013, 288 (1) :499–513. |
| [17] | Lau W, Sattely E S. Six enzymes from mayapple that complete the biosynthetic pathway to the etoposide aglycone[J]. Science , 2015, 349 (6253) :1224–1228. DOI:10.1126/science.aac7202 |
| [18] | Noguchi A, Fukui Y, Iuchi-Okada A, et al. Sequential glucosylation of a furofuran lignan, (+)-sesaminol, by Sesamum indicum UGT71A9 and UGT94D1 glucosyltransferases[J]. Plant J , 2008, 54 (3) :415–427. DOI:10.1111/j.1365-313X.2008.03428.x |
| [19] | Ono E, Kim H J, Murata J, et al. Molecular and functional characterization of novel furofuran-class lignan glucosyltransferases from Forsythia[J]. Plant Biotechnol , 2010, 27 (4) :317–324. DOI:10.5511/plantbiotechnology.27.317 |
| [20] | Ghose K, Selvaraj K, McCallum J, et al. Identification and functional characterization of a flax UDP-glycosyltransferase glucosylating secoisolariciresinol (SECO) into secoisolariciresinol monoglucoside (SMG) and diglucoside (SDG)[J]. BMC Plant Biol , 2014, 14 (1) :1–17. DOI:10.1186/1471-2229-14-1 |
| [21] | Kranz K, Petersen M. β-Peltatin 6-O-methyltransferase from suspension cultures of Linum nodiflorum[J]. Phytochemistry , 2003, 64 (2) :453–458. DOI:10.1016/S0031-9422(03)00196-1 |
| [22] | Berim A, Ebel R, Schneider B, et al. UDP-glucose: (6-methoxy) podophyllotoxin 7-O-glucosyltransferase from suspension cultures of Linum nodiflorum[J]. Phytochemistry , 2008, 69 (2) :374–381. DOI:10.1016/j.phytochem.2007.07.030 |
| [23] | Kim H J, Ono E, Morimoto K, et al. Metabolic engineering of lignan biosynthesis in Forsythia cell culture[J]. Plant Cell Physiol , 2009, 50 (12) :2200–2209. DOI:10.1093/pcp/pcp156 |
| [24] | Murata J, Matsumoto E, Morimoto K, et al. Generation of triple-transgenic Forsythia cellcultures as a platform for the efficient, stable, and sustainable production of lignans[J]. PLos One , 2015, 10 (12) :e0144519. DOI:10.1371/journal.pone.0144519 |
| [25] | Lorenc-Kukula K, Zuk M, Kulma A, et al. Engineering flax with the GT family 1 Solanum sogarandinum glycosyltransferase SsGT1 confers increased resistance to Fusarium infection[J]. J Agric Food Chem , 2009, 57 (15) :6698–6705. DOI:10.1021/jf900833k |
| [26] | Czemplik M, Kulma A, Bazela K, et al. The biomedical potential of genetically modified flax seeds overexpressing the glucosyltransferase gene[J]. BMC Complement Altern Med , 2012, 12 (4) :674–678. |
| [27] | Zuk M, Kulma A, Dyminska L, et al. Flavonoid engineering of flax potentiate its biotechnological application[J]. BMC Biotechnol , 2011, 11 (1) :92–95. DOI:10.1186/1472-6750-11-92 |
| [28] | 赵鹃, 王霞, 李炳志, 等. 合成生物学中的DNA组装技术[J]. 生命科学 , 2013, 25 (10) :983–992. |
| [29] | Shan Q W, Wang Y P, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nat Biotechnol , 2013, 31 (8) :686–688. DOI:10.1038/nbt.2650 |
| [30] | Luo Y Z, Li B Z, Liu D, et al. Engineered biosynthesis of natural products in heterologous hosts[J]. Chem Soc Rev , 2015, 44 (15) :5265–5390. DOI:10.1039/C5CS00025D |
 2016, Vol. 47
2016, Vol. 47


