甘草Glycyrrhiza uralensis Fisch.为豆科(Leguminosae)甘草属Glycyrrhiza L.多年生草本植物,是中国传统大宗补益中草药,药用部位是根及根茎。甘草具有祛痰止咳、清热解毒、抗癌和免疫调节等诸多功能,已被列入多国药典(《中国药典》2010年版、《日本药典》JP16、《美国药典》USP34和《欧洲药典》Ph.Eur.7)[1]。甘草主要分布于新疆、宁夏、内蒙古和甘肃等省区的干旱、半干旱的荒漠草原、沙漠边缘和黄土丘陵地带,根系发达,耐逆性强,具有防风固沙的功能,对于我国西北部荒漠地区生态环境的保护和改善具有重要意义[2]。
microRNA(miRNA)是一类在真核生物中发现的长度约为21 nt的单链非编码小分子RNA。最早发现的miRNA是秀丽隐杆线虫Caenorhabditis elegans的lin-4和let-7[3-4]。miRNA的成熟形式主要在细胞质中通过与靶基因的碱基互补配对,进而介导靶基因的降解或翻译抑制,在转录后水平对基因表达进行调控[5]。在植物中,miRNA通过负调控转录因子和其他重要的功能基因,广泛参与植物器官的生长发育、形态建成、逆境胁迫应答和次生代谢调控[6]。
目前已经鉴定了超过72种植物的7 385种miRNA,在每种植物中发现的miRNA种类数介于几十个到几百个之间[7]。整体说来,对于拟南芥、水稻、毛果杨等模式植物的miRNA研究较多,对于中草药等植物的研究相对较少。中草药在中国传统医药中具有重要地位。miRNA在植物体内发挥重要的调控作用,参与植物环境逆境应答以及次生代谢产物的生物合成,研究药用植物miRNA的生物学功能对于理解药用成分形成和积累的生态调控机制,培育高产优质的中药材具有重要意义。近年来,对药用植物miRNA的研究正在迅速开展起来,研究人员利用高通量测序、miRNA芯片、生物信息分析等方法研究了人参、毛地黄、半夏、千里光等中草药的miRNA[8-9]。但迄今为止,未见甘草miRNA的相关研究。本研究拟应用生物信息学工具,基于甘草转录本序列鉴定miRNA,并确定其靶基因,为进一步研究miRNA在甘草逆境应答和次生代谢途径调控中的作用奠定基础。
1 材料甘草种子来自内蒙古自治区鄂尔多斯市,经中央民族大学生命与环境科学学院刘博博士鉴定为甘草Glycyrrhiza uralensis Fisch.的根及根茎。
Trizol试剂购自美国英杰生命技术有限公司(Invitrogen),逆转录试剂和荧光定量PCR试剂盒购自天根生化科技(北京)有限公司,引物由生工生物工程(上海)股份有限公司合成。其余试剂均为分析纯。
2 方法 2.1 甘草核酸数据的获取和转录本数据拼接在NCBI网站(http://www.ncbi.nlm.nih.gov)下载甘草的高通量测序数据和EST序列。高通量数据登录号为DRR006519、DRR006520、DRR006521、DRR006522、DRR006523、DRR006524、DRR006525和DRR006526。表达序列标签(EST)数据有55 942条。数据过滤软件使用Trimmomatic(http://www.usadellab.org/cms/index.php?page=trimmomatic)[10]和solexaQA(http://solexaqa.sourceforge.net/)[11]。拼接软件使用Trinity(http://trinityrnaseq.github.io/)[12]和iAssembler(v1.3.2,http://bioinfo.bti.cornell.edu/tool/iAssembler/)[13],拼接采用默认参数进行。聚类去冗余软件使用CD-HIT(http://weizhong-lab.ucsd.edu/cd-hit/)[14]。
2.2 甘草miRNA的生物信息学预测整体预测流程参考文献方法[15]进行。简言之,首先从植物miRNA数据库(http://bioinformatics.c au.edu.cn/PMRD)中下载10 898个植物来源的miRNA茎环结构前体,去除不同物种miRNA之间的重复序列以及不可靠的数据后,剩余的4 835条miRNA前体序列作为预测甘草miRNA前体的参照序列与拼接得到的甘草转录本序列进行同源比对分析(Blastn),筛选出E<1e-6的转录本序列。这些转录本序列去除冗余后,用Mfold软件(http://mfold.rna.albany.edu/q=mfold/RNA-Folding-Form)[16]进行折叠,预测候选miRNA前体序列的二级结构,分析是否有miRNA成熟形式存在茎环结构上茎的部分,评价茎环结构的稳定性。miRNA前体的二级结构筛选标准参照Zhang等[17]的方法。
2.3 甘草根中miRNA的检测基于甘草的主要药用部位是根茎,选取培养了8周的甘草幼苗根部,用Trizol法提取根中的总RNA,并分别用Nano DropTM2000分光光度计、琼脂糖凝胶电泳验证总RNA的纯度、浓度、完整性。
随机选取生物信息学预测中的12条miRNA,并分别设计逆转录茎环引物及荧光定量PCR引物(表 1),利用stem-loop荧光定量PCR验证miRNA。
|
|
表 1 待验证的miRNAs及其对应引物 Table 1 Selected miRNAs and their corresponding primers |
2.4 靶基因的预测和GO分析
将全部靶基因序列比对到拟南芥蛋白数据,用拟南芥蛋白注释靶基因,以了解靶基因的生物学功能,截取标准为E<1e-5。使用agriGO( http://bioinfo.cau.edu.cn/agriGO/analysis.php)中的GO分析工具对靶基因进行功能分类和富集分析。
3 结果与分析 3.1 甘草转录本数据的拼接首先用Trimmomatic软件对Illumina HiSeq2000测序所得数据进行滤过;用solexaQA对EST数据进行滤过,获得去除测序接头、去除低质量数据、去除冗余序列的可用于拼接的数据。然后分别用Trinity和iAssembler混合拼接Illumina Heseq 2000和EST数据。最后将上述2部分拼接好的数据用CD-HIT合并聚类。
本研究拼接获得的unigene的平均长度为896 bp,略小于Ramilowski等[18]的拼接结果,但远大于Li等[19]的拼接结果。本研究的unigene的N50(1 493 bp)和拼接总长度(67 122 825 bp)明显大于其他2个拼接结果。总的来看,本研究获得的转录本数据覆盖度最高,质量较好,可以用于miRNA的生物信息学预测等分析。
3.2 预测到的甘草miRNA、前体及其二级结构以已知植物miRNA前体序列为探针与拼接获得的88 263条甘草转录本序列进行BLASTN比对分析,筛选出685条候选miRNA前体序列。去除其中的蛋白编码基因后,进一步对这些转录本进行手动筛选,确定了其中能够用Mfold软件折叠形成稳定的茎环结构,并符合相应筛选标准的核酸序列共50条,代表 30个茎环前体序列(表 2和图 1)。在这些前体序列上分布着分属于17个不同家族的49个miRNA的成熟序列(表 3)。其中,家族成员最多的是miR156家族,有8个成员;其次为miR159家族,有7个成员。
|
|
表 2 甘草中新发现的miRNA前体 Table 2 Precursors of miRNA newly discovered from G. uralensis |
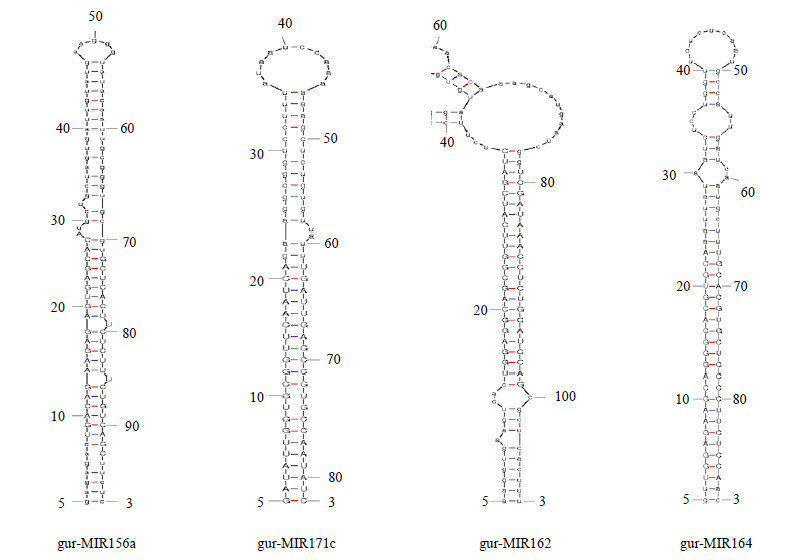
|
图 1 部分miRNA前体的二级结构 Fig.1 econdary structures of some precursors of miRNA |
|
|
表 3 甘草中新发现的miRNA Table 3 miRNAs newly discovered from G. uralensis |
在甘草中预测到的49条成熟miRNA的长度范围为20~24 nt,其中21 nt长度的miRNA最多(31/49,63%),其次为22 nt的miRNA(11/49),其他长度的miRNA数量较少。预测的30个stem-loop前体的序列长度介于82~187 nt,这些前体都能折叠成稳定的二级结构,最小自由能MFE介于-101.9~-30.4 kcal/mol,最小自由能指数MFEI介于0.67~1.03(表 2)。
本实验考察了49个预测的甘草miRNA与其他植物来源的同源miRNA的序列相似性(表 3)。71%(35/49)的甘草miRNA与其他植物中对应miRNA的序列完全相同,10%(5/49)的甘草miRNA出现1个差异碱基,出现多于1个差异碱基的甘草miRNA共有8个,占16%。这说明本研究预测的miRNA多数为保守miRNA。还考察了31个预测的甘草miRNA前体与其他植物来源的miRNA前体的序列相似性(表 3)。发现所有前体与其他植物来源的miRNA前体都有程度不同的相似性,E值最大为3e-12,最小为7e-42。这一结果说明,与蛋白编码基因类似,同源miRNA的成熟形式和茎环结构前体在序列上也具有保守性。
3.3 甘草根中miRNA的验证为了确定生物信息学预测中的miRNA真实存在,本研究随机选取了12条新发现的miRNA,通过stem-loop qRT-PCR实验进行验证。实验结果显示,12条miRNA荧光定量扩增产物的熔解曲线均为单峰,将12条miRNA荧光定量PCR产物进行琼脂糖凝胶电泳,均出现明显单一条带,大小约60 bp,与荧光定量PCR扩增产物的实际大小一致(图 2),证明本研究通过生物信息学预测的miRNA在甘草中确实存在。
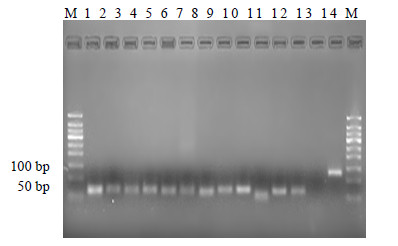
|
图 2 miRNA stem-loop qRT-PCR产物电泳图 Fig.2 Electrophoresis of miRNA stem-loop qRT-PCR products |
3.4 甘草miRNA靶基因的预测及功能分析
根据预测的49条甘草miRNA序列以及拼接获得的转录本序列,通过在线软件psRNATarget对其靶基因进行了预测,共预测到32条甘草miRNA的273个靶基因,其中有172个靶基因可以用拟南芥蛋白序列进行注释。靶基因编码的蛋白具体包括以下几类:(1)转录因子,包括SBP类、myb类、GRAS类、bHLH类、NAC类、bZIP类、TCP类等转录因子;(2)激酶和磷酸酶,包括MAPKKK5、富含亮氨酸重复的蛋白激酶Leucine-rich repeat protein kinase、U-box domain-containing protein kinase、protein phosphatase 2CA等;(3)激素信号转导成员,包括ARF(生长素响应因子)、生长调节因子5(GRF5 | growth-regulating factor 5)、AFB2 | auxin signaling F-box 2等;(4)抗病蛋白,如NB-ARC domain-containing disease resistance protein、LRR and NB-ARC domains-containing disease resistance protein、Disease resistance protein(TIR-NBS-LRR class)family;(5)其他类别的酶等。GO分类和富集分析结果(图 3)表明被富集的生物学过程为DNA依赖的转录调控(GO:0006355)、器官发育(GO:0048513)和防御反应(GO:0006952)。被富集的分子功能包括转录因子活性(GO:0003700)和泛素蛋白连接酶活性(GO:0004842)。以上分析结果表明,甘草miRNA主要通过调控转录因子等靶基因参与基因转录调控、信号转导、胁迫应答、蛋白翻译后修饰等生物学过程。
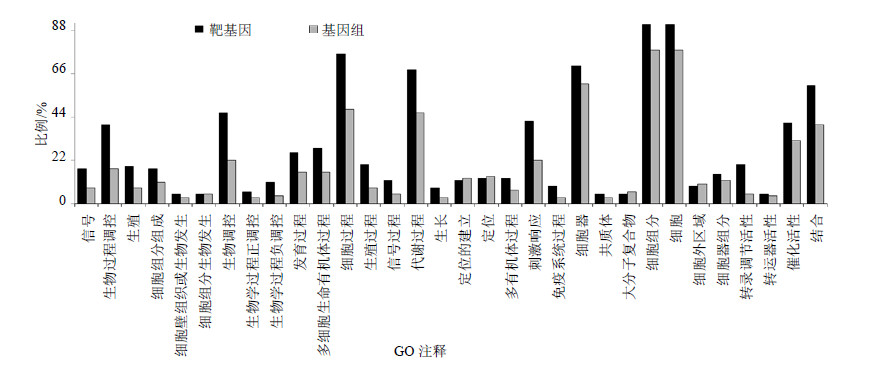
|
图 3 甘草miRNA靶基因编码蛋白的GO分类情况 Fig.3 GO classification of predicted miRNA target genes in G. uralensis |
4 讨论
与动物miRNA相比,植物miRNA的研究相对较晚。尽管如此,随着第二代测序技术的飞速发展,研究人员已从数十种植物中鉴定出少到几十个、多至几百个的miRNA,并对部分植物的miRNA进行了进一步的功能分析。这些研究表明miRNA广泛参与调控植物的生长发育、形态建成、次生代谢物生成、信号转导以及环境因子应答等[20]。甘草作为我国传统大宗中草药,同时也是维持我国西北部干旱区域生态系统健康的重要植物[1-2],其次生代谢产物的调控、积累机制和干旱等逆境适应机制都与miRNA存在密切的内在联系,鉴定甘草miRNA、研究miRNA的生物学功能将促进对其相关分子机制的了解。
目前对于中草药等非模式植物的miRNA的鉴定主要依赖于转录组序列。转录组序列有两类来源:表达序列标签(EST)和高通量转录组测序数据。目前随着高通量测序费用的不断降低,对中草药植物进行的高通量转录组测序研究不断增加[21],为miRNA的鉴定提供了数据基础。miRNA鉴定的关键是找到miRNA的茎环结构前体,因此获得高覆盖度的转录本数据是鉴定miRNA前体的前提。在GenBank中的甘草核酸数据中,既有5万余条来自Sanger测序的EST序列,也有若干个高通量测序数据。为了获得高质量的转录本序列数据,应用Trinity等常用拼接软件,将不同来源的转录本数据拼接在一起,拼接得到的转录本数据覆盖度较好,这一高质量的转录本数据将有助于后续的miRNA的生物信息学鉴定和靶基因预测研究。
在miRNA的诸多鉴定方法中,生物信息学预测无须进行测序,简便快捷,因而得到较多的应用[15, 17, 22-23]。在这些研究中,主要采用2种方法进行miRNA的生物信息学鉴定。第1种鉴定方法是,将相关物种的miRNA成熟形式比对到转录本上,允许3~4个碱基错配,然后取一定长度的侧翼序列(如200 nt),用mfold等RNA折叠软件进行分析[17]。第2种方法是本研究采用的方法[15]。这种做法与第一种方法的主要区别在于,比对到转录本序列上的是相关物种的已知miRNA的茎环前体序列,而不是成熟形式的序列。这2种方法各有特点,也都有一定的应用。本研究在预试验中比较了这2种鉴定方法,发现第2种方法更方便易行,人工验证部分工作量相对较少,因而采用了这个方法。
总之,本研究应用生物信息学手段,在转录本序列拼接的基础上,预测了甘草miRNA,确定了一批甘草miRNA的靶基因,为认识甘草miRNA的生物学功能提供了初步数据。
| [1] | 刘洋洋, 刘春生, 曾斌芳, 等. 甘草种质资源研究进展[J]. 中草药 , 2013, 44 (24) :3593–3598. |
| [2] | 李学斌, 陈林. 中国甘草资源的生态分布及其繁殖技术研究[J]. 生态环境学报 , 2013, 22 (4) :718–722. |
| [3] | Lee R C, Feinbaum R L, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14 [J]. Cell, 1993, 75(5): 843-854. |
| [4] | Reinhart B J, Slack F J, Basson M, et al. The 21 nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans[J]. Nature , 2000, 403 (6772) :901–906. DOI:10.1038/35002607 |
| [5] | Qin Z, Li C, Mao L, et al. Novel insights from non-conserved microRNAs in plants[J]. Front Plant Sci , 2014, 5 :586–589. |
| [6] | Budak H, Akpinar B A. Plant miRNAs: biogenesis, organization and origins[J]. Funct Integr Genomics , 2015 . |
| [7] | Kozomara A, Griffiths-Jones S. miRBase: annotating high confidence microRNAs using deep sequencing data[J]. Nucl Acids Res , 2014, 42 . |
| [8] | 曾奇峰, 曾光尧, 周应军. MicroRNA与药用植物[J]. 中国生物化学与分子生物学报 , 2015, 31 (4) :346–351. |
| [9] | 周芳名, 白志川, 卢善发. 药用植物microRNA[J]. 中草药 , 2013, 44 (2) :232–237. |
| [10] | Bolger A M, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics , 2014, 30 (15) :2114–2120. DOI:10.1093/bioinformatics/btu170 |
| [11] | Cox M P, Peterson D A, Biggs P J. SolexaQA: At-a-glance quality assessment of Illumina second-generation sequencing data[J]. BMC Bioinformatics , 2010, 11 :485–489. DOI:10.1186/1471-2105-11-485 |
| [12] | Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-seq data without a reference genome[J]. Nat Biotechnol , 2011, 29 (7) :644–652. DOI:10.1038/nbt.1883 |
| [13] | Zheng Y, Zhao L, Gao J, et al. iAssembler: a package for de novo assembly of Roche-454/Sanger transcriptome sequences[J]. BMC Bioinformatics , 2011, 12 :453–456. DOI:10.1186/1471-2105-12-453 |
| [14] | Fu L M, Niu B F, Zhu Z W, et al. CD-HIT: accelerated for clustering the next generation sequencing data[J]. Bioinformatics , 2012, 28 (23) :3150–3152. DOI:10.1093/bioinformatics/bts565 |
| [15] | Khan B M Y, Irfan M, Yousaf R, et al. Identification of micro-RNAs in cotton[J]. Plant Physiol Biochem , 2008, 46 (8/9) :739–751. |
| [16] | Zuker M. Mfold web server for nucleic acid folding and hybridization prediction[J]. Nucl Acids Res , 2003, 31 (13) :3406–3415. DOI:10.1093/nar/gkg595 |
| [17] | Zhang B H, Pan X P, WU Q L, et al. Identification and characterization of new plant microRNAs using EST analysis[J]. Cell Res , 2005, 15 (5) :336–360. DOI:10.1038/sj.cr.7290302 |
| [18] | Ramilowski J A, Sawai S, Seki H, et al. Glycyrrhiza uralensis transcriptome landscape and study of phytochemicals[J]. Plant Cell Physiol , 2013, 54 (5) :697–710. DOI:10.1093/pcp/pct057 |
| [19] | Li Y, Luo H M, Sun C, et al. EST analysis reveals putative genes involved in glycyrrhizin biosynthesis[J]. BMC Genomics , 2010, 11 :268–275. DOI:10.1186/1471-2164-11-268 |
| [20] | Voinnet O. Post-transcriptional RNA silencing in plant-microbe interactions: A touch of robustness and versatility[J]. Curr Opin Plant Biol , 2008, 11 (4) :464–470. DOI:10.1016/j.pbi.2008.04.006 |
| [21] | 赵春丽, 李先恩, 都晓伟, 等. 地黄microRNAs和靶基因的生物信息学预测及验证[J]. 中草药 , 2014, 45 (8) :1129–1135. |
| [22] | 李崇奇, 沈文涛, 言普, 等. 巨桉miRNA及其靶基因生物信息学预测[J]. 南方农业学报 , 2014, 45 (9) :1532–1538. |
| [23] | 夏薇, 李静, 周丽霞. 椰子保守microRNA预测和特征分析[J]. 广东农业科学 , 2014, 41 (14) :130–135. |
 2016, Vol. 47
2016, Vol. 47


