鱼腥草Houttuynia cordata Thunb.为三白草科(Saururaceae)蕺菜属Houttuynia Thunb.植物[1],因其全株有特殊的鱼腥气味而得名,鱼腥草别名侧耳根、猪鼻孔、狗贴耳等。鱼腥草作为一种具有清热解毒,利尿消肿等功效的中药材,使用历史悠久[2]。其注射液常用来治疗呼吸道、泌尿系统炎症疾病,外科手术中也常用鱼腥草提取液进行消炎、抑菌。对消化系统疾病、皮肤病等也有很好的疗效[3]。
DNA分子标记能在分子水平上检测出DNA遗传多态性,具有较大的应用价值[4]。迄今在鱼腥草植物中所用到的分子标记仍局限于随机扩增多态DNA(RAPD)[5-6]和扩增简单重复序列间区(ISSR)[7]等随机引物标记。由于其稳定性和重现性一直备受质疑,因此目前已较少使用。最常用的特异标记简单序列重复(simple sequence sepeat,SSR)因其重现性好,扩增靶序列在基因组中位置确定等优点,备受研究者青睐。然而,SSR标记的开发难度大,不仅费时费力,且费用很高,多局限在具备全基因组数据或大量表达序列标签的物种中进行开发[8]。目前尚未见鱼腥草植物中开发SSR标记的报道。
前期课题组成员对鱼腥草进行了转录组测序,获得了大量的unigenes序列,本研究将尝试利用这些序列,首次开发鱼腥草的SSR标记,期望这些标记可以用于鱼腥草的亲缘关系分析、种源鉴定及遗传图谱构建等方面的研究。
1 材料与方法 1.1 鱼腥草样品采集、转录组测序与SSR的筛选采集不同产地的样品16份(表 1),经怀化学院伍贤进教授鉴定为鱼腥草Houttuynia cordata Thunb.。鱼腥草地下茎、地上茎、花和叶片RNA混合样品的转录组测序由华大基因公司进行,共获得185 276条Contigs,组装出63 954条unigenes。以组装出来的Unigene作为参考序列,使用SSR软件MicroSAtellite(http://pgrc.ipk-gatersleben.-de/misa/),来找出所有的SSR。参数设置为“1-12、2-6、3-5、4-5、5-4、6-4”,1-12代表单碱基重复至少12次才算SSR,双碱基6次,三碱基5次,以此类推,重复单元最多有6个碱基。
|
|
表 1 不同来源的鱼腥草材料 Table 1 H. cordata from different sources |
1.2 SSR引物设计
对所有SSR重复单元在unigene上前后序列的长度进行筛选。只保留前后序列均不小于150 bp的SSR,用Primer3软件对其设计引物。为验证SSR引物是否可用和检测SSR的可靠性,随机选择合成50对引物,由生工生物工程(上海)股份有限公司合成,然后进行PCR扩增。
1.3 鱼腥草基因组DNA提取与PCR扩增采用改进的CTAB法提取鱼腥草基因组DNA。利用核酸/蛋白定量仪定量。定量后的DNA稀释至30 μg/mL以备PCR扩增用。反应体系:10×buffer 2 μL,1 mol/L MgCl2 1 μL,10 μmol/L上游引物和下游引物各0.7 μL,2.5 mmol/L dNTPs 1 μL,Taq DNA polymerase 0.5 μL,10 mg/L模板DNA 2 μL,加ddH2O至总16 μL。反应条件为94 ℃预变性1 min,94 ℃变性30 s,最佳Tm退火30 s,72 ℃延伸30 s,循环35次,72 ℃延伸5 min。取扩增产物2 μL先用1.5%琼脂糖判断是否具有多态性条带,确定具有多态性条带的,将剩余产物用8%非变性聚丙烯酰胺凝胶检测,进行基因分型与条带读取。
1.4 数据处理采用人工读带的方法,将电泳图上可重复的、清晰的条带计为“1”,同一位置无带或不易分辨的弱带计为“0”,建立原始数据矩阵。利用软件Popgene 32计算每对引物扩增位点的等位位点数、多态性信息量、遗传距离,用Mega4软件进行UPGMA聚类分析。
2 结果与分析 2.1 鱼腥草转录组SSR位点数量与分布对鱼腥草转录组测序获得总长度为43 395 361 bp的63 954条unigenes数目进行检测,在4 413条unigenes数目中找到符合条件的SSR,发生频率(含有SSR的unigenes数目与总unigenes数目之比)为6.9%,其中357条unigenes含1个以上SSR位点,4 056条unigenes数目含单个SSR位点,共检出4 800个SSR位点,复合SSR位点164个。转录组中SSR位点的出现频率(SSR数目与总unigene的数目比值)为7.51%。从分布情况看,鱼腥草转录组中平均9.04 kb有1个SSR。但不同重复基序长度SSR分布的平均距离差别巨大,其中三核苷酸重复最多(1 994个),分布平均距离21.76 kb/SSR,Quad-nucleotide repeat最少(39个),每条SSR分布平均距离1 112.7 kb(表 2)。鱼腥草转录组SSR位点的序列总长达80 754 bp,其中单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸重复基元的SSR位点的碱基总长分别是17 587、25 362、33 012、800、1 365、2 628 bp。各类型SSR位点的平均长度分别是13、19、17、21、21、24 bp(表 2)。
|
|
表 2 SSR在鱼腥草转录组中的出现频率 Table 2 Occurrence frequency of SSRs in H. cordata transcriptome |
2.2 鱼腥草转录组SSR的特性
鱼腥草转录组SSR种类丰富,一至六核苷酸重复类型都能看到,但各类型出现频率不同,主要是单核苷酸、二核苷酸和三核苷酸重复,占总SSR的95.52%,其中三核苷酸重复最多,占41.54%,其次是二核苷酸重复和单核苷酸重复类型,分别占26.63%和27.35%,四核苷酸、五核苷酸和六核苷酸重复类型的数量较少,分别占0.81%、1.38%、2.29%。从重复次数来看,发现重复基元以重复5次出现的频率最高,有1 426个,占总SSRs的29.71%,其次是重复6次、≥13次、12次、7次和8次,分别有980、710、608、492、223个SSR,分别占总SSR的20.42%、14.77%、12.59%、10.25%和4.65%,最少的为重复基元重复11次,只有80个,占1.7%(表 3)。对鱼腥草转录组SSR统计分析,发现共有232种重复基元,其中单核苷酸的A/T、G/C重复基元2种。
|
|
表 3 鱼腥草SSR不同基序长度和重复的数量分布 Table 3 Distribution of SSR with different motif types and repeat numbers in H. cordata |
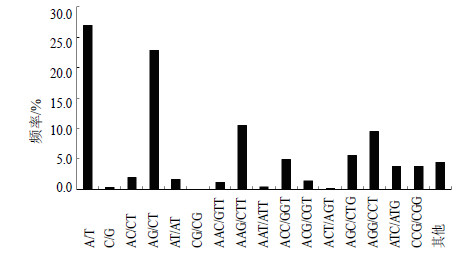
二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸重复基元分别有12、58、26、52、84种。从出现频率来看,占优势的重复基元类型是单核苷酸重复基元,其次是二核苷酸和三核苷酸重复基元,其他四核苷酸、五核苷酸和六核苷酸重复基元类型分布较少,出现频率较低(图 1)。
|
图 1 鱼腥草不同基序SSR类型的频率分布 Fig.1 Frequency distribution of SSR based on different motif sequence types in H. cordata |
对二核苷酸和三核苷酸重复基元深入分析,按照4种碱基随机随机组合的方式统计后,发现二核苷酸和三核苷酸重复基元分别为4种和10种,即这2种核苷酸重复基元类型在鱼腥草SSR序列中全部存在。其中二核苷酸重复基元出现最多的是AG/CT,共有1 108个,占该重复基元的86.70%,其他的2种AT/GC、AC/GT则分别仅占6.34%和6.96%;三核苷酸重复种类较多,最多的重复基元为GAA/CTT,共有336个,GAA/CCT也有333个,分别占三核苷酸重复基元的16.85%、16.70%。其次的为GGT/CCA(共有168个)、AGA/TCT(共有153个)、GAG/CTC(共有153个)、GCT/CGA(共有89个)、CAG/GTC(共有62个)。
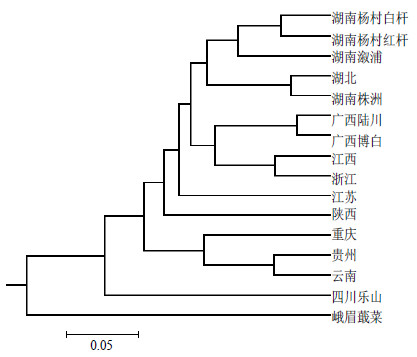
2.3 SSR引物筛选与验证选择前后序列均不小于150 bp的SSR 50个,根据引物设计原则,应用软件Primer3设计引物,对所获得的4 800个SSR位点设计引物,共开发出3 068个候选SSR引物。随机挑选了50对引物,其中有43对引物能扩增出清晰条带,并表现出多态性差异,多态信息量(PIC)为0.49~0.96,平均值为0.72;等位基因数范围为3~10,平均为5.74,表观杂合度(Ho),期望杂合度(He)见表 4。利用SSR多态性对16个鱼腥草材料进行聚类分析,聚类结果显示,峨嵋蕺菜单独聚为一类;其他群体聚为一类。四川群体与其他各个省群体分别列为2个亚类;云南、贵州、重庆等的遗传距离较近聚为一群;湖南、湖北的遗传距离较近聚为一群;广西、江西、浙江等的遗传距离较近聚为一群(图 2)。
|
|
表 4 SSR-PCR 筛选引物 Table 4 Screening primers of SSR-PCR |
|
图 2 鱼腥草的UPGMA聚类图 Fig.2 UPGMA dendrogram for H. cordata |
3 讨论
基于PCR技术的分子标记,较常用的有随机扩增多态DNA(RAPD)[9]、扩增片段长度多态性(AFLP)[10]和扩增简单重复序列间区(ISSR)[11]等。这些标记因其特异性不强而广泛应用于物种的遗传多样性与亲缘关系等方面的研究,尤其对不具任何基因组信息的非模式生物的研究,凸显其优势。对不具任何基因组信息的鱼腥草,目前已利用RAPD[5-6]、AFLP[12]、ISSR[7]等分子标记进行了研究,由于这些标记的扩增产物是不确定、非特异的,因而用它们得出的结果不能有效地应用于遗传研究和标记辅助育种。SSR分子标记是特异性较强的标记,能有效地克服不确定、非特异的缺点,而且对已开发出大量特异标记的植物,已很少使用非特异标记对其进行遗传多样性与亲缘关系等方面的研究。为了更加有效的开发和利用药用植物鱼腥草的遗传资源,完全有必要开发其特异性较强的分子标记,而且迄今尚未见鱼腥草植物中开发SSR标记的报道。
从鱼腥草转录组中搜索出4 800个SSRs,平均出现频率是1/9.04 kb,和其他物种相比,鱼腥草SSRs出现频率高于小麦(1/15.6 kb)[13]、拟南芥(1/13.83kb)、番茄(1/11.1 kb)、棉花(1/20.0kb)[14]、杨树(1/14.0kb)[15]等植物,表明鱼腥草转录组中SSR数量相当丰富。从鱼腥草SSR重复基元分析,鱼腥草SSR与其他植物相比存在相似性和差异性。其相似性表现在鱼腥草SSR序列的重复基元种类繁多,单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸、六核苷酸重复基元等。而重复基元主要集中在单核苷酸、二核苷酸和三核苷酸重复。其二核苷酸重复基元以AG/CT为主(占总数的86.70%),这与烟草[16]、茶树[15]、马尾松[17]、杏树[18]等的SSR序列二核苷酸重复基元的情况相似。鱼腥草三核苷酸重复以GAA/CTT重复为主(占16.85%),这与拟南芥[19]、柑橘[20]、烟草[21]等报道的情况基本一致,这可能是这些重复基元与其关键蛋白编码的密码子使用频率有较大的关系[22]。鱼腥草SSR重复基元与其他物种的情况也存在一定的差异性,鱼腥草SSR序列以三核苷酸重复基元重复为主,与茶树[15]、烟草[16]等有所差异。
鱼腥草转录组SSR不但出现频率高,而且类型丰富;利用unigenes序列开发鱼腥草的SSR标记进行试验验证,所开发的SSR标记在鱼腥草基因组中的检测结果多表现为清晰可读的单一主带或2条带,表明被检测的标记在基因组DNA中的位置是特异的,能有效反映其多态性,从多态性潜能的角度考虑,搜索到的这些SSR也具有较高的可用性。可成为鱼腥草基因组DNA遗传多样及其分子生物学其他相关研究的有力工具。本研究结果为进一步发掘新的鱼腥草功能基因SSR标记奠定了基础,这种标记的建立对于加速鱼腥草功能基因资源的开发利用、遗传资源评价及实现特定性状的辅助选择标记育种和开展鱼腥草的比较基因组学研究均具有重要意义与应用价值。
| [1] | 马林, 吴丰, 陈若芸. 三白草科植物化学及生物活性研究进展[J]. 中国中药杂志, 2003,28 (3) :196–198. |
| [2] | 中国药典[S]. 一部. 2015. |
| [3] | 伍贤进, 张俭, 钟晓丽, 等. 鱼腥草储藏过程中化学成分含量的变化[J]. 食品工业科技, 2007,28 (5) :215–217. |
| [4] | 方宣钧, 吴为人, 唐纪良. 作物DNA标记辅助育种[M]. 北京 : 科学出版社 , 2001 . |
| [5] | 吴卫, 郑有良, 陈黎, 等. 鱼腥草种质资源的RAPD分析[J]. 药学学报, 2002,37 (12) :986–992. |
| [6] | 蓝云龙, 吴令上, 裘波音, 等. 鱼腥草RAPD分子标记的多态性[J]. 浙江林学院学报, 2008,25 (3) :309–313. |
| [7] | Wei L, Wu X J. Genetic variation and population differentiation in a Medical Herb Houttuynia cordata in China revealed by ISSRs[J]. Int J Mol Sci, 2012, 13 (16) :8159–8170 . |
| [8] | 易官美, 黎建辉, 王冬梅, 等. 南方红豆杉SSR分布特征分析及分子标记的开发[J]. 园艺学报, 2013,40 (3) :571–578. |
| [9] | Williams J G K, Kubelik A R, Livak K J, et al. DNA polymorphism amplified by arbitrary primers are useful as genetic markers[J]. Nucl Acids Res, 1990, 18 (13) :6531–6535 . |
| [10] | Vos P, Hogers R, Bleeker M, et al. AFLP:A new technique for DNA fingerprinting[J]. Nucl Acid Res, 1995, 23 :4407–4414 . |
| [11] | 黎晓英, 魏麟, 伍贤进, 等. 中国不同地理居群鱼腥草遗传多样性分析[J]. 中草药, 2010,41 (2) :285–288. |
| [12] | Zietkiewicz E, Rafalski A, Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification[J]. Genomics, 2003, 19 (20) :176–183 . |
| [13] | Kantety R V, Rota M L, Matthews D E, et al. Data mining for simple sequence repeats in expressed sequence tags from barley, maize, rice, sorghum and wheat[J]. Plant Mol Biol, 2002, 48 (9) :501–506 . |
| [14] | Gao L F, Tang J F, Li H W, et al. Analysis of microsatellites in major crops assessed by computational and experimental approaches[J]. Mol Breed, 2003, 12 (13) :245–256 . |
| [15] | 金基强, 崔海瑞, 陈文岳, 等. 茶树EST-SSR的信息分析与标记建立[J]. 茶叶科学, 2006,26 (1) :17–23. |
| [16] | 赵世浩. 烟草EST-SSR标记的建立及其应用[D]. 长沙:湖南农业大学, 2007. |
| [17] | 刘公秉, 季孔庶. 基于松树EST序列的马尾松SSR引物开发[J]. 分子植物育种, 2009,7 (4) :833–838. |
| [18] | Jung S, Abbott A, Jesudurai C, et al. Frequency type distribution and annotation of simple sequence repeats in Rosaceae ESTs[J]. Funct Integr Genomics, 2005, 19 (5) :136–143 . |
| [19] | Morgante M, Hanafey Wayne Powell. Microsatellites are prefertially associated with nonrepetitive DNA in plant genomes[J]. Nat Genet, 2000, 30 (16) :194–200 . |
| [20] | Chen C X, Zhou P, Choi YA, et al. Mining and characterizing microsatellites from citrus ESTs[J]. Theor Appl Genet, 2006, 112 (19) :1248–1257 . |
| [21] | 赵世浩. 烟草EST-SSR标记的建立及其应用[D]. 长沙:湖南农业大学, 2007. |
| [22] | 范三红, 郭蔼光, 单丽伟, 等. 拟南芥基因密码子偏爱性分析[J]. 生物化学与生物物理进展, 2003,23 (30) :221–225. |
 2016, Vol. 47
2016, Vol. 47


