2. 中国人民解放军第二军医大学药学院 药用植物学教研室, 上海 200433
2. Department of Pharmaceutical Botany, School of Pharmacy, Second Military Medical University, Shanghai 200433, China
丹参Salviae Miltiorrhizae Radix etRhizoma是唇形科(Labiatae)鼠尾草属Salvia L. 植物丹参Salvia miltiorrhiza Bge. 的干燥根及根茎,味苦,性微寒,归心、肝经,具有祛瘀止痛、活血通经、清心除烦的功效[1]。丹参酮类化合物,是公认的丹参有效成分之一。现代药理学研究表明,丹参酮具有清除氧自由基,改善缺氧后引起的心肌代谢紊乱及心功能障碍;修复血管内皮细胞,抗动脉粥样硬化形成;降低心肌耗氧量,缩小心肌梗死面积等作用;还具有抗菌、抗炎、钙拮抗、抗心肌肥厚以及性激素样作用[2]。
丹参酮属于脂溶性二萜醌类化合物,主要来源于MEP途径(2-C-甲基-D-赤藓糖醇-4-磷酸途径)[3],研究发现,由柯巴基焦磷酸(copalyl diphosphate,CPP)形成的松香二烯可能是丹参中松香烷型二萜醌类化合物生物合成途径中的前体物质[4, 5]。而柯巴基焦磷酸合酶(copalyl diphosphate synthase,CPS)的作用是催化线性结构的牻牛儿基牻牛儿基焦磷酸(geranylgeranyl pyrophosphate,GGPP)环化形成4种可能的CPP异构体。高伟等[6]通过基因芯片等技术发现,丹参酮IIA量高的样品中CPS基因表达也升高,表明该基因可能参与了丹参酮类化合物的生物合成。Zhou等[7]在酿酒酵母中融合表达GGPP合酶、法尼基焦磷酸(FPP)合酶、CPS和类贝壳杉烯合酶(KSL)后,产生了丹参酮的前体物质次丹参酮二烯,再次证实了CPS在丹参酮类化合物生物合成过程中的作用。
CPS是植物三环二萜类化合物生物合成过程中的重要环化酶,正在受到越来越多的关注,但是关于其功能的系统研究还未见报道,利用生物信息学方法,对CPS进行全面预测,能为后续研究指明方向。通过生物信息学方法对丹参中已克隆的3个CPS基因编码蛋白进行了一级结构、二级结构和三级结构的预测分析,并与其他15种植物进行比较,同时进行了这16种植物CPS蛋白序列同源比对及系统进化树构建,旨在探讨丹参CPS蛋白功能,为丹参CPS蛋白的后续研究提供有利参考。
1 数据下载以“Copalyl diphosphate synthase”为搜索关键词,从美国国立生物技术信息中心(National Center of Biotechnology Information,NCBI)下载完整的丹参CPS蛋白序列,并下载其他15种植物的CPS蛋白序列以供比较。药材信息见表 1。
| 表 1 丹参及其他植物CPS基因的基本信息 Table 1 Basic information of CPS in Salviae Miltiorrhizae Radix et Rhizoma and other plants |
利用各网站提供的生物信息学工具对丹参及其他植物CPS蛋白序列进行在线分析。用ProtParam(http://web.expasy.org/protparam/)预测CPS蛋白的组成成分和理化性质;用ProtScale(http://web.expasy.org/protscale/)对CPS蛋白的疏水/亲水性进行预测;CPS蛋白的导肽和信号肽的预测分别用TargetP 1.1 Server(http://www.cbs.dtu.dk/services/TargetP/)[8]和SignalP 4.1 Server(http://www.cbs. dtu.dk/services/SignalP/)[9]完成;跨膜结构用TMHMM Server v. 2.0(http://www.cbs.dtu.dk/services/ TMHMM-2.0/)进行预测;CPS蛋白二级结构的预测用SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)完成,功能域的预测用Pfam 27.0(http://pfam.janelia.org/)和SMART(http://smart.embl-heidelberg.de/)[10]进行;用Phyre2(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi? id=index)[11]和PDBsum Generate(http://www. ebi.ac.uk/thornton-srv/databases/pdbsum/Generate.html)完成CPS蛋白高级结构同源建模和建模结果的检验。
CPS蛋白序列同源比对采用blastall 2.2.26本地版完成;CPS蛋白家族系统进化树的构建采用邻位连接法(neighbor-joining,NJ),用Bootstrap method检验进化树的拓扑结构,重复抽样次数(No. of bootstrap replication)设为1 000,Gaps/Missing Data Treatment选择Pairwise deletion,由MEGA 5.0软件完成。
3 结果与分析 3.1 CPS蛋白序列的组成成分及理化特性分析利用ProtParam在线工具对丹参及其他植物CPS蛋白序列进行理化性质分析。16种植物CPS蛋白的氨基酸残基(amino acids,aa)数除蒺藜苜蓿为158 aa外,其他都在730 aa以上;各蛋白序列的相对分子质量为17 780~99 540,中位值为91 610;理论等电点均在6左右,平均5.87,提示CPS蛋白为酸性蛋白。从CPS氨基酸组成中可以看到,16种植物的CPS蛋白所含酸性氨基酸残基比例均高于所含碱性氨基酸残基比例,进一步提示CPS蛋白为酸性蛋白。各种植物CPS蛋白中,含量最丰富的氨基酸残基主要集中在亮氨酸(Leu)、谷氨酸(Glu)、丝氨酸(Ser)、丙氨酸(Ala)、天冬氨酸(Asp)和赖氨酸(Lys)。在这几种植物氨基酸残基中,Leu是各种植物中量最高的氨基酸(蒺藜苜蓿除外)。蒺藜苜蓿中,量最高的氨基酸是Glu,占10.1%,其次是Leu和天冬酰胺(Asn),均为9.5%。通过ProtParam分析,除丹参SmiCPS2、笋瓜、向日葵和毛萼香茶菜的CPS蛋白不稳定系数小于40,为稳定蛋白,其他几种植物的CPS蛋白均为不稳定蛋白。
3.2 CPS蛋白导肽的预测和分析导肽(leader peptide)是一段引导新合成的肽链进入细胞器的识别序列[12],导肽的预测与分析对蛋白质的功能分析、作用机制和作用途径等具有重要意义[13]。通过在线预测工具TargetP 1.1 Server,将置信区间设为0.95,对丹参及其他多种植物的CPS蛋白序列进行了预测(表 2)。结果显示,SmiCPS1、SmiCPS2和SmiCPS3 3条丹参CPS蛋白的导肽预测可靠性分别为V、IV和II级,SmiCPS1和SmiCPS3可能具有叶绿体转运肽(chloroplast transit peptide),且在各自蛋白序列第35位和第21位分别存在一个导肽分裂位点。无法确定SmiCPS2是否具有导肽,也未发现其导肽分裂位点。其他15种植物CPS蛋白的导肽预测分析结果显示,迷迭香的可靠性为V级,其余植物都在III级以上。与SmiCPS1和SmiCPS3相似,巨冷杉、小粒咖啡、笋瓜、野甘草、番茄、可可和台湾杉都具有导肽分裂位点,具有导肽性,且它们的导肽很可能都是叶绿体转运肽,提示这些植物的CPS蛋白合成后,可能转运到叶绿体中发挥作用。剩下植物与SmiCPS2相似,都不存在导肽分裂位点,不能确定具有何种导肽。
| 表 2 丹参及其他植物CPS蛋白的导肽预测 Table 2 Leader peptide prediction of CPS protein in Salviae Miltiorrhizae Radix et Rhizoma and other plants |
信号肽位于新合成肽链N端,是引导新合成肽链转移到内质网上的一段多肽[14]。从CPS蛋白导肽的预测结果中发现,所有蛋白都不存在信号肽,为了验证这个结果,利用更专业的信号肽在线分析工具SignalP 4.1 Server,对丹参CPS蛋白信号肽的存在位置进行了分析,结果表明丹参CPS蛋白中不存在信号肽。对其他15种植物CPS蛋白序列进行相同预测,也得到类似结果。
3.4 CPS蛋白疏水性/亲水性的预测和分析蛋白质亲疏水性氨基酸组成是蛋白质折叠的主要驱动力,ProtScale程序能用来绘制蛋白质亲疏水性序列谱,反映蛋白质的折叠情况[15]。利用ProtScale对丹参CPS蛋白序列进行疏水性/亲水性预测,结果显示,SmiCPS1多肽链第600位氨基酸具有最低分值-2.733,亲水性最强;第160位氨基酸具有最高分值2.333,疏水性最强;SmiCPS2的最低分值-2.878位于第8位,最高分值2.9位于第17位;SmiCPS3第685位亲水性最强,为-3.244,第163位疏水性最强,为2.367。从总体上看,3条序列的疏水区域明显小于亲水区域,均表现为亲水性。用相同方法对其他植物CPS蛋白序列进行亲疏水性分析,结果与丹参相似,提示CPS可能是亲水性蛋白。
3.5 CPS蛋白跨膜结构域的预测和分析跨膜结构域一般由20个左右的疏水性氨基酸残基组成,主要形成α-螺旋,常由跨膜蛋白的效应区域所展现[12]。利用在线工具TMHMM Server v. 2.0对丹参CPS蛋白进行跨膜结构分析,结果显示,丹参CPS蛋白整条肽链都位于细胞膜之外,不存在跨膜结构。其他植物的CPS蛋白跨膜结构域分析结果与丹参一致,提示本实验中的CPS蛋白均不具跨膜结构域。
3.6 CPS蛋白二级结构的预测和分析蛋白质二级结构是指蛋白质多肽链氨基酸残基借助氢键折叠和盘绕形成的α-螺旋、β-折叠、无规则卷曲以及模体等组件,其中,α-螺旋和β-折叠是最常见的蛋白质二级结构[13]。利用SOPMA对丹参CPS蛋白序列进行二级结构预测(表 3),结果显示,α-螺旋和无规则卷曲是丹参CPS蛋白二级结构的主要结构元件,其次是延伸链和β-折叠。其他植物CPS蛋白序列的二级结构预测结果显示,除江南卷柏和台湾杉外,均与丹参一致,都是α-螺旋所占比例最多,其次是无规则卷曲和延伸链,所占比例最少的是β-折叠。在江南卷柏中,延伸链和β-折叠所占比例一致,都是3.79%;而在台湾杉中,β-折叠(5.45%)的比例略高于延伸链(5.33%)。
| 表 3 丹参等植物CPS蛋白二级结构主要构成组件比例 Table 3 Main component proportion of secondary structure of CPS protein in Salviae Miltiorrhizae Radix et Rhizoma and other plants |
功能域(functional domain)又称结构域,是蛋白质分子中介于二级与三级结构之间的一种独立结构和功能单位,具有特定的生物学功能[16, 17]。利用Pfam 27.0程序对丹参CPS蛋白序列进行功能结构域预测,结果显示,丹参CPS蛋白具有一个terpene synthase N terminal domain和一个terpene synthase C terminal domain,行使植物次级代谢过程中萜类物质合成作用。为确保预测结果的准确性,通过在线工具SMART进行验证,结果与Pfam 27.0一致。对其他植物进行同样的功能域预测和分析后发现,除蒺藜苜蓿只具有一个terpene synthase C terminal domain,其他植物的CPS蛋白均存在同丹参一致的2个结构域。蒺藜苜蓿的CPS蛋白序列明显短于其他植物,这可能是导致它缺少terpene synthase N terminal domain的原因。
3.8 CPS蛋白三级结构的预测和分析蛋白质的功能与其三级结构密切相关,对蛋白质高级结构的预测和分析,有助于理解蛋白质结构与功能之间的相关性[18, 19]。利用Phyre2线串法(从头计算模式)对丹参CPS蛋白进行三级结构预测,结果如图 1所示。
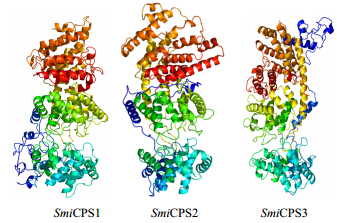 | 图 1 丹参CPS蛋白三维结构预测Fig. 1 3D structures prediction of SmiCPS in Salviae Miltiorrhizae Radix et Rhizoma |
利用基于PROCHECK程序的蛋白质结构检验工具PDBsum Generate对预测结果进行检测,计算得出Ramachandran图,结果如表 4所示,丹参SmiCPS2落在不允许区的比例刚好为1%,而SmiCPS1和SmiCPS3都大于1%,另外,三者氨基酸残基位于最佳区的比例都小于90%,不过分布在最佳区和次允许区的比例超过了90%,表明丹参CPS蛋白的空间构象虽然没有落在最有利区,但其构象是合理的,不过稳定性不足。另外,SmiCPS1、SmiCPS2和SmiCPS3的G-Factors值分别为-1.83、-0.09和-1.56,提示SmiCPS2的结构属于正常范围,SmiCPS1和SmiCPS3的结构十分罕见。
| 表 4 丹参及其他植物CPS蛋白三维结构稳定性预测 Table 4 Prediction of stability about 3D structures of CPS protein in Salviae Miltiorrhizae Radix etRhizoma and other plants |
对其余植物CPS蛋白进行同样的预测和检验(表 4),结果显示,所有植物CPS落在最佳区和次允许区的比例均超过了90%,其空间构像都是合理的;笋瓜和江南卷柏的CPS蛋白有超过90%的氨基酸残基落在最有利区;笋瓜、江南卷柏、巨冷杉和台湾杉的CPS蛋白落在不允许区的比例不到1%,空间结构稳定性最好。与SmiCPS2相似,迷迭香、蒺藜苜蓿、台湾杉、巨冷杉、江南卷柏和笋瓜的G-Factors值均大于-0.5,它们的空间结构都为正常结构,而其他植物CPS蛋白可能具有罕见的空间结构。笋瓜和江南卷柏每个预测值都较理想,提示这2个蛋白的空间结构真实性较高。
3.9 CPS蛋白序列同源比对分析及系统进化树构建利用blastall 2.2.26本地版软件将丹参CPS蛋白序列与其他植物进行同源比对(表 5),结果显示,丹参SmiCPS1蛋白与SmiCPS2、RoCPS和HaCPS具有较高相似性,SmiCPS2与RoCPS、TcaCPS和HaCPS具有较高相似性,而SmiCPS3与SdCPS、CaCPS和CmoCPS具有较高相似性。
| 表 5 丹参与其他植物CPS蛋白同源性比较 Table 5 Homology analysis of CPS protein in Salviae Miltiorrhizae Radix etRhizoma and other plants |
来源于同一祖先的不同植物在进化过程中的关系可以通过进化树来描述,通过构建植物进化树,可以了解一种植物在进化过程中的地位。本研究利用MEGA 5.0软件对丹参等16种植物的CPS蛋白序列进行系统进化树构建(图 2),结果显示,丹参SmiCPS1和SmiCPS2与同科植物迷迭香聚为一类,SmiCPS3先与玄参科植物野甘草聚为一类,又和唇形科植物毛萼香茶菜位于同一枝上。玄参科和唇形科都属于双子叶植物纲合瓣花亚纲管状花目,它们的亲缘关系较近。另外,西洋梨和同科植物一个苹果的新品种聚在一起;两个裸子植物巨冷杉和台湾杉聚在一起,又和蕨类植物江南卷柏聚为一类;蒺藜苜蓿单独成为一支,这可能是由于其序列过短,变异较大引起的。
 | 图 2 丹参及其他植物CPS蛋白序列的NJ树Fig. 2 NJ phylogenetic tree of CPS protein in Salviae Miltiorrhizae Radix et Rhizoma and other plants |
CPS是植物三环二萜类化合物生物合成过程中的起始环化酶,具有特殊作用,特别是SmiCPS,与丹参酮合成密切相关,正在受到越来越多的关注。
生物信息学是一门包括生物学、计算机科学、数学、统计学的交叉学科,被广泛应用于基因组学、蛋白质组学、系统生物学和比较基因组学,是一门备受关注的学科,是当今生命科学和自然科学的重大前沿领域之一,也是2l世纪自然科学的核心领域之一[20, 21]。采用各种分子生物学数据库和分析软件对已知的核酸和蛋白序列进行分析,从而推断及预测其结构和功能,已成为生物信息学研究的一种趋势[22]。通过生物信息学方法预测,能为实验验证指明方向,减少实验误区,有利于科学研究的成功率。
本实验利用生物信息学方法,将丹参作为主要分析对象,对包括蕨类植物、裸子植物和被子植物的13科16属16种植物的18条CPS蛋白进行了序列组成、生化特性、结构特点和功能等预测分析。蛋白序列理化性质分析表明,CPS蛋白为酸性蛋白,其中丹参SmiCPS2和笋瓜、向日葵、毛萼香茶菜的CPS蛋白为稳定类蛋白。导肽预测结果显示,丹参SmiCPS1和SmiCPS3以及巨冷杉、小粒咖啡、笋瓜、野甘草、番茄、可可和台湾杉的CPS蛋白可能具有叶绿体转运肽。结合信号肽预测结果,可推知CPS蛋白在游离核糖体上合成后,可能通过两种途径发挥作用,一是通过导肽进入叶绿体发挥作用;二是不进行蛋白转运,保留在细胞质基质中产生催化作用。这与萜类化合物的生物合成途径相符,文献报道,萜类化合物的生物合成途径有两条,位于细胞质基质中的甲羟戊酸途径(MVA)和位于质体中的MEP途径[23],叶绿体即是质体的一种。
蛋白质折叠时能形成亲水表面和疏水内核,并于潜在跨膜区出现高疏水值区域。本实验对丹参等植物CPS蛋白进行疏水性/亲水性预测,结果显示CPS蛋白不存在高疏水值区域,推测CPS蛋白可能不存在跨膜结构域。之后的跨膜结构域预测结果证实了这一推测。
目前,主要通过核磁共振(NMR)和X-射线晶体衍射(X-ray crystallography)等实验方法获得蛋白质的三维结构,但所需实验技术和设备费用要求都很高。借助计算机软件进行蛋白质高级结构预测可从一定程度上缓解这一难题。本文通过同源建模方法获得了丹参等植物CPS蛋白高级结构模型,并通过PROCHECK对所构建模型进行了检验,得到Ramachandran图。Ramachandran图用来评估模拟结构与自然结构的相同程度,能够反映蛋白质立体化学质量(stereochemical quality)[12]。根据构象的稳定性,Ramachandran图分为最佳区(most favoured regions)、次允许区(additional allowed regions)、一般允许区(generously allowed regions)和不允许区(disallowed regions)[13]。通常情况下,所预测模型氨基酸分布在最佳区的比例超过整个蛋白质的90%,可认为该蛋白氨基酸位于构象最有利区;超过90%的模型氨基酸落在最佳区和允许区,则表明所构建的空间构像是合理的;不允许区低于1%的模型结构空间稳定性较好[13, 15]。从Ramachandran图上可以看出,丹参等植物的CPS蛋白模型理论上都符合立体化学规则,丹参SmiCPS2、迷迭香、蒺藜苜蓿、台湾杉、巨冷杉、江南卷柏和笋瓜的CPS蛋白具有正常的空间结构,笋瓜、江南卷柏、巨冷杉和台湾杉的CPS蛋白空间结构稳定性最好。
利用生物信息学方法对丹参CPS蛋白序列的生理生化特性进行预测和分析,可以为CPS蛋白及其编码基因的克隆提供可靠的依据;对其序列结构的预测和分析,可为其蛋白表达与修饰提供指导;对其二级及高级结构的预测和分析有利于深入探讨该酶结构和功能之间的关系、作用机制和代谢过程。本研究的结论虽属推断和预测,但能为进一步深入研究丹参CPS蛋白的功能提供参考。
| [1] | 中国药典 [S]. 一部. 2010. |
| [2] | 张建虎, 叶芊. 丹参酮药理作用 [J]. 中华医学研究杂志, 2005, 5(6): 151-152. |
| [3] | Wang J W, Wu J Y. Tanshinone biosynthesis in Salvia miltiorrhiza and production in plant tissue cultures [J]. Appl Microbiol Biotechnol, 2010, 88(2): 437-449. |
| [4] | Cyr A, Wilderman P R, Determan M, et al. A modular approach for facile biosynthesis of labdane-related diterpenes [J]. J Am Chem Soc, 2007, 129(21): 6684-6685. |
| [5] | 秦双双, 陈新. 丹参次生代谢产物丹参酮的调控研究 [J]. 武汉工业学院学报, 2009, 28(4): 34-37. |
| [6] | 高伟, 崔光红, 孔建强, 等. 丹参柯巴基焦磷酸合酶基因的优化表达、纯化及抗体制备 [J]. 药学学报, 2008, 43(7): 766-772. |
| [7] | Zhou Y J, Gao W, Rong Q X, et al. Modular pathway engineering of diterpenoid synthases and the mevalonic acid pathway for miltiradiene production [J]. J Am Chem Soc, 2012, 134(6): 3234-3241. |
| [8] | Emanuelsson O, Nielsen H, Brunak S, et al. Predicting subcellular localization of proteins based on their n-terminal amino acid sequence [J]. J Mol Biol, 2000, 300(4): 1005-1016. |
| [9] | Petersen T N, Brunak S, von Heijne G, et al. SignalP 4. 0: discriminating signal peptides from transmembrane regions [J]. Nat Methods, 2011, 8(10): 785-786. |
| [10] | Letunic I, Doerks T, Bork P. SMART 7: recent updates to the protein domain annotation resource [J]. Nucl Acids Res, 2012, 40(Database issue): 302-305. |
| [11] | Kelley L A, Sternberg M J. Protein structure prediction on the web: a case study using the Phyreserver [J]. Nat Protoc, 2009, 4(3): 363-371. |
| [12] | 董娇, 周军, 辛培尧, 等. 不同植物LDOX/ANS基因的生物信息学分析 [J]. 基因组学与应用生物学, 2010, 29(5): 815-822. |
| [13] | 龙芳, 李绍鹏, 李茂富. 7种植物ALAD基因的生物信息学分析 [J]. 基因组学与应用生物学, 2013, 32(6): 802-814. |
| [14] | 韦雪芳, 王冬梅, 刘思, 等. 信号肽及其在蛋白质表达中的应用 [J]. 生物技术通报, 2006(6): 38-42. |
| [15] | 薛庆中. DNA和蛋白质序列数据分析工具 [M]. 北京: 科学出版社, 2012. |
| [16] | 薛永常, 聂会忠, 刘长斌. 木质素合成酶C3H基因的生物信息学分析 [J]. 生物信息学, 2009, 7(1): 13-17. |
| [17] | 王镜岩, 朱圣庚, 徐长法. 生物化学 [M]. 北京: 高等教育出版社, 2002. |
| [18] | 蔡娜娜, 陈月辉, 李伟. 基于神经网络的蛋白质三级结构预测 [J]. 计算机工程, 2010, 36(9): 176-177. |
| [19] | 陈克克, 武雪. 植物查耳酮异构酶生物信息学分析 [J]. 生物信息学, 2009, 7(3): 163-167. |
| [20] | Mount D W. 生物信息学 [M]. 曹志伟, 译. 北京: 科学出版社, 2006. |
| [21] | 许忠能. 生物信息学 [M]. 北京: 清华大学出版社, 2008. |
| [22] | 王汉屏. 不同植物防御素的生物信息学分析 [J]. 植物生理学通讯, 2008, 44(1): 25-32. |
| [23] | Osbourn A E, Lanzotti V. Plant-derived Natural Products [M]. New York: Springer US, 2009. |
 2015, Vol. 46
2015, Vol. 46


