2. 中国科学院藏药研究重点实验室, 青海 西宁 810008;
3. 中国科学院大学, 北京 100049;
4. 青海省铁卜加草原改良试验站, 青海 共和 813000
2. Key Laboratory of Tibetan Medicine Research, Chinese Academy of Sciences, Xining 810008, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Tiebujia Grassland Improvement Experiment Station, Gonghe 813000, China
遗传多样性是生物种内表现在从DNA分子到形态特征各个层次上可遗传所有变异的总称,是濒危植物保护生物学研究的核心之一。一个种群遗传多样性越丰富,对环境的适应能力就越强,越容易扩展其分布范围和开拓新的环境。可见物种或种群进化潜力和适应环境的能力取决于遗传多样性的大小。对濒危物种遗传多样性和群体遗传结构的研究是揭示其适应潜力的基础,为进一步探讨濒危物种的濒危机制和制定相应的保护措施提供科学依据[1]。
羌活Notopterygium incisum Ting ex H. T. Chang是我国特有的羌活属的多年生草本植物[2, 3],与同属的宽叶羌活N. franchetii H. de Boiss. 同为药材羌活的基原植物,以根茎及根入药[4],主要分布在四川、青海、甘肃等省的高寒山区[5, 6]。羌活是中、藏、羌医药体系中常用药材,主要含挥发油和香豆素类成分;具散寒祛风、除湿止痛之功效,用于风寒感冒头痛、风湿麻痹、肩背酸痛等症[7]。现代药理研究表明其对心脑血管疾病也有确切疗效。目前,用羌活的中(藏)成药有200余种,用药需求量非常大。羌活多生长于海拔较高的地方,垂直分布范围在海拔1 700~5 000 m,其种子发育期较短,成熟度低,种群更新困难[6]。此外,由于资源短缺使得羌活药材价格逐渐攀升,使羌活遭受掠夺性采挖,该药用植物的生境片段化严重,生物多样性丧失以及资源利用的无序性和破坏性造成其野生资源濒临灭绝,1987年即被国务院颁布的《中国野生药材资源保护管理条例》列为III级保护物种,2005年又载入《中国物种红色名录》[8]。目前,羌活植物化学[9, 10]、药理[11]、地理分布[12]、分类学[2, 3]、环境土壤学[13]、核型和驯化栽培[14, 15]等方面的研究比较多,但从DNA分子水平上对羌活群体遗传结构、遗传分化及多样性等方面的研究较少[16],唐学芳等[17]用RAPD标记研究了四川西部4个居群33株羌活的遗传分化,但由于其样品数量有限还不能反映羌活群体遗传状况全貌,需要更多的分子生物学标记研究结果给以补充。
由于叶绿体DNA(cpDNA)trnT-trnL间隔区属于cpDNA非编码区,在植物中具有稳定性,经常被用来分析植物的种群遗传变异和系统地理学问题。另外,技术上选择DNA测序的方法还可以避免在利用限制片段长度多态性(RFLP)和基于PCR的指纹图谱法时经常遇到的长度同塑性(length homoplasy)问题,从而提高估算种群遗传结构和基因流的分辨率[18]。本研究利用cpDNA为分子标记,对羌活31个种群的遗传多样性和遗传结构进行分析,旨在了解羌活现存种群的遗传变异水平,阐述种群遗传信息对物种保护的重要意义,为保护植物遗传资源提供依据。
1 材料与方法 1.1 材料本实验选取的31个居群为2014年6月至9月分别采自青海、四川和甘肃3省区,基本包括了我国羌活主要产区(表 1)。采样时每个居群选取成年植株4~17株,根据随机取样原则,取样的羌活株距在50 m以上。野外采集新鲜、完整的植物幼嫩叶片,置于盛有硅胶的塑胶袋中干燥保存。供试材料由中国科学院西北高原生物研究所周国英研究员鉴定为羌活Notopterygium incisum Ting ex H. T. Chang,凭证标本存放于中国科学院西北高原生物研究所。
| 表 1 31个羌活居群的经度、纬度、海拔及Hd和Pi Table 1 Longitude, latitude, altitude, cpDNA Hd, and Pi averaged across loci of 31 N. incisum populations |
依据改良的CTAB法从硅胶干燥的叶片中提取总DNA。通过测定紫外光吸收值来确定DNA浓度和纯度。利用0.8%琼脂糖凝胶电泳检查DNA完整性。
1.2.2 PCR扩增与DNA测序对cpDNA的trnT-trnL片段进行扩增,使用通用引物trnT:5’-CATTACAAATGCGATGCTCT-3’和trnL:5’-TCTACCGATTTCGCCATATC-3’;扩增反应在Eppendorf Mastercycler Gradient PCR仪上进行,反应体系为25 μL,内含25 mmol/L MgCl2 2 μL、2.5 mmol/L dNTPs 2 μL、10 μmol/L 引物各0.6 μL、25 ng 模板、10×缓冲液 2.5 μL和1 U Taq DNA聚合酶(TaKaRa)。扩增程序为:95 ℃预变性2 min,94 ℃变性30 s,53 ℃退火45 s,72 ℃延伸1 min 45 s,共5个循环,每个循环退火温度降1 ℃;94 ℃预变性30 s,48 ℃退火45 s,72 ℃延伸1 min 45 s,共30个循环;72 ℃延伸7 min,16 ℃保温。将PCR扩增产物经电泳检测纯化后由上海美吉生物医药科技有限公司测序。
1.3 数据分析用Clustal X软件进行序列对位排列,并加以手工适当校正。在所有序列校对正确后,用DnaSP4.0程序统计cpDNA单倍型,计算各居群的核苷酸多样性(Pi)、单倍型多样性(Hd)、居群间基因流值(Nm)。居群内和居群间的基因流用每代雌性个体迁移数(FST)估计,该值可由公式FST=1/(1+2 Nm)求出,其中N是有效居群大小雌性数目,m是雌性迁移率。选用Arlequin软件进行分子方差分析(AMOVA),计算居群内、居群间的变异方差分布。用MEGA4.1软件进行核苷酸组成分析,采用Kitmura-2-Parameter distance双参数模型(Kimura,1980)计算遗传距离,并用邻接(Neighbour-Joining,NJ)法,通过1 000次重复获得的自展检验(bootstrap)数值标记在分支上。
2 结果与分析 2.1 序列变异对羌活31自然居群245个个体的cpDNA trnT-trnT基因间片段进行了测序和比对。序列长度在753~847 bp,排序后长915 bp。合并相同的序列共得到38个单倍型(H1~H38),单倍型序列注册于GenBank数据库中。通过统计这一片段所有个体的序列发现,碱基A与T在整个序列中所占比例为72.72%,这与大多数叶绿体DNA基因间区碱基组成成分相符[19, 20]。
2.2 遗传多样性种群遗传多样性参数统计表明(表 1),在物种水平上,羌活Hd=0.873,Pi=0.004 07;在居群水平上 Hd的变化范围为0~0.900,Pi变化范围为0~0.054 4;其中四川红原居群的Hd最高(0.900),四川马尔康居群的Pi最高(0.054 4),而青海互助居群、青海门源居群、青海兴海居群、青海玛沁居群、青海久治居群、青海达日居群、青海囊谦居群、甘肃山丹居群、甘肃合作居群、甘肃卓尼居群、甘肃临潭居群、四川若尔盖居群、四川丹巴居群这些居群中的每一个居群都是由相同的序列组成,因此其Hd和Pi都最低。
2.3 居群的遗传结构基于羌活cpDNA trnT-trnL非编码区序列变异的AMOVA分析表明,羌活大部分的遗传变异发生在居群间(77.55%),只有22.45%的遗传变异发生在居群内,居群间的遗传分化大于居群内的分化,说明群体间变异是羌活的主要变异来源。由FST估算的居群间的Nm为0.145,表明羌活各居群间的基因流较低。
2.4 居群间的系统发育关系根据不同单倍型之间的遗传距离,以伞形科(Umbelliferae)的Rhodosciadium argutum (Wall. ex Hook.) Ching(登录号JQ304919)为外类群,构建羌活38个不同单倍型的NJ分子系统树(图 1),NJ树上各分支上的数字代表 1 000次Bootstrap统计分析后对该支的支持百分比(即置信度)。NJ树聚为3大分支:Q9、Q10、Q11、Q12、Q13、G5、G6、G7、S2、S3、S4、S5、S6、S7、S8、S9聚为一支,而Q1、Q2、Q3、Q4、Q5、Q6、Q7、G2、G3、G4、G8、G9、S1、S6、S9聚为另一支,外类群的为第3支。由图 1可见,S1居群(四川若尔盖)与同一个地理区域的群体(S2~S10)并未聚成一支,且S6、S9等多个居群由于单倍型的共享在聚类时出现交叉重叠。
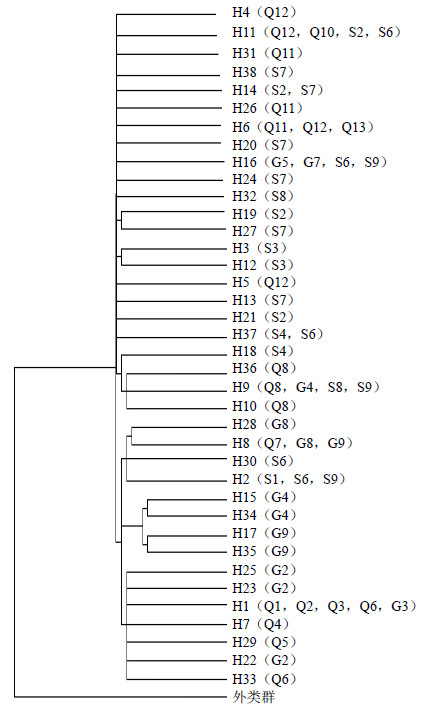 | 图 1 羌活cpDNA trnT-trnL单倍型NJ树Fig.1 NJtree based on cpDNA trnT-trnL haplotypes of N. incisum |
普遍认为稀有的或分布区狭窄的物种遗传多样性水平偏低[21, 22, 23],但近年来也有研究报道显示有些特有种、狭域种甚至濒危种也能保持较高水平的遗传多样性[24, 25]。本实验通过对濒危植物羌活31个自然居群的叶绿体DNA(cpDNA)trnT-trnL分析得到其物种水平的Hd、Pi分别为0.873和0.004 07,表明羌活物种水平上的遗传多样性相对较高。与同属的宽叶羌活[26]相比,其物种水平的遗传多样性稍低于宽叶羌活(Hd=0.750 3,Pi=0.007 111)。但其遗传多样性又略高于其他濒危物种[27, 28, 29],说明羌活的遗传多样性处于中等水平。
遗传变异水平受多方面因素影响,诸如生活型、繁殖方式、基因流、遗传漂变及自然选择和人为干扰等。羌活的遗传多样性处于中等水平,可能与生境的严重片段化以及本身的生长特性有关。羌活是多年生草本,其分布范围相对较广,属异花授粉植物,有利于维持较高的遗传多样性水平。但是近几年来,由于资源短缺使得羌活药材价格逐渐攀升,使羌活遭受掠夺性采挖,羌活在人为的破坏中经历了严重的瓶颈效应,居群的大量减少及各居群间相互孤立,个体数量稀少的居群内容易发生近交,增加纯合度,造成居群内的遗传多样性的减少。虽然珍稀濒危物种一般表现出低水平的遗传变异,但遗传变异水平的高低并不一定与濒危相关,濒危的结果之一是遗传多样性水平下降,低水平的遗传多样性使物种更加濒危。羌活在遗传多样性上没有表现出濒危特性,需要加强其生物学特点、生殖生态、传粉生态等方面的研究,以便揭示濒危的机制。
3.2 羌活的遗传结构一个物种或群体的进化潜力,在很大程度上取决于它的居群遗传结构[30]。而物种的遗传结构是指该物种遗传多样性在种群内和种群间的分布,即遗传分化。一般情况下,对于濒危物种来说,居群间的遗传变异相对较大,分化明显,如中国疵粒野生稻[31]、银杉[32]、宽叶羌活[26]、独一味[33]等。本研究中AMOVA数据表明羌活的遗传变异主要分布在居群间(77.55%),也显示了相似的结果。植物种群遗传结构一般解释为交配系统、种子传播方式、生活史、分布区大小等因素综合作用的结果。与Nybom[34]对116种植物种群遗传分化系数统计分析的平均值比较,羌活野生种群遗传分化系数(0.775 5)高于自交系统植物(0.65),这与羌活异花授粉的特征不符,可能是与青藏高原独特的地形和气候有关。在青藏高原内部千山万壑、峡谷纵横,而羌活主要分布于青藏高原的高山林缘下和灌丛中,青藏高原独特而复杂的地形通过阻断种子或花粉的传播来阻止居群间的交流,居群间有限的Nm(0.145)进一步证明了这一点。因此,青藏高原地区的地理格局可能导致了羌活居群的隔离,加大了居群间的遗传分化。对大钟花Megacodon stylophorus (C. B. Clarke) H. Smith [35]、大花红景天Rhodiola crenulata (Hook. f. & Thomson) H. Ohba [36]、独一味Lamiophlomis rotat (Benth. ex Hook. f.) Kudo [32]青藏高原植物居群的遗传多样性及遗传分化研究也表明,分布地区内复杂的地理条件,往往是导致居群隔离,促进居群间遗传分化的原因。此外,作为传统的藏药,近年来的人为过度采挖,加上人类活动对自然生境的破坏,导致羌活生境的破碎化,阻断了居群间的基因交流,进一步加剧了居群的遗传分化。
3.3 羌活的保护探讨物种濒危机制是当前生物多样性研究的热点之一,也是保护生物学的核心工作。本研究表明,尽管羌活物种和种群都维持中等水平的遗传变异,但由于残存的羌活野生种群很小、片段化分布、种群内个体散生等种群特征,会产生遗传漂变和近交衰退的遗传后果,将可能导致遗传多样性降低、有害等位基因积累和适合度降低,最终使物种面临更高的灭绝风险,因此,开展羌活遗传资源保护工作迫在眉睫。基于本研究结果,羌活物种水平上具较中等的遗传多样性,且主要存在于居群间,居群分化明显,提出以下4点保护策略:(1)保护好其赖以生存的生境;(2)保护尽可能多的居群和个体;(3)可以在9月中下旬人工采集部分成熟种子撒播到其他居群中,以便加强居群间的基因流动;(4)在迁地保护采样过程中,在各个居群内可以少取样,但要在尽可能多的居群中取样,以便提高栽培居群的遗传多样性。
本研究是基于叶绿体DNA(cpDNA)trnT-trnL序列来研究羌活种群遗传多样性与遗传结构,以期探讨其濒危机制。虽然叶绿体基因能反映基因多态性及基因流动大小等种群遗传学参数,也能反映羌活种群叶绿体的变异模式,但由于单基因片段所能反映的遗传多样性有限,因此,建议进一步开展多基因联合和核基因组ISSR、RAPD、AFLP等标记来检测羌活的遗传多样性和遗传结构,并结合生殖生态学、生殖解剖学研究,以期获得更多、更可靠的羌活种群遗传多样性及其濒危信息,为科学制定羌活遗传资源保护策略提供更全面的理论依据。
| [1] | Hamrick J L, Godt M J M, Murawski K A. Correlations between species traits and Allozyme Diversity implications for Conservation Biology[M]. New York: Oxford University Press, 1991. |
| [2] | 溥发鼎, 王萍莉, 郑中华, 等. 重订羌活属的分类[J]. 植物分类学报, 2000, 38(5): 430-436. |
| [3] | 王幼平, 溥发鼎, 王萍莉, 等. 中国特有属——羌活属的系统分类研究[J]. 云南植物研究, 1996, 18(4): 424. |
| [4] | 中国药典[S]. 一部. 2010. |
| [5] | 吴征镒. 论中国植物区系的分区问题[J]. 云南植物研究, 1979, 1(1): 1-22. |
| [6] | 周 毅, 蒋舜媛, 马小军, 等. 羌活资源危机和保护[J]. 中草药, 2003, 34(10): 12-14. |
| [7] | 徐惠波, 孙晓波. 羌活挥发油的药理作用研究[J]. 中草药, 1991, 22(1): 28-30. |
| [8] | 汪 松, 解 焱. 中国物种红色名录 (第1卷)[M]. 北京: 高等教育出版社, 2004. |
| [9] | 刘卫根, 王亮生, 徐文华, 等. 不同商品等级羌活中有机酸和香豆素类化合物的测定[J]. 中成药, 2012, 34(11): 2181-2186. |
| [10] | 刘卫根, 王亮生, 周国英, 等. 羌活不同部位有机酸和香豆素类化合物含量的比较研究[J]. 药物分析杂志, 2012, 32(11): 1950-1956. |
| [11] | 张明发, 沈雅琴. 羌活药理学研究[J]. 药学服务, 2008(5): 28-30. |
| [12] | 马玉林, 李建民, 马 莉, 等. 药用植物羌活的研究进展[J]. 安徽农业科学, 2010, 38(24): 13092-13093. |
| [13] | 蒋舜媛, 孙 辉, 黄雪菊, 等. 羌活和宽叶羌活的环境土壤学研究[J]. 中草药, 2005, 36(6): 917-921. |
| [14] | 董生健. 野生羌活驯化栽培技术[J]. 农业科技与信息, 2010(1): 38-39. |
| [15] | 方子森, 陈小莉, 张恩和. 野生羌活的生态环境与驯化栽培[J]. 中草药, 2004, 35(9): 1071-1073. |
| [16] | 孙稚颖, 陈士林, 姚 辉, 等. 基于ITS2序列的羌活及其混伪品的DNA条形码鉴定[J]. 中草药, 2012, 43(3): 568-571. |
| [17] | 唐学芳, 蒋舜媛, 孙 辉, 等. 川产羌活种质遗传多样性的RAPD分析[J]. 中草药, 2008, 39(9): 1387-1392. |
| [18] | Chiang T Y, Chiang Y C, Chen Y J, et al. Phylogeography of Kandelia candel in East Asiatic mangroves based on nucleotide variation of chloroplast and mitochondrial DNAs[J]. Mol Ecol, 2001, 10(11): 2697-2710. |
| [19] | Kokita T and Nohara K. Chloroplast DNA phylogeography of Rhodiola alsia (Crassulaceae) in the Qinghai-Tibet Plateau[J]. Botany-Botanique, 2009, 87(11): 1077-1088. |
| [20] | Zheng W, Wang L Y, Meng L H, et al. Genetic variation in the endangered Anisodus tanguticus (Solanaceae), an alpine perennial endemic to the Qinghai-Tibetan Plateau[J]. Genetica, 2008, 132(2): 123-129. |
| [21] | 李 昂, 葛 颂. 植物保护遗传学研究进展[J]. 生物多样性, 2002, 10(1): 61-71. |
| [22] | Richter T S, Soltis P S and Soltis D E. Genetic-variation within and among populations of the narrow endemic, delphinium-viridescens (Ranunculaceae)[J]. Am J Bot, 1994, 81(8): 1070-1076. |
| [23] | 苏志龙, 殷寿华, 吴成军, 等. 濒危物种山红树居群遗传结构的RAPD分析[J]. 云南植物研究, 2005, 27(2): 181-186. |
| [24] | 张颖娟, 王玉山. 濒危灌木长叶红砂遗传多样性的RAPD分析[J]. 生态学杂志, 2008, 27(2): 167-171. |
| [25] | 张永明, 金 洪, 马万里, 等. 濒危植物绵刺8个种群遗传多样性的AFLP分析[J]. 生态学报, 2009, 29(5): 2688-2692. |
| [26] | 杨路存, 周国英, 聂学敏. 濒危植物宽叶羌活天然居群cpDNA非编码区多态性分析[J]. 西北植物学报, 2013, 33(8): 1535-1543. |
| [27] | Tan B, Liu K, Yue X L, et al. Chloroplast DNA variation and phylogeographic patterns in the Chinese endemic marsh herb Sagittaria potamogetifolia[J]. Aquatic Bot, 2008, 89: 372-378. |
| [28] | Huang J, Wang W, Peng C, Chiang T. Phylogeography and conservation genetics of Hygrophila pogonocalyx (Acanthaceae) based on atp B-rbcL noncoding spacer cpDNA[J]. J Plant Res, 2005, 118: 1-11. |
| [29] | Ribeiro R A, Lemos J P, Ramos A C S, et al. Phylogeography of the endangered rosewood Dalbergia nigra (Fabaceae): insights into the evolutionary history and conservation of the Brazilian Atlantic Forest[J]. Heredity, 2011, 106: 46-57. |
| [30] | Brown A H D, Clegg M T, Kahler A L, et al. Plant Population Genetics, Breeding and Genetic Resources[M]. Sunderland: Sinauer Associates Press, 1990. |
| [31] | 钱 韦, 葛 颂, 洪德元. 采用RAPD和ISSR标记探讨中国疣粒野生稻的遗传多样性[J]. 植物学报, 2000, 42(7): 741-750. |
| [32] | 汪小全, 邹喻苹, 张大明, 等. 银杉遗传多样性的RAPD分析[J]. 中国科学 (C辑), 1996, 26(5): 436-441. |
| [33] | Liu J M, Wang L, Geng Y P, et al. Genetic diversity and population structure of Lamiophlomis rotata (Lamiaceae), an endemic species of Qinghai-Tibet Plateau[J]. Genetica, 2006, 128(1/3): 385-394. |
| [34] | Nybom H. Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants[J]. Mol Ecol, 2004, 13(5): 1143-1155. |
| [35] | Ge X J, Zhang L B, Yuan Y M, et al. Strong genetic differentiation of the East-Himalayan Megacodon stylophorus (Gentianaceae) detected by inter-Simple sequence repeats (ISSR)[J]. Biodi Cons, 2005, 14(4): 849-861. |
| [36] | Lei Y D, Gao H, Tsering T, et al. Determination of genetic variation in Rhodiola crenulata from the Hengduan Mountains Region, China using inter-simple sequence repeats[J]. Genet Mol Biol, 2006, 29(2): 339-344. |
 2015, Vol. 46
2015, Vol. 46


