2. 北京中医药大学基础医学院, 北京 100191
2. School of Basic Medical Sciences, Beijing University of Chinese Medicine, Beijing 100191, China
丹参酮(tanshinone)又称总丹参酮,是丹参Salvia miltiorrhiza Bunge中的脂溶性二萜醌类化学成分,包括丹参酮I、丹参酮IIA、丹参酮IIB、隐丹参酮、异隐丹参酮等10余种化合物。这些化合物在改善冠状动脉供血、抑制血小板聚集、消炎、抑菌及抗肿瘤等方面疗效显著,同时具有抗氧化和提高耐缺氧能力等药理作用[1,2,3]。丹参酮的独特功效使其成为国际上广泛认可的有效治疗心脑血管疾病的天然药物之一。随着人类生活水平的不断提高和饮食结构的逐渐变化,心脑血管疾病已位居威胁人类健康的重大疾病之首,由此导致对丹参酮的需求量剧增。天然存在的丹参酮主要分布于丹参属植物中,丹参是其主要来源。然而,丹参的资源有限,并且其中丹参酮的量低,造成丹参酮的产量不能满足日益增长的临床需要。研究人员试图通过干预丹参酮生物合成过程达到提高丹参中丹参酮量的目的,因此,对丹参酮的生物合成途径及其关键酶进行了大量的研究。本文对这方面的研究进展进行了总结与展望。 1 生物合成途径
丹参酮属于二萜醌类化合物,而所有的天然萜类化合物都来自2个基本的五碳通用前体,即异戊烯基焦磷酸(IPP)和二甲烯丙基焦磷酸(DMAPP)[4]。 然而,生物合成却有2条截然不同的途径(图 1),一条是经典的甲羟戊酸(MVA)途径[5],另一条为丙酮酸/磷酸甘油醛(DXP)途径[6]。这2条途径分别在不同的亚细胞区域内进行,MVA途径位于细胞质中,DXP途径位于质体中,并且这2条途径中所涉及的酶和基因也完全不同。但是,这2条途径却通过IPP和DMAPP联系在一起[7]。表 1中列出了目前发现的这2条途径中关键酶基因的基本信息。
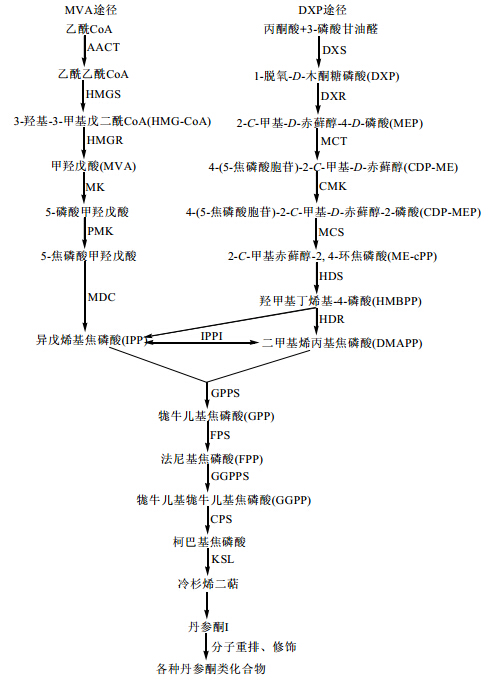 | 图 1 丹参酮的生物合成途径 Fig. 1 Biosynthesis pathway of tanshinone |
| 表 1 MVA和DXP途径中关键酶基因的基本信息 Table 1 Basic information of key enzyme genes in MVA and DXP pathways |
这2条途径中关键酶基因的研究结果表明,丹参酮类化合物的生物合成途径主要是DXP代谢途径,同时也受MVA途径的影响。 2 MVA途径及其关键酶 2.1 乙酰CoA酰基转移酶(AACT)
AACT是生物合成萜类化合物MVA途径的起始酶,其作用是将2分子的乙酰CoA缩合生成乙酰乙酰CoA[32]。崔光红等[8]利用cDNA微阵列和RACE策略克隆得到了SmAACT基因,其cDNA全长1 623 bp,含有1 200 bp开放阅读框,编码399个氨基酸。通过对其序列进行同源性和系统发育分析,结果表明该基因具有乙酰CoA酰基转移酶的重要结构域。同时,利用实时荧光定量PCR技术对SmAACT基因在丹参植株不同器官内的表达以及在毛状根中的诱导表达进行了分析。结果表明,其在丹参根、茎、叶中均有表达,在根中的表达量最高,并且其表达量受生物和非生物诱导子Ag+和酵母菌的诱导,并伴随丹参酮的积累。SmAACT基因单核苷酸多态性分析表明,其在不同的个体中存在着大量的单核苷酸变异位点,且部分位点表现出产地特异性。 2.2 3-羟基-3-甲基戊二酰CoA合成酶(HMGS)
HMGS将乙酰乙酰CoA与乙酰CoA缩合生成3-羟基-3-甲基戊二酰CoA(HMG-CoA)[33]。王敬[11]成功地从丹参中克隆出编码该蛋白的基因,命名为SmHMGS,并进行了生物信息学和组织特征表达分析。氨基酸多重序列比对分析显示HMGS在不同物种中表现出高度的保守性,说明其在进化过程中是相对稳定的。组织特征表达分析结果表明SmHMGS为组成型表达基因,在叶中表达最高,茎中稍低,根中最弱。随后还考察了不同诱导子对SmHMGS基因表达的影响,结果表明茉莉酸甲酯(MJ)和酵母提取物(YE)能够较好地诱导SmHMGS基因的表达,并伴随着丹参酮的积累。 2.3 3-羟基-3-甲基戊二酰CoA还原酶(HMGR)
在HMGR的催化下,HMG-CoA生成MVA。该反应是MVA途径中的限速反应[34]。廖攀等[14]在2009年第1次成功地从丹参中克隆出编码该蛋白的基因,命名为SmHMGR。其cDNA全长为2 115 bp,包含1 695 bp的开放阅读框,编码565个氨基酸。氨基酸多重序列比对显示SmHMGR与其他植物的HMGR有较高的同源性,和其他植物的HMGRs一样,包含2个跨膜结构域和催化结构域。进化树分析表明,其与胡黄连HMGR的亲缘关系最近。组织表达谱分析表明,SmHMGR是一个组成型表达的基因,在丹参根中表达最强,在茎中次之,在叶中最弱,水杨酸(SA)和MJ能够上调其表达。同样Dai等[35]利用RACE技术从丹参中克隆得到了HMGR基因,将其命名为SmHMGR2,其cDNA全长为1 959 bp,开放阅读框长度为1 653 bp,编码550个氨基酸。序列比对分析和进化树分析表明,SmHMGR2与苍术的HMGR同源性最高,组织特征表达分析表明其在叶、茎、根中的表达较强。同时,实验结果表明上调SmHMGR2的表达伴随着丹参酮量的增加。
在甲羟戊酸激酶(MK)及磷酸甲羟戊酸激酶(PMK)催化的连续2步磷酸化步骤中,MVA生成5-焦磷酸甲羟戊酸(MD),随后,MD在5-焦磷酸甲羟戊酸脱羧酶(MDD)催化作用下脱羧生成IPP[36,37]。 3 DXP途径及其关键酶 3.1 脱氧-D-木酮糖-5-磷酸合成酶(DXS)
该途径最初的前体物质是3-磷酸甘油醛(G3P)和丙酮酸(PA),二者在DXS催化作用下生成1-脱氧-D-木酮糖磷酸(DOXP),DXS所催化的反应是该途径中的第1步限速反应[38,39]。王敬[11]成功地克隆出了丹参的2个DXS成员,分别命名为SmDXS1和SmDXS2,并且表明SmDXS1是组成型表达的基因,在叶中表达最强,茎中稍弱,根中最弱。SmDXS2基因不是组成型基因,只在根部表达,在茎和叶中检测不到其表达。 3.2 DXP还原异构酶(DXR)
在DXR作用下,DXP经原子重排和还原生成2-C-甲基-D-赤藓醇-4-D-磷酸(MEP),DXR是DXP途径中的第2步催化酶,也是DXP途径重要的限速酶[40]。Yan等[41]从丹参中克隆得到了SmDXR基因,通过生物信息学分析证明其与其他植物具有很高的同源性,进化树分析表明其和番茄的亲缘关系较近,在根、茎、叶中均有表达,属于组成型基因。通过诱导子分析表明,其在叶片中能够被SA所诱导,而在根、茎、叶中则被MJ所阻遏。Wu等[42]利用DXR功能缺陷的大肠杆菌菌株互补实验鉴定了SmDXR蛋白的酶活性,并首次报道在渗透压和YE处理后,SmDXR转录水平表达量提高的同时,丹参酮量也提高,暗示SmDXR可能参与控制代谢流流动,并且可能是丹参酮生物合成代谢调控的靶点。 3.3 CMK、MCS、HDR
在2-甲基-D-赤藓醇-4-磷酸胱氨酰转移酶(MCT)作用下,MEP和5-焦磷酸胞苷(CDP)缩合生成4-(5-焦磷酸胞苷)-2-C-甲基-D-赤藓醇(CDP-ME)[43],然后,CDP-ME在4-(5-焦磷酸胞苷)-2-C-甲基-D-赤藓醇激酶(CMK)催化作用下磷酸化生成4-(5-焦磷酸胞苷)-2-C-甲基-D-赤藓醇-2-磷酸(CDP-MEP)[44]。接下来,在2-C-甲基赤藓醇-2,4-环焦磷酸合成酶(MCS)作用下,CDP-MEP转化为2-C-甲基赤藓醇-2,4-环焦磷酸(ME-cPP)[45]。在上述3步酶促反应中,王学勇等[20]从丹参毛状根中克隆得到了中间一步反应的CMK酶基因(命名为SmCMK),并且初步证明了SmCMK基因表达量与丹参酮类成分积累之间的关系。高伟等[22]采用RACE法克隆得到了SmMCS基因,其cDNA全长988 bp,编码234个氨基酸,并且,其表达受Ag+的诱导。之后,ME-cPP在羟甲基丁烯基-4-磷酸合成酶(HDS)催化作用下生成羟甲基丁烯基-4-磷酸(HMBPP)[46],最后HMBPP在1-羟基-2-甲基-2-(E)-丁烯基-4-焦磷酸还原酶(HDR)催化下转化为IPP和DMAPP[47]。程琪庆等[25]成功地从丹参中克隆得到了SmHDR基因,其由1 647个核苷酸组成,编码463个氨基酸,序列比对和系统进化分析表明SmHDR与其他植物的HDR家族具有较高的同源性,和库洛胡黄连HDR的亲缘关系较近。并且,受Ag+诱导后其表达水平升高,同时伴随着丹参酮类成分量的增加,说明其可能参与丹参酮的生物合成。而后,IPP和DMAPP在IPP异构酶(IPPI)的作用下相互转化,实现了MVA途径和DXP途径之间的交流。 4 下游途径及关键酶 4.1 牻牛儿基牻牛儿基焦磷酸环化酶(GGPPS)
GGPPS催化法尼基焦磷酸(FPP)生成牻牛儿基牻牛儿基焦磷酸(GGPP),而GGPP是多种初级和次级代谢产物的共同前体,因此GGPPS成为丹参酮代谢途径中的关键酶。廖攀[14]成功地克隆出SmGGPPS基因,其cDNA全长1 234 bp,编码364个氨基酸。氨基酸多重序列比对显示SmGGPPS与其他植物的GGPPS有较高的同源性。进化树分析表明其与其他GGPPS蛋白都来自同一祖先,组织特异性分析表明SmGGPPS是一个组成型表达的基因,在叶片和根中表达较强,在茎中表达较弱。SmGGPPS在丹参叶片中受到SA的诱导,但在丹参根、茎、叶中均不受MJ的诱导。张蕾等[48]对二萜类物质代谢途径中的共同前体合酶GGPPS进行了研究,克隆得到一条cDNA序列,其全长1 298 bp,开放阅读框为1 095 bp,编码364个氨基酸,包含一段由52个氨基酸组成的质体定位信号肽。同时对其组织表达特异性进行了分析,结果表明该基因为组成型表达的基因,在根、茎、叶等多个组织的多个时期中均有表达,尤以花期叶片的表达最强。 4.2 柯巴基焦磷酸合酶(CPS)和类贝壳杉烯合酶(KSL)
CPS、KSL是丹参酮GGPP下游生物合成途径的2个关键酶基因。高伟等[30]从丹参中克隆得到了SmCPS和SmKSL。SmCPS的cDNA全长为2 688 bp,包含了一个2 382 bp的开放阅读框,编码793个氨基酸,氨基酸序列N端含有“DXDD”基序的结构功能域,推测其具有起始环化GGPP的功能。通过对SmCPS的mRNA表达进行分析,证明了该基因参与了丹参酮类成分的生物合成。通过对SmCPS基因的表达条件进行优化,制备了SmCPS的多克隆抗体[30]。SmKSL的cDNA全长共2 110 bp,包含了一个1 788 bp的开放阅读框,编码595个氨基酸,其氨基酸序列C端含有“DDXXD”基序的结构域,推测其具有磷酸离子化底物的功能,其mRNA表达分析结果说明该基因参与丹参酮类成分的生物合成。Cheng等[49]利用实时荧光定量PCR技术考察了诱导子对SmCPS基因表达的影响,实验结果表明,诱导子能够显著提高SmCPS的表达。该作者同时还考察了诱导子对丹参酮量的影响,发现YE+Ag+,Ag++MJ和YE+Ag++MJ诱导子的组合能够提高毛状根中隐丹参酮和二氢丹参酮的量。 5 展望
丹参酮是目前国际上广泛认可的治疗心脑血管疾病的有效天然药物之一,而心脑血管疾病是人类健康的“头号杀手”,因此,丹参酮的市场需求量很大。由于丹参酮药源匮乏问题使其临床应用受限。丹参毛状根的培养被认为是获得丹参有效成分的重要途径,且这方面的研究也较成熟[50]。然而丹参毛状根中有用的次生代谢产物量较低等原因阻碍了利用丹参毛状根培养生产丹参酮等有用次生代谢产物的工业化。通过基因工程技术,改造并克隆丹参酮生物合成途径中的关键酶基因并将其导入丹参植株内,获得高产丹参酮的丹参毛状根或再生植株,并对其进行大规模培养是进一步提高丹参酮产量的前提和基础。目前的研究局限在基于同源性的丹参酮生物合成途径中关键酶基因的克隆、生物学信息和表达特征分析等方面。这些只能从序列上表明其与已知的其他物种的具催化功能的基因同源,并推测其活性,但若要确定其功能,需要进行蛋白表达纯化后的酶活研究。因此,对改造后的基因进行表达谱分析和酶活分析,寻找适合的载体以获得高产、稳定的再生丹参植株是今后研究者需要克服的难题和努力的方向。
| [1] | 中国药典 [S]. 一部. 2010. |
| [2] | 王 昕. 丹参酮药理研究及临床应用进展 [J]. 光明中医, 2011, 26(7): 1514-1517. |
| [3] | 葛宇清, 杨 波, 程汝滨, 等. 隐丹参酮对K562细胞凋亡的影响及其机制 [J]. 中草药, 2013, 44(22): 3188-3194. |
| [4] | Capell T, Christou P. Progress in plant metabolic engineering [J]. Curr Opinion Biotechnol, 2004, 15: 148-154. |
| [5] | Newman J D, Chappell J. Isoprenoid biosynthesis in plants: carbon partitioning within the cytoplasmic pathway [J]. Crit Rev Biochem Mol Biol, 1999, 34(2): 95-106. |
| [6] | Rohmer M, Knani M H, Simonin P, et al. Isoprenoid biosynthesis in bacteria: a novel pathway for the early steps leading to isopentenyl diphosphate [J]. Biochem J, 1993, 295: 517-524. |
| [7] | Laule O, Fürholz A, Chang H S, et al. Crosstalk between cytosolic and plastidial pathways of isoprenoid biosynthesis in Arabidopsis thaliana [J]. Proceed Nat Acad Sci USA, 2003, 100(11): 6866-6871. |
| [8] | 崔光红, 王学勇, 冯 华, 等. 丹参乙酰CoA酰基转移酶基因全长克隆和SNP分析 [J]. 药学学报, 2010, 45(6): 785-790. |
| [9] | 张 琳, 谭晓风, 胡 姣, 等. 油茶乙酰CoA酰基转移酶基因cDNA克隆及序列特征分析 [J]. 中南林业科技大学学报: 自然科学版, 2011, 31(8): 108-112. |
| [10] | 唐 其. 罗汉果转录组、表达谱的高通量测序及甜苷V生物合成关键酶的克隆 [D]. 北京: 北京协和医学院, 2010. |
| [11] | 王 敬. 丹参酮生物合成途径相关酶基因的克隆和特征分析 [D]. 上海: 上海师范大学, 2009. |
| [12] | 陈林波, 刘本英, 汪云刚, 等. 茶树HMGS基因的克隆与序列分析 [J]. 西北农业学报, 2013, 22(5): 72-76. |
| [13] | 王 伟. 喜树毛状根培养体系的建立及喜树hmgs基因的克隆分析 [D]. 上海: 上海师范大学, 2009. |
| [14] | 廖 攀. 丹参酮生物合成相关基因的克隆及其代谢调控 [D]. 上海: 上海师范大学, 2010. |
| [15] | 叶生晶. 杜仲MVA途径相关基因表达差异及全长cDNA序列特征 [D]. 长沙: 中南林业科技大学, 2013. |
| [16] | Liu Y, Xu Q X, Xi P Y, et al. Cloning and characterization of a cDNA coding 3-hydroxy-3-methylglutary CoA reductase involved in glycyrrhizic acid biosynthesis in Glycyrrhiza uralensis [J]. 药学学报, 2013, 48(5): 773-779. |
| [17] | 郑 鹏, 化文平, 王喆之. 秦艽HMGR基因家族的克隆及序列分析 [J]. 陕西师范大学学报: 自然科学版, 2012, 44(6): 67-72. |
| [18] | 陈大华, 叶和春. 马铃薯HMGR基因的克隆, 序列分析及其表达特征 [J]. 植物学报, 2000, 42(7): 724-727. |
| [19] | 张春荣, 杨 全, 陈虎彪, 等. 高良姜 1-脱氧-D-木酮糖-磷酸还原异构酶cDNA克隆与表达调控 [J]. 中国中药杂志, 2012, 37(21): 3208-3214. |
| [20] | 王学勇, 崔光红, 黄璐琦, 等. 丹参4-(5'-二磷酸胞苷)-2-C-甲基-D-赤藓醇激酶的cDNA全长克隆及其诱导表达分析 [J]. 药学学报, 2008, 43(12): 1251-1257. |
| [21] | 陈 洁, 雷美玉, 李辉亮, 等. 巴西橡胶树HbCMK基因的克隆及表达 [J]. 西北植物学报, 2009, 29(2): 215-220. |
| [22] | 高 伟, 程琪庆, 马晓惠, 等. 丹参2-C-甲基-D-赤藓糖醇2, 4-环焦磷酸合成酶 (SmMCS) 基因全长克隆及其生物信息学分析 [J]. 中国中药杂志, 2012, 37(22): 3365-3370. |
| [23] | 雷美玉, 李辉亮, 彭世清. 巴西橡胶树HbMCS1基因的克隆及表达分析 [J]. 农业生物技术学报, 2009, 17(3): 482-487. |
| [24] | 王 英, 贾伟章, 谭明俊, 等. 黄花蒿 MCS 基因的克隆及其序列分析与原核表达 [J]. 中草药, 2013, 44(16): 2288-2293. |
| [25] | 程琪庆, 何云飞, 李 耿, 等. 丹参4-羟基-3-甲基-2-丁烯基焦磷酸还原酶基因的全长克隆与诱导表达分析 [J]. 药学学报, 2013, 48(2): 236-242. |
| [26] | 杨颖舫, 杨春贤, 冯国庆, 等. 银杏 HDR 基因的克隆与功能分析 [J]. 安徽农业科学, 2010, 38(13): 6663-6665. |
| [27] | 李辉亮, 雷美玉, 彭世清. 巴西橡胶树4-羟基-3-甲基- 2-(E)-丁烯基-4-磷酸还原酶基因 (Hb-HDR) 的克隆及表达分析 [J]. 基因组学与应用生物学, 2009, 28(1): 15-21. |
| [28] | 钱 丹, 江雪飞, 乔 飞. 海南粗榧GGPPs基因克隆与诱导表达分析 [J]. 分子植物育种, 2013, 11(2): 204-210. |
| [29] | 唐 俊. 甘薯GGPPS基因的克隆分析及抗草甘膦半夏的获得 [D]. 重庆: 西南大学, 2008. |
| [30] | 高 伟, 崔光红, 孔建强, 等. 丹参柯巴基焦磷酸合酶基因的优化表达、纯化及抗体制备 [J]. 药学学报, 2008, 43(7): 766-772. |
| [31] | 姚 攀. 穿心莲二萜环合酶基因的克隆与原核表达 [D]. 广州: 广州中医药大学, 2011. |
| [32] | Lgual J C, Gonzalez-Bosch C, Dopazo J, et al. Phylogenetic analysis of the thiolase family implications for the evolutionary origin of peroxisomes [J]. J Mol Evol, 1992, 35(2): 147-155. |
| [33] | Luskey K L, Stevens B. Human 3-hydroxy-3- methylglutaryl coenzyme A reductase conserved domains responsible for catalytic activity and sterol-regulated degradation [J]. J Biol Chem, 1985, 260(18): 10271-10277. |
| [34] | Basson M E, Thorsness M, Finer-Moore J, et al. Structural and functional conservation between yeast and human 3-hydroxy-3-methyIglutaryl coenzyme A reductases, the rate-limiting enzyme of sterol biosynthesis [J]. J Mol Cell Biol, 1988, 8: 3797-3808. |
| [35] | Dai Z, Cui G, Zhou S F, et al. Cloning and characterization of a novel 3-hydroxy-3-methylglutaryl coenzyme A reductase gene from Salvia miltiorrhiza involved in diterpenoid tanshinone accumulation [J]. J Plant Physiol, 2011, 68(2): 148-157. |
| [36] | Dhe-Paganon S, Magrath J, Abeles R H. Mechanism of mevalonate pyrophosphate decarboxylase: evidence for carbocationic transition state [J]. Biochemistry, 1994, 33(45): 13355-13362. |
| [37] | McGarvey D J, Croteau R. Terpenoid metabolism [J]. Plant Cell, 1995, 7(7): 1015-1026. |
| [38] | Sprenger G A, Sehrken U, Wiegert T, et al. Identification of a thiamin-dependent synthase in Escherichia coli required for the formation of the 1-deoxy-D-xylulose 5-phosphate precursor to isoprenoids, thiamin, and Pyridoxo [J]. Proceed Nat Acad Sci USA, 1997, 94(24): 12857-12862. |
| [39] | Esteévez J M, Cantero A, Reindl A, et al. l-deoxy-D-xylulose-5-phosphate synthase, a limiting enzyme for plastidic isoprenoid biosynthesis in plants [J]. J Biol Chem, 2001, 276(25): 22901-22909. |
| [40] | Lange B M, Croteau R. Isoprenoid biosynthesis via a mevalonate-independent pathway in plants: cloning and heterologous expression of 1-deoxy-D-xylulose-5- phosphate reductoisomerase from peppermint [J]. Archiv Biochem Biophys, 1999, 365(l): 170-174. |
| [41] | Yan X M, Zhang L, Wang J. Molecular characterization and expression of 1-deoxy-D-xylulose-5-phosphate reductoisomerase (DXR) gene from Salvia miltiorrhiza [J]. Acta Physiol Plant, 2009, 31: 1015-1022. |
| [42] | Wu J Y, Shi M. Ultrahigh diterpenoid tanshinone production through repeated osmotic stress and elicitor stimulation in fed-batch culture of Salvia miltiorrhiza hairy roots [J]. Appl Microbiol Biotechnol, 2008, 78(3): 441-448. |
| [43] | Rohdieh F, Wungsintaweekul J, Luttgen H, et al. Biosynthesis of isoprenoids: 4-diphosphocytidyl-2-C- methyl-D-erythritolkinase from tomato [J]. Proceed Nat Acad Sci USA, 2000, 97: 8251-8256. |
| [44] | Steinbacher S, Kaiser J, Eisenreich W, et al. Structural basis of fosmidomycin action revealed by the complex with 2-C-methyl-erythritol-4-phosphate synthase (IspC). Implications for the catalytic mechanism and anti-malaria drug development [J]. J Biol Chem, 2003, 278: 18401-18407. |
| [45] | Herz S, Wungsintaweekul J, Schuhr C A, et al. Biosynthesis of terpenoids: YgbB protein converts 4-diphosphocytidyl-2C-methyl-D-eyrthritol 2-phosphate to 2C-methyl-D-erythritol 2, 4-cyclodiphosphate [J]. Proceed Nat Acad Sci USA, 2000, 97(6): 2486-2490. |
| [46] | Baker J, Franklin D B, parker J. Sequence and characterization of the gcpE gene of Escherichia coli [J]. FEMS Microbiol Lett, 1992, 94(1/2): 175-180. |
| [47] | Cunningham F X, Lafond T P, Gantt E. Evidence of a role for LytB in the nonmevalonate pathway of isoprenoid biosynthesis [J]. J Bacteriol, 2000, 182(20): 5841-5848. |
| [48] | 张 蕾, 戴住波, 崔光红, 等. 丹参牻牛儿基牻牛儿基焦磷酸合酶基因的克隆和分析 [J]. 中国中药杂志, 2009, 34(21): 2704-2708. |
| [49] | Cheng Q, He Y, Li G, et al. Effects of combined elicitors on tanshinone metabolic profiling and SmCPS expression in Salvia miltiorrhiza hairy root cultures [J]. Molecules, 2013, 18(7): 7473-7485. |
| [50] | 盛东峰, 陈 龙. 聚乙二醇-6000胁迫对丹参毛状根中丹参酮积累的影响 [J]. 中草药, 2013, 44(9): 1181-1185. |
 2015, Vol. 46
2015, Vol. 46


