2. 南加州大学 凯克医学中心 诺里斯综合癌症中心, 美国 洛杉矶 90089;
3. 福建医科大学附属泉州第一医院 泌尿外科, 福建 泉州 362000;
4. 天津医科大学 药学院 天津市临床药物关键技术重点实验室, 天津 300070
2. Norris Comprehensive Cancer Center, Keck School of Medicine, University of Southern California, Los Angeles 90089, USA;
3. Department of Urology, the First Hospital of Quanzhou Affiliated Fujian Medical University, Quanzhou 362000, China;
4. Tianjin Key Laboratory on Technologies Enabling Development of Clinical Therapeutics and Diagnostics (Theranostics), School of Pharmacy, Tianjin Medical University, Tianjin 300070, China
与遗传突变不同,表观遗传学改变是可逆的,通过两种DNA去甲基化药物使一些肿瘤抑制基因(TSG)再表达,如阿扎胞苷在2004年被FDA批准用于骨髓增生异常综合征(MDS)、急性骨髓性白血病(AML)和慢性骨髓单核细胞性白血病(CMML),以及地西他滨在2006年被FDA批准用于抗MDS和AML[1]。从治疗的角度看,DNA甲基化是一个很有潜力的目标。然而,两种临床批准的DNA去甲基化药物对于不同的DNA甲基转移酶(DNMT)不具有选择性,化学性质上不稳定,并且具有强烈的次级效应,如肾毒性、骨髓毒性[2]。阿扎胞苷和地西他滨属于核苷类似物,需要并入DNA中并用作共价结合DNMT,化学稳定性差,低特异性,毒性副作用显著[3-4]。相比之下,具有各种化学支架的非核苷化合物已经引起了很大的关注[5-6]。使用联合甲基化抑制测定的计算机筛选方法,设计出新的选择性非核苷DNA去甲基化药物DC_517,在癌细胞增殖中具有显着的抑制作用[7]。SGI-1027,一种喹诺酮衍生物,已经很好地研究了其生化和细胞低甲基化作用[8],其对一系列DNMT显示出一般的选择性。关于另一个实例RG108,基于化学数据库的虚拟筛选出来,在其效力和选择性方面也是有争议的。此外,几种天然产物,如染料木黄酮和表没食子儿茶素-3-O-没食子酸酯(EGCG),也显示有限的去甲基活性。本文对近几年DNA去甲基化药物的研究进展进行了综述。
1 核苷类似物 1.1 阿扎胞苷对于阿扎胞苷主要的临床试验目前主要关注联合用药。如一项II期临床试验,旨在确定阿扎胞苷单独用药和与组蛋白去乙酰化酶(HDAC)抑制剂mocetinostat联合用药治疗年龄超过60岁的AML或MDS患者。一些临床试验也用阿扎胞苷治疗实体瘤。直到最近,很少有观察到成功的,可能是因为需要调整用药时间和剂量的原因。此外,一项随机II期临床试验也是以阿扎胞苷和HDAC抑制剂恩替诺特联合治疗非小细胞肺癌[9]。前期对多数药物干预患者的研究取得了非常令人鼓舞的结果,包括一个完整的响应,而且还观察到与4个基因(APC、RASSF1A、CDH13和CDKN2A)的甲基化状态相关。另一个策略是探索应用去甲基化药物使肿瘤细胞对其他抗肿瘤药物敏感,用以评价其安全性。口服单药阿扎胞苷和联合卡铂或紫杉醇纳米粒(ABI-007)治疗复发或难治性实体瘤,以确定最大耐受剂量或最大剂量。
1.2 地西他滨许多试验中,阿扎胞苷的存在是为了确定地西他滨与HDAC抑制剂或其他抗癌药物,甚至免疫系统刺激因子联合用药的疗效,目的是找到在实体瘤的治疗策略。地西他滨结合基质细胞衍生因子抑制剂-1a(SDF-1a)普乐沙福对老年AML患者诱导和缓解治疗,通过激动白血病干细胞和改变地西他滨的药效学来提高治疗结果。地西他滨联合KRAS野生型肿瘤EGFR靶向生物制剂帕尼单抗用于晚期转移性结直肠癌的二线或三线治疗,主要目的在于评价其安全性和用药顺序的可行性。还观察参与大肠癌或EGFR信号通路的TSG再表达或其启动子甲基化的减少,以及评估无进展生存的总体反应。基于这样一个事实:DNA去甲基化药物能够重新编程细胞,已经尝试应用地西他滨重新编程骨髓增生异常细胞,使其像正常干细胞一样。DNA去甲基化药物与免疫系统刺激因子结合,主要考虑地西他滨可能通过重新编程活性,阻止细胞分裂,进而发挥抗肿瘤活性。地西他滨与干扰素α-2b治疗不能手术切除或转移性实体肿瘤的患者。一项I/II期试验提出了一种基于DNMT抑制剂重新编程性质的有趣理性方法(ClinicalTrials.gov,NCT01194908)。这里,三阴性乳腺癌转移患者,即不表达雌激素受体(ER)、孕激素受体(PR)和人表皮生长因子受体2(HER2),以及不对药物如曲妥珠单抗和他莫昔芬敏感,经过地西他滨和HDAC抑制剂帕比司他治疗,恢复ER表达,可继续用他莫昔芬进行治疗。这表明,ER在三阴性乳腺癌治疗中表观遗传沉默。联合用药可以删除的表观遗传沉默标记通过DNA去甲基化和组蛋白去乙酰化再次激活ER。这些被激活的ER细胞再次表现出对药物如他莫昔芬的敏感,试验通过激活的雌激素受体被跟踪和肿瘤生长对他莫昔芬的反应被评估两部分完成。
1.3 其他两个胞嘧啶类似物阿扎胞苷(5-氮杂胞苷)和地西他滨(5-氮-2’-脱氧胞苷)分别连着核糖或脱氧核糖,都是在5位的碳原子被氮原子所取代。基于这个原理,合成了其他几个类似物,并发现对DNMT有抑制活性。NPEOC-DAC是地西他滨衍生物,考虑保护4位氨基功能,引入2-(对硝基苯基)-乙氧羰基(NPEOC)基团被细胞内羧酸酯酶去除。NPEOC-DAC在体内转化为地西他滨,但没有关于发展为潜在药物进一步报道[10]。作为阿扎胞苷的前体药物,CP-4200是阿扎胞苷的反式十八碳烯酸酯,具有延缓药物释放的特点,但相关临床试验并没有开展[11]。另外,还有5, 6-二氢-5-氮杂胞苷和5-氟-2’-脱氧胞苷等。折布拉林是另一种核苷类似物,最初作为一种抗代谢药物被合成出来,但后来用于DNMT1抑制剂[12]。在裸鼠实验,折布拉林具有去甲基化和抑制移植瘤生长作用,在猴实验,折布拉林的口服生物利用度低下[13],因此该药物尚未进行临床试验。虽然折布拉林是目前在许多实验室广泛使用阿扎胞苷和地西他滨参考物,但是没有药物开发和临床试验的文献报道。RX-3117的临床研究I期试验于2012年已在欧洲开始,但没有任何具体指标显示与表观遗传作用机制相关。SGI-110目前进入临床试验研究II期,用于MDS和AML的治疗。
2 小分子抑制剂 2.1 吲哚衍生物吲哚类化合物可以作用于不同的受体,是具有多种生物活性的配体[14]。一个典型的例子是2-arylindoles可作为高效的G蛋白偶联受体配体[15]。虽然有许多关于含有吲哚骨架的药物在肿瘤治疗领域中的报道,但是通过基于1H-吲哚骨架的非共价抑制剂诱导DNA甲基化的抑制作用仍然是难以确定的[16-17]。最近,研究人员陆续发现以吲哚为骨架的DNA甲基化调节剂。体外酶实验中,通过低剂量放射性同位素来检测人DNMT1,以氚标记的AdoMet(3H-SAM)作为甲基供体。在27个吲哚衍生物中,有17个化合物的DNA甲基化抑制IC50值为50 μmol/L或更小,其中化合物1抑制活性最强,对于DNMT1抑制的IC50值为3.53 μmol/L。辅因子S-腺苷同型半胱氨酸(SAH)作为标准阳性对照,IC50值为0.25 μmol/L。吲哚衍生物表现出对多种癌细胞生长的抑制活性,包括乳腺癌、前列腺癌、结肠癌、肺癌、肝癌、胰腺癌、黑色素瘤、纤维肉瘤、血液、肾、骨肉瘤、脑、卵巢、宫颈癌、多发性骨髓瘤、表皮样癌细胞系。通过计算细胞存活率,观察到化合物2在大多数不同的癌细胞株中有潜在的抑制作用,可进一步结构优化。此外,化合物3对白血病CCRF-CEM细胞和Jurkat细胞表现出更强有力的抑制活性,EC50值分别为6.5、8.5 μmol/L。然而,其他几个衍生物显示出对细胞生长的抑制活性,但对不同肿瘤细胞株的选择性不明显,EC50值大于10 μmol/L[18]。各吲哚衍生物结构见图 1。
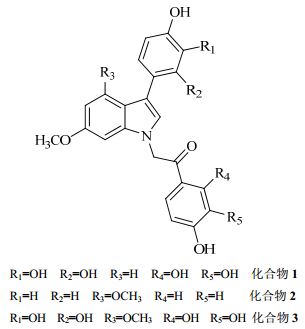
|
图 1 吲哚衍生物的结构 Fig. 1 Structures of indole derivatives |
2.2 杂芳基化合物
一个设计有效的DNA去甲基化药物的策略是模仿辅因子SAM或其催化产物SAH,所以寻找新的DNA去甲基化药物重点放在SAM或SAH的相似结构上[19]。杂环化合物含有容易协调的DNA结合区域,特别是胞嘧啶结合口袋的官能团,可通过一个共价或非共价地结合抑制DNMT活性。体外酶实验是在人全长DNMT1、人DNMT3A/DNMT3L的催化域进行。3H-SAM提供甲基化反应的供体,并且便于进行检测来评价杂环化合物对酶抑制活性。新合成的5个化合物中化合物4对DNMT3A/3L具有最强的效力,IC50值为3 μmol/L,并同时显示出良好的选择性。有报道,120个杂芳基化合物进行体外DNMT1、DNMT3B/3L活性实验中,对DNMT1抑制的IC50值在19.1~300 μmol/L。其中7个化合物对DNMT1抑制的IC50值小于100 μmol/L。24个化合物对DNMT3B/3L抑制的IC50值小于1 μmol/L。几乎所有的化合物都与化合物5一样含有丙酰胺部分。然而,目前尚不清楚这些化合物的构效关系。除了体外酶抑制活性,同时发明了药物的组合物,并制定了有效的药物组合物的各种方法,旨在为杂芳基化合物单独给药或与其他化疗药物联合提供可能,而且在被DNMT异常介导一系列癌症和过度增殖性疾病上,对杂芳基化合物的潜在效用需进行详细说明。各杂芳基化合物结构见图 2。
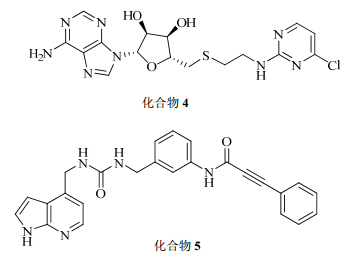
|
图 2 杂芳基化合物的结构 Fig. 2 Structures of heteroaryl compounds |
2.3 喹唑啉衍生物
2015年,研究人员合成了一系列作为DNA去甲基化药物的喹唑啉衍生物,通过体外DNMT1和DNMT3A抑制试验来评估这类化合物的DNA甲基化抑制活性。抑制DNMT3A分析方法是基于限制酶切的荧光检测,甲基化敏感的限制性内切酶HpyCH4IV被参与[20]。底物DNA 5’上标记生物素,互补链3’端连接6-FAM。人C-末端DNMT3A(氨基酸从623到908)被认为是催化结构域。相比之下,通过放射性基甲基化抑制试验检测DNMT1是传统方法。研究人员对36种化合物进行检测,其中化合物6、7、8显示高效的抑制活性,对DNMT3A抑制EC50值分别为2.4、1.1、1.1 μmol/L。然而,在人白血病细胞KG-1和淋巴瘤细胞Karpas 299的细胞增殖活性实验中,除化合物7,其余二者对DNMT1效力都很低。同时考察了喹唑啉衍生物对人黑色素瘤细胞WM-266-4、人胰腺癌细胞PANC1和人恶性胶质母细胞瘤细胞U87MG的选择性进行了评价。总之,化合物7、8表现出较高的白血病细胞的抑制活性,而在所有测试的细胞中化合物6都显示出潜在的抑制活性。各喹唑啉衍生物结构见图 3。
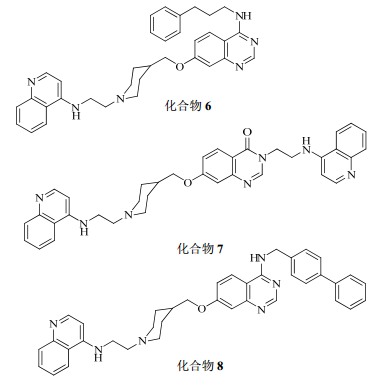
|
图 3 喹唑啉衍生物的结构 Fig. 3 Structures of quinazoline derivatives |
3 天然产物 3.1 表没食子儿茶素没食子酸酯
表没食子儿茶素-3-没食子酸酯(EGCG)通过逆转RECK基因的超甲基化状态抑制口腔癌细胞系的癌侵袭能力,从而增强RECK mRNA的表达水平。在这些细胞中,50 μmol/L EGCG处理中也观察到了MMP活性,特别是MMP-2和MMP-9水平的抑制。各种研究已经证明RECK甲基化与人类癌症中的转移和侵袭的增强相关。它可以用作治疗口腔癌的组合化学预防/化疗方法的治疗策略[21]。口腔癌的体外研究表明,多阶段肿瘤发生模型证明细胞增殖在EGCG的致癌作用的所有阶段被阻止,在发育不良的细胞中证明具有最佳功效。已经显示用EGCG处理导致细胞周期的G1期中细胞比例的增加,并且还诱导凋亡[22]。已发现绿茶提取物和EGCG通过S、G2/M期停滞抑制3种鳞状细胞系(CAL-27、SCC-25和KB)的生长。受绿茶提取物和EGCG影响的主要信号级联显示为EGFR和Notch途径,另外还影响细胞周期的相关网络[23]。
绿茶提取物有防止癌前病变的恶性转化的潜力。与安慰剂治疗的患者相比,更高剂量的绿茶提取物(750或1 000 mg/m2)的响应率更大。在日本,无口腔癌病史、40~79岁的20 550名男性和29 671名女性患者在平均10.3年的随访期间,仅观察到37例口腔癌患者。对于女性来说,每天绿茶消费量1~2、3~4或5杯是每天1杯量罹患口腔癌风险的0.51、0.60、0.31;对于男性,由于癌症病例数相对较低,关联度不高,无统计学意义[24]。在法国,观察到口腔癌与茶、咖啡摄取之间呈负相关,优势比(OR)分别为0.39、0.60,并且当两者由同一受试者消耗时具有协同效应。没有观察到男性和女性之间的风险差异。研究发现,EGCG使通过7, 12-二甲基苯并[a]蒽(DMBA)诱导的口腔癌仓鼠发育不良,口腔癌的风险降低。相对于4-硝基喹啉1-氧化物诱导的大鼠,绿茶多酚治疗的大鼠中观察到肿瘤数目、肿瘤体积和口腔鳞状细胞癌均显著减少[25]。
3.2 染料木黄酮染料木黄酮是大豆中的主要异黄酮组分,具有众多的药理活性,包括对各种酶如激酶、拓扑异构酶和最近的DNMT的抑制性质[11]。大豆中存在的主要异黄酮和相关的大豆异黄酮可抑制DNA甲基转移酶活性,逆转DNA超甲基化,并在口腔癌细胞系中重新激活甲基化沉默基因[26]。染料木黄酮也是表观遗传事件的有效修饰物,包括直接DNA甲基化和/或组蛋白乙酰化或通过类固醇受体依赖的过程。已显示染料木黄酮(2~20 μmol/L)通过逆转人食管癌细胞KYSE510细胞中的DNA超甲基化而重新激活RARβ、p16INK4a和MGMT基因,并且可以在这些浓度下抑制细胞增殖[27]。染料木黄酮(20~50 μmol/L)以剂量相关性方式抑制DNA甲基转移酶活性,底物与甲基供体表现相关性抑制。当与曲古抑菌素、萝卜硫素或地西他滨组合时,染料木黄酮可以增强这些甲基化沉默基因的再活化和细胞生长的抑制。在口腔鳞状细胞癌实验动物模型中,在对照组和染料木黄酮处理的组之间观察到肿瘤/荷瘤动物模型的平均数目、平均肿瘤体积或潜伏期在统计学上没有显著差异[28-29]。
3.3 姜黄素姜黄素存在于姜黄中的多酚类化合物,也被设计为基于DNMT1的同源性模型的虚拟筛选中的DNMT抑制剂。通过评估姜黄素对CpG甲基转移酶(M.SssI)的抑制性能,获得了确认,IC50为30 nmol/L[30]。另一研究小组得到姜黄素的药物组合物可增加其血浆浓度,还提供了在白血病MV4-11细胞中姜黄素诱导的DNMT1、DNMT3a和DNMT3b下调的蛋白质印迹分析,并且给出了TSG再活化的实验数据,但通过直接结合酶抑制剂的DNMT下调的机制还不清楚。当以100 mg/kg姜黄素处理小鼠而没有明显的毒性时,在异种移植的MV4-11模型中肿瘤生长降低了70%。由聚乙二醇和聚氧乙烯蓖麻油组合组成的用于改善姜黄素的生物利用度的药物组合物是关键点之一。考虑到归因于姜黄素的多种活性,需要进一步研究以证实其对表观遗传调节的潜在作用。在临床前实验中,姜黄素已经证明了具有抗癌活性,如在多种癌细胞系中的生长抑制和凋亡诱导。姜黄素的作用机制包括转录因子NF-κB抑制,HSP90抑制以及与直接抑制DNMT1的催化位点相关的表观遗传调节。姜黄素的临床开发受限于较差的药动学特征和低效力[31]。
已开发超过100种姜黄素类似物具有改善的溶解度和效力。EF31、UBS109是两种有希望的姜黄素类似物,其初步数据表明比以前的类似物如EF24具有更高的效力[32]。DNMT-1的转录由Sp1/NF-κB控制。NF-κB在两种胰腺细胞系中的过表达导致DNMT1的过表达。NF-κB在超过70%的胰腺癌中过表达,表明靶向NF-κB作为抑制DNMT1和甲基化方法的潜在作用。姜黄素通过抑制IKK、Akt活化来抑制NF-κB[33]。EF31、UBS109在胰腺癌细胞系中显示出比姜黄素更有效的NF-κB抑制活性。EF31、UBS109的作用是通过抑制上游IKK-α和-β介导。此外,DNMT1依赖于HSP90的活化和稳定性。姜黄素、EF31或UBS109通过降低HSP90与DNMT1的结合以及降低HSP90表达水平导致HSP90活性的抑制。总之,姜黄素、EF31和UBS109通过多种途径抑制DNMT-1活性和表达。
4 寡核苷酸一系列特异性小RNA分子及其衍生物参与抑制哺乳动物DNMT活性已经设计出来。RNA分子可以改变甲基化模式作为DNMT抑制剂,并反过来增加或恢复由DNMT异常活化下调的基因表达。DNMT1的晶体结构及其与DNA的相互作用已经得到很好的研究,特别是对于参与DNA结合的催化结构域[34]。理论上,长度为4~8个核苷酸的RNA分子可以足够长以占据DNMT的催化袋,并且作为竞争性抑制剂是有效的。虽然具有30个核苷酸或甚至更长的RNA分子也可以用作有效的抑制剂,但使用不超过30个核苷酸的RNA更方便。
在RNA结合测定中,asCEBPα-1HPE最有效地结合到DNMT1上,可能归因于缺乏任何茎-环结构。在竞争性抑制测定中,表达并纯化全长人DNMT1重组蛋白,并将半甲基化DNA以及3H-SAM作为底物。4个示例性RNA分子证明是在半甲基化DNA底物上的人DNMT1的竞争性抑制剂。其中,asCEBPα-1HPE被证明比asCEBPα-1更有效,Ki值分别为0.92、2.0 μmol/L。类似地,asCEBPα-2HPE比asCEBPα-2更有效,Ki值为0.14、0.43 μmol/L。此外,还评估了在RNA结构中,大小和序列方面与RNA分子不同的微小RNA抑制人DNMT1的能力。在14个测试的miRNA中,Ki值从miR-19b-3p的3.3 μmol/L变化到最有效的miR-155-5p的0.03 μmol/L。关于人DNMT和细菌甲基转移酶(包括M.Sssl、M.Hhal和M.Hpall)之间的选择性,RNA分子和微RNA显示对3种细菌甲基转移酶中任一种有很少抑制或没有抑制。此外,基于细胞的测定表明,当在人类结肠直肠癌细胞系HCT-116中施用时,miR-155-5p可引起显著的基因组低甲基化,类似于阿扎胞苷。MicroRNA与DNMT1的共免疫沉淀进一步证实miR-155-5p可以与HCT-116细胞中的DNMT1结合,从而抑制酶活性。研究了RNA分子的序列和二级或三级结构如何影响对人DNMT1的结合亲和力。因此,在3'末端附近含有鸟嘌呤碱基的RNA序列连同最小化二级结构或形成G-四联体(G-四联体)可显著改善抑制活性。此外,RNA分子需要与人基因的DNA序列不超过80%互补,并且它们对DNMT的靶基因的抑制活性不是特异性的。
5 小结DNA去甲基化药物作用机制主要有抑制DNA甲基转移酶,干扰甲基转移反应途径和影响三羧酸循环途径等[35]。非核苷小分子DNA去甲基化药物通常具有弱的效力和/或缺乏选择性,使得难以设计出良好的构效关系。天然的DNA去甲基化药物难以通过合成和化学改性进一步优化。此外,普鲁卡因、普鲁卡因胺和肼屈嗪也被报道具有DNA去甲基活性,但其作用仍然有争议。迄今为止,多数DNA去甲基化药物的疗效是否仅仅依赖于它们的脱甲基化活性还不清楚。从临床试验看来,剂量和方案的选择是重要的因素。DNA去甲基化药物的重新编程性质似乎对治疗效果起重要作用。同样,对于靶点的有效催化抑制剂的发现将有助于给出新的治疗方向。即使单独测定DNA甲基化水平可能不具有全面预测性,但对于患者肿瘤组织相关基因的甲基化状态的测定还是有助于给出个体化的精准治疗方案的。此外,甲基化状态调节可以诱导肿瘤细胞对其他抗癌药的敏感性,这对于DNA去甲基化药物与抗癌药联用是非常重要的。因此,存在迫切需要发现以高效力和高选择的靶向性新一代DNA去甲基化药物。
| [1] |
Issa J P, Kantarjian H M.
Targeting DNA methylation[J]. Clin Cancer Res, 2009, 15(12) : 3938–3946.
DOI:10.1158/1078-0432.CCR-08-2783 ( 0) 0)
|
| [2] |
Boumber Y, Issa J P.
Epigenetics in cancer:what's the future?[J]. Oncology (Williston Park), 2011, 25(3) : 220–226.
( 0) 0)
|
| [3] |
Stresemann C, Lyko F.
Modes of action of the DNA methyltransferase inhibitors azacytidine and decitabine[J]. Int J Cancer, 2008, 123(1) : 8–13.
DOI:10.1002/(ISSN)1097-0215 ( 0) 0)
|
| [4] |
Gros C, Fahy J, Halby L, et al.
DNA methylation inhibitors in cancer:recent and future approaches[J]. Biochimie, 2012, 94(11) : 2280–2296.
DOI:10.1016/j.biochi.2012.07.025 ( 0) 0)
|
| [5] |
Medina-Franco J L, Méndez-Lucio O, Dueñas-González A, et al.
Discovery and development of DNA methyltransferase inhibitors using in silico approaches[J]. Drug Discov Today, 2015, 20(5) : 569–577.
DOI:10.1016/j.drudis.2014.12.007 ( 0) 0)
|
| [6] |
Hu J, Chen S, Kong X, et al.
Interaction between DNA/histone methyltransferases and their inhibitors[J]. Curr Med Chem, 2015, 22(3) : 360–372.
( 0) 0)
|
| [7] |
Chen S, Wang Y, Zhou W, et al.
Identifying novel selective non-nucleoside DNA methyltransferase 1 inhibitors through docking-based virtual screening[J]. J Med Chem, 2014, 57(21) : 9028–9041.
DOI:10.1021/jm501134e ( 0) 0)
|
| [8] |
Datta J, Ghoshal K, Denny W A, et al.
A new class of quinoline-based DNA hypomethylating agents reactivates tumor suppressor genes by blocking DNA methyltransferase 1 activity and inducing its degradation[J]. Cancer Res, 2009, 69(10) : 4277–4285.
DOI:10.1158/0008-5472.CAN-08-3669 ( 0) 0)
|
| [9] |
Juergens R A, Wrangle J, Vendetti F P, et al.
Combination epigenetic therapy has efficacy in patients with refractory advanced non-small cell lung cancer[J]. Cancer Discov, 2011, 1(7) : 598–607.
DOI:10.1158/2159-8290.CD-11-0214 ( 0) 0)
|
| [10] |
Byun H M, Choi S H, Laird P W, et al.
2'-Deoxy-N4-[2-(4-nitrophenyl) ethoxycarbonyl]-5-azacytidine:a novel inhibitor of DNA methyltransferase that requires activation by human carboxylesterase 1[J]. Cancer Lett, 2008, 266(2) : 238–248.
DOI:10.1016/j.canlet.2008.02.069 ( 0) 0)
|
| [11] |
Fahy J, Jeltsch A, Arimondo P B.
DNA methyltransferase inhibitors in cancer:a chemical and therapeutic patent overview and selected clinical studies[J]. Expert Opin Ther Pat, 2012, 22(12) : 1427–1442.
DOI:10.1517/13543776.2012.729579 ( 0) 0)
|
| [12] |
Zhou L, Cheng X, Connolly B A, et al.
Zebularine:a novel DNA methylation inhibitor that forms a covalent complex with DNA methyltransferases[J]. J Mol Biol, 2002, 321(4) : 591–599.
DOI:10.1016/S0022-2836(02)00676-9 ( 0) 0)
|
| [13] |
Lyko F, Brown R.
DNA methyltransferase inhibitors and the development of epigenetic cancer therapies[J]. J Natl Cancer Inst, 2005, 97(20) : 1498–1506.
DOI:10.1093/jnci/dji311 ( 0) 0)
|
| [14] |
Horton D A, Bourne G T, Smythe M L.
The combinatorial synthesis of bicyclic privileged structures or privileged substructures[J]. Chem Rev, 2003, 103(3) : 893–930.
DOI:10.1021/cr020033s ( 0) 0)
|
| [15] |
Costantino L, Barlocco D.
Privileged structures as leads in medicinal chemistry[J]. Curr Med Chem, 2006, 13(1) : 65–85.
DOI:10.2174/092986706775197999 ( 0) 0)
|
| [16] |
Ahmad A, Sakr W A, Rahman K M.
Anticancer properties of indole compounds:mechanism of apoptosis induction and role in chemotherapy[J]. Curr Drug Targets, 2010, 11(6) : 652–666.
DOI:10.2174/138945010791170923 ( 0) 0)
|
| [17] |
Ishikawa H, Colby D A, Seto S, et al.
Total synthesis of vinblastine, vincristine, related natural products, and key structural analogues[J]. J Am Chem Soc, 2009, 131(13) : 4904–4916.
DOI:10.1021/ja809842b ( 0) 0)
|
| [18] |
Xu P, Hu G, Luo C, et al.
DNA methyltransferase inhibitors:an updated patent review (2012-2015)[J]. Expert Opin Ther Pat, 2016, 26(9) : 1017–1030.
DOI:10.1080/13543776.2016.1209488 ( 0) 0)
|
| [19] |
Isakovic L, Saavedra O M, Llewellyn D B, et al.
Constrained (l-)-S-adenosyl-l-homocysteine (SAH) analogues as DNA methyltransferase inhibitors[J]. Bioorg Med Chem Lett, 2009, 19(10) : 2742–2746.
DOI:10.1016/j.bmcl.2009.03.132 ( 0) 0)
|
| [20] |
Ceccaldi A, Rajavelu A, Champion C, et al.
C5-DNA methyltransferase inhibitors:from screening to effects on zebrafish embryo development[J]. Chembiochem, 2011, 12(9) : 1337–1345.
DOI:10.1002/cbic.v12.9 ( 0) 0)
|
| [21] |
Kato K, Long N K, Makita H, et al.
Effects of green tea polyphenol on methylation status of RECK gene and cancer cell invasion in oral squamous cell carcinoma cells[J]. Br J Cancer, 2008, 99(4) : 647–654.
DOI:10.1038/sj.bjc.6604521 ( 0) 0)
|
| [22] |
Masuda M, Suzui M, Weinstein I B.
Effects of epigallocatechin-3-gallate on growth, epidermal growth factor receptor signaling pathways, gene expression, and chemosensitivity in human head and neck squamous cell carcinoma cell lines[J]. Clin Cancer Res, 2001, 7(12) : 4220–4229.
( 0) 0)
|
| [23] |
Liu X, Zhang D Y, Zhang W, et al.
The effect of green tea extract and EGCG on the signaling network in squamous cell carcinoma[J]. Nutr Cancer, 2011, 63(3) : 466–475.
DOI:10.1080/01635581.2011.532901 ( 0) 0)
|
| [24] |
Ide R, Fujino Y, Hoshiyama Y, et al.
A prospective study of green tea consumption and oral cancer incidence in Japan[J]. Ann Epidemiol, 2007, 17(10) : 821–826.
DOI:10.1016/j.annepidem.2007.04.003 ( 0) 0)
|
| [25] |
Radoï L, Paget-Bailly S, Menvielle G, et al.
Tea and coffee consumption and risk of oral cavity cancer:results of a large population-based case-control study, the ICARE study[J]. Cancer Epidemiol, 2013, 37(3) : 284–289.
DOI:10.1016/j.canep.2013.02.001 ( 0) 0)
|
| [26] |
Jha M, Aggarwal R, Jha A K, et al.
Natural compounds:DNA methyltransferase inhibitors in oral squamous cell carcinoma[J]. Appl Biochem Biotechnol, 2015, 177(3) : 577–594.
DOI:10.1007/s12010-015-1768-y ( 0) 0)
|
| [27] |
Fang M Z, Chen D, Sun Y, et al.
Reversal of hypermethylation and reactivation of p16INK4a, RARbeta, and MGMT genes by genistein and other isoflavones from soy[J]. Clin Cancer Res, 2005, 11(19 Pt 1) : 7033–7041.
( 0) 0)
|
| [28] |
Yang Y, Zhou Z T, Ge J P.
Effect of genistein on DMBA-induced oral carcinogenesis in hamster[J]. Carcinogenesis, 2006, 27(3) : 578–583.
DOI:10.1093/carcin/bgi234 ( 0) 0)
|
| [29] |
Myoung H, Hong S P, Yun P Y, et al.
Anti-cancer effect of genistein in oral squamous cell carcinoma with respect to angiogenesis and in vitro invasion[J]. Cancer Sci, 2003, 94(2) : 215–220.
DOI:10.1111/cas.2003.94.issue-2 ( 0) 0)
|
| [30] |
Liu Z, Xie Z, Jones W, et al.
Curcumin is a potent DNA hypomethylation agent[J]. Bioorg Med Chem Lett, 2009, 19(3) : 706–709.
DOI:10.1016/j.bmcl.2008.12.041 ( 0) 0)
|
| [31] |
Fu S, Kurzrock R.
Development of curcumin as an epigenetic agent[J]. Cancer, 2010, 116(20) : 4670–4676.
DOI:10.1002/cncr.v116:20 ( 0) 0)
|
| [32] |
Nagaraju G P, Zhu S, Wen J, et al.
Novel synthetic curcumin analogues EF31 and UBS109 are potent DNA hypomethylating agents in pancreatic cancer[J]. Cancer Lett, 2013, 341(2) : 195–203.
DOI:10.1016/j.canlet.2013.08.002 ( 0) 0)
|
| [33] |
Kunnumakkara A B, Guha S, Krishnan S, et al.
Curcumin potentiates antitumor activity of gemcitabine in an orthotopic model of pancreatic cancer through suppression of proliferation, angiogenesis, and inhibition of nuclear factor-kappaB-regulated gene products[J]. Cancer Res, 2007, 67(8) : 3853–3861.
DOI:10.1158/0008-5472.CAN-06-4257 ( 0) 0)
|
| [34] |
Song J, Rechkoblit O, Bestor T H, et al.
Structure of DNMT1-DNA complex reveals a role for autoinhibition in maintenance DNA methylation[J]. Science, 2011, 331(6020) : 1036–1040.
DOI:10.1126/science.1195380 ( 0) 0)
|
| [35] |
许嵘, 张文州, 陈琳琳, 等.
DNA去甲基化药物作用机制的研究进展[J]. 现代药物与临床, 2017, 32(1) : 152–157.
( 0) 0)
|
 2017, Vol. 32
2017, Vol. 32


