2. 中山大学生命科学学院, 广东广州 510275
2. School of Life Sciences, Sun Yat-sen University, Guangzhou 510275, China
大戟科(Euphorbiaceae)是被子植物中的一个大科,约300属,8 000种以上;除北极及寒冷的高山带以外,遍布全球;中国有66属864种[1],主要分布在西南、华南;是热带和亚热带植物区系中的重要组成部分[2]。海南大戟科植物包含了粮食作物木薯,战略植物橡胶树,能源植物麻风树、油桐;药用植物巴豆和大戟等约46属139种(不含栽培种)[3]。大戟科植物多数种类有毒,许多植物因为其毒性或药用价值被人们所熟知,如巴豆Croton tiglium L.、大戟Euphorbia pekinensis Rupr.、泽漆Euphorbia helioscopia L.、甘遂Euphorbia kansui T. N. Liou ex S. B. Ho、续随子Euphorbia lathyris L.、蓖麻Ricinus communis L. 等[4]。因此对寻求准确快速的鉴定大戟科植物的方法显得尤为重要。一直以来,对大戟科的分类处理争议众多,实验研究很不充分。基于形态学的分类将大戟科分为4个亚科叶下珠亚科(Phyllanthoideae Ascherson)、铁苋菜亚科(Acalyphoideae Ascherson)、巴豆亚科(Crotonoideae Pax)、大戟亚科(Euphorbioideae)[5]。在传统形态分类基础上,有人通过花粉亚显微形态特征、核型以及保守基因序列分析[6, 7, 8]等方法对该科植物分类进行了探索性研究,取得了一定的进展。但是目前对于大戟科分类研究仍然缺乏一个能被普遍接受的分类系统。
2003年由加拿大动物学家Hebert等[9]首次提出的DNA条形码技术,给人们对生物分类的研究提供了新的方法。这一技术利用基因组中一段公认的标准短序列来进行物种鉴定的分子诊断;与传统分类学方法相比DNA条形码技术不依赖标本的形态特征也能对其实现正确鉴定。截止目前,研究者们已经提出了10多条植物候选DNA条形码序列[10, 11, 12, 13, 14, 15, 16],并在不同的类群中对各条形码的鉴定能力进行了考察。其中,ITS2被有些研究者提出可作为药用植物鉴定的标准条形码序列[17, 18, 19],但ITS2对植物分类鉴定的效率仍缺少在更大样本量中的验证。本研究选取采自海南地区的大戟科植物25个种56个样品评估ITS2序列作为DNA条形码对海南大戟科植物的鉴定能力。
1 材料与方法 1.1 材料以采自海南地区的大戟科植物25个种56个样品为实验材料。由海南医学院唐历波教授鉴定,数字影像信息及凭证标本保存于海南医学院理学院动物学与植物学教研室。实验材料来源详见表 1。
| 表 1 样本来源 Table 1 Sources of samples |
取变色硅胶(青岛海洋化工有限公司)干燥的叶片,利用DNA球磨仪(Retsch MM300,德国)研磨30 s(2 000 r/min)。采用植物基因组DNA提取试剂盒(天根生化科技有限公司,中国)提取总DNA。PCR扩增测序引物一致,正向为5’-GCGAT-ACTTGGTGTGAAT-3’,反向为5’-GACGCTTCTC-CAGACTACAAT-3’(引物的合成由上海生工生物工程股份有限公司完成)。PCR反应体系为25 μL,体系包括MgCl2 2 μL(25 mmol/L),dNTP 2 μL(2.5 mmol/L),PCR缓冲液2.5 μL,引物各1.0 μL(2.5 μmol/L)(生工生物工程股份有限公司,中国),聚合酶1.0 U(博彩生物科技有限公司,中国),总DNA 1 μL(约30 ng)。扩增程序:94 ℃,变性5 min;94 ℃变性30 s;56 ℃退火30 s;72 ℃延伸45 s(进行40个循环);72 ℃延伸10 min[7]。使用的PCR仪为promega M7501 PCR Master Mix 10 reactions TAQ。PCR扩增产物的测序工作由深圳华大基因生物医学工程有限公司完成。测序峰图经Codon Code Aligner拼接后,利用MEGA 6.0进行相关数据分析,并构建邻接(NJ)树。利用TAXON DNA软件分析序列种内、种间变异并作bar coding gap分析。从http://its2.bioapps.biozentrum.uniwuerzburg.de/获得供试样本的预测ITS2二级结构。具体方法参考文献方法[20]。
2 结果与分析 2.1 DNA模板及PCR扩增效率、测序成功率本实验56份样本中提取的DNA,获得较高质量DNA序列占42.4%。56份DNA样品进行扩增,扩增成功率为95.2%;扩增产物测序成功率为93.3%,即序列获得率88.8%。
2.2 序列长度和变异系数分析用MEGA 6.0软件对所得序列进行比对分析,比较不同序列变异位点数与信息位点数。结果表明,ITS2序列长度199~229 bp,全序列排序比对长度为300个位点,其中有252个变异位点,209个简约信息位点,分别占总序列长度的84%和69.67%。
2.3 种内、种间变异距离值及barcoding gap分析应用MEGA 6.0软件基于K2P距离模型计算供试样本种内、种间变异距离值,结果表明供试样本最大种内变异距离值为0.067 3;最小种间变异距离值为0.119 1。再通过TAXON DNA软件做barcoding gap图(图 1)。从图 1中可以看出ITS2序列种内、种间变异的分布情况,种内变异和种间变异分布呈两边开的趋势,存在明显的barcoding gap。此结果表明,ITS2序列对供试样本在种水平的鉴定能力较强。另外从图 1中可以发现种间变异距离值在0.15~0.35、0.90~1.28出现2个密集区间,进一步分析这2个区间发现:0.15~0.35这一区间主要是由巴豆亚科与大戟亚科、巴豆亚科与铁苋菜亚科的种间变异距离值组成,而0.90~1.28主要由叶下珠亚科与大戟科另外3个亚科之间的种间变异距离值组成。
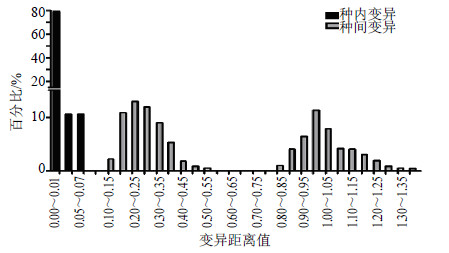 | 图 1 56个供试样本基于ITS2序列的barcoding gap Fig.1 Bar coding gap of ITS2 sequence for 56 samples |
用MEGA 6.0软件对ITS2基因进行序列比对和系统发育分析,结果见图 2,供试的25个种56个样本被分为2分支(Ⅰ和Ⅱ)。分支Ⅰ中包括17个种41个样本,分支Ⅱ包括8个种15个样本,从图 2中可以看出ITS2序列对供试的25个种56个样本在种的水平上能很好地分开,鉴定成功率达100%。
 | 图 2 基于ITS2序列构建的56个样本的NJ树Fig.2 NJ Dendrogram of 56 species based on ITS2 sequences |
根据ITS2数据库(http://its2.bioapps.biozentrum.uni-wuerzburg.de/)所预测的供试样本ITS2二级结构结果可以看出所有物种的二级结构均由1个中心环及4个螺旋区构成,每个螺旋上又有或多或少的大小不一的茎、环结构(图 3)。在比较样本中各种种内ITS2二级结构时发现,红背桂、白背叶、大飞扬草、蓖麻、一品红、绿玉树、春峰、红雀珊瑚、麻风树的种内ITS2二级结构在各螺旋区长度、茎环数目、大小、位置以及各螺旋之间的夹角等方面基本相同;红桑种内2样本ITS2二级结构差异主要表现在螺旋I区和螺旋III区,螺旋II区和螺旋IV区较保守;变叶木2样本ITS2二级结构在螺旋IV区较保守,在螺旋I区、螺旋II区和螺旋III区均存在差异;龙骨2样本ITS2二级结构在螺旋I区、螺旋II区和螺旋III区较为保守,在螺旋IV区有较小差异。
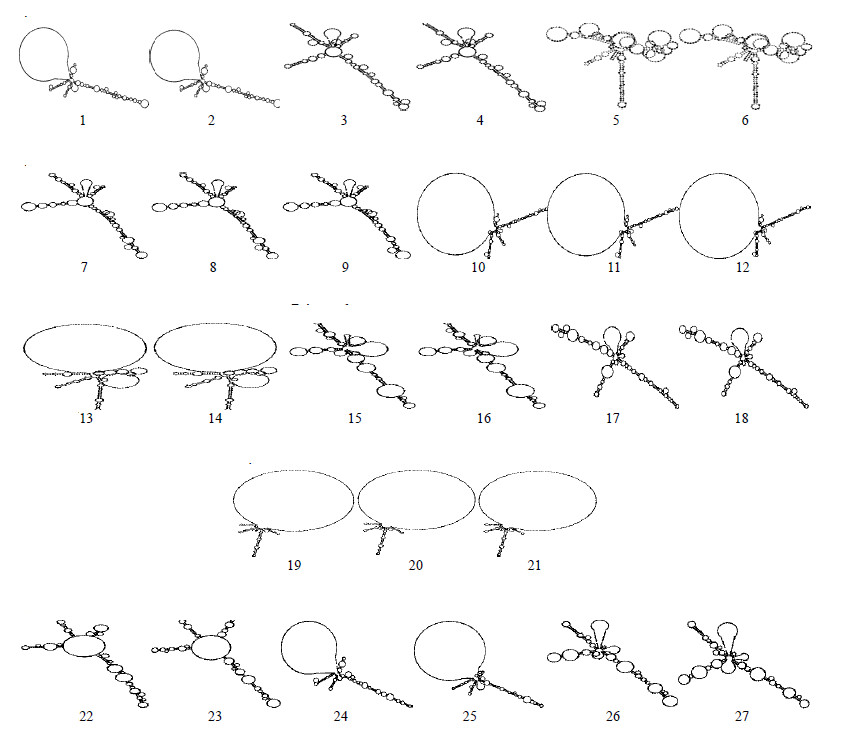 |
1、2-红背桂 3、4-白背叶 5、6-大飞扬草 7~9-蓖麻 10~12-一品红 13、14-绿玉树 15、16-春峰 17、18-红雀珊瑚 19~21-麻风树 22、23-红桑 24、25-变叶木 26、27-龙骨 1,2-Excoecaria cochinchinensis 3,4-Mallotus apelta 5,6-Euphorbia hirta 7—9-Ricinus communis 10—12-Euphorbia pulcherrima 13,14-Euphorbia tirucalli 15,16-Euphorbia lactea f.cristata 17,18-Pedilanthus tithymaloides 19—21-Jatropha carcas 22,23-Acalypha wikesiana 24,25-Codiaeum variegatum 26,27-Euphorbia trigona 图 3 ITS2序列预测二级结构图 Fig.3 Comparison on ITS2 secondary structure |
在比较样本种间ITS2二级结构时发现,种间ITS2二级结构在中心环大小及4个螺旋区结构上均存在明显差异。因此,通过ITS2二级结构,可以将供试样本中的各个种直观地鉴别出来。
3 讨论DNA条形码技术旨在建立一种准确、方便、快捷且对形态学分类技术要求不高的一种生物分类鉴定方法。这一技术方法的建立必须基于大量样本对DNA条形码鉴定能力的测定与评估。虽然近年来国内外学者在这方面做了不少的工作,但样本的数量还远远不够。本研究通过海南地区基于形态学上的大戟科植物25个种56个样本评估ITS2序列作为DNA条形码对海南大戟科植物的鉴定能力。
本研究的结果显示变异位点与简约信息位点在ITS2序列中均占较高的比例,说明ITS2序列中存在大量的物种分类进化信息,因此,ITS2序列适于对大戟科植物在种的水平上进行鉴定。在对种间的遗传距离进行分析时发现,较大种间变异距离值主要由叶下珠亚科与大戟科另外3个亚科之间产生,此结果揭示了叶下珠亚科与其他3个亚科之间在亲源关系上距离较远。
综合种内、种间的变异距离值、barcoding gap分析、NJ树构建及ITS2序列预测二级结构图分析等方法从多方面对ITS2条形码序列对海南大戟科植物鉴定能力进行了分析鉴定,结果均显示ITS2条形码序列对海南大戟科植物在种的水平上鉴定能力强,可作为DNA条形码序列对海南大戟科植物进行快速鉴定。另外,在DNA提取量少与完整性不好的情况下,ITS2序列也能获得并扩增,且不影响鉴定效果。因此在利用ITS2条形码序列鉴定植物时,对实验室的条件及操作者要求均不高,从而有利于该技术的广泛推广与应用。
| [1] | 刘明生. 黎药学概论[M]. 北京:人民卫生出版社, 2008. |
| [2] | 钟捷东. 黎族医药[M]. 海口:海南出版社, 2008. |
| [3] | 曾渝, 刘明生, 杨俊斌. 海南黎族医药亟待抢救发掘[J]. 中国药业, 2006, 15(11):18-19. |
| [4] | 吴征镒. 新华本草纲要第二册[M]. 上海:上海科技出版社, 1991. |
| [5] | 吴征镒. 中国植被[M]. 北京:科学出版社, 1980. |
| [6] | 杨士雄, 郑卓, 陈碧珊, 等. 大戟科现代植物花粉形态[J]. 植物学报, 2013, 48(5):550-560. |
| [7] | 薛恒钢, 周颂东, 何兴金, 等. 中国大戟属13种15个居群的核型报道[J]. 植物分类学报, 2007, 45(5):619-626. |
| [8] | 邹智, 杨礼富, 安锋, 等. 大戟科植物MVK基因家族的全基因组鉴定与分析[J]. 西南林业大学学报, 2013, 33(4):64-71. |
| [9] | Hebert P D, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes[J]. Proc R Soc Lond B Biol Sci, 2003, 270(1512):313-321. |
| [10] | Kress W J, Wurdack K J, Zimmer E A, et al. Use of DNA barcodes to identify flowering plants[J]. Proc Natl Acad Sci USA, 2005, 102(23):8369-8374. |
| [11] | Pennisi E. Wanted:a barcode for plants[J]. Science, 2007, 318(5848):190-191. |
| [12] | Sass C, Little D P, Stevenson D W, et al. DNA barcoding in the Cycadales:testing the potential of proposed barcoding markers for species identification of Cycads[J]. PLoS One, 2007, 2(11):e1154. |
| [13] | Lahaye R, vander Bank M, Bogarin D, et al. DNA Barcoding the floras of biodiversity hotspots[J]. Proc Natl Acad Sci USA, 2008, 105(8):2923-2928. |
| [14] | Newmaster S G, Fazekas A J, Steeves R A D, et al. Testing candidate Plant barcode regions in the Myristicaceae[J]. Mol Ecol Res, 2008, 8(3):480-490. |
| [15] | Newmaster S G, Fazekas A J, Ragupathy S. DNA barcoding in landplants:evaluation of rbcL in a multigene tiered approach[J]. Can J Bot, 2006, 84(3):335-341. |
| [16] | Chen S L, Yao H, Han J P, et al. Validation of the ITS2 Region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS One, 2010, 5(1):e8613. |
| [17] | Gao T, Yao H, Song J Y, et al. Identification of medicinal plants in the family Fabaceae using a potential DNA barcode ITS2[J]. J Ethnopharmacol, 2010, 130(1):116-121. |
| [18] | Pang X H, Song J Y, Zhu Y J, et al. Applying plant DNA barcodes for Rosaceae species identification[J]. Cladistics, 2010, 27(2):165-170. |
| [19] | 辛天怡, 姚辉, 罗焜, 等. 羌活药材ITS/ITS2条形码鉴定及其稳定性与准确性研究[J]. 药学学报, 2012, 47(8):1098-1105. |
| [20] | 李栎, 肖憬, 苏振宇, 等. ITS2条形码序列对茜草科黎药植物的鉴定[J]. 中草药, 2013, 44(13):1814-1818. |
 2016, Vol. 47
2016, Vol. 47


