白桦Betula platyphylla Suk.,桦木科(Betulaceae)桦木属Betula L.落叶乔木,是我国东北及内蒙古林区的主要树种之一。白桦三萜类物质具有抗菌、抗病毒、利胆和保肝等作用,白桦酯酸及其衍生物具有抗肿瘤和抗HIV等活性。此外,白桦植株中的齐墩果酸还具有抗炎、增强免疫功能、纠正体内异常蛋白代谢、防止肝硬化等功能,是治疗肝炎的有效药物[1],由于其高效低毒等特性近年来受到科学家和医学工作者越来越多的关注[2,3,4,5]。陆生生物,例如植物,进化出具有一定可塑性的新陈代谢来适应环境的变化以及完成其生长发育进程,而这些过程需要合成大量类异戊二烯产物[6,7]。其中高等植物通过异戊二烯基二磷酸(isopentenyl diphosphate,IPP)及其同分异构体烯丙基二磷酸合成出上千种类异戊二烯化合物和派生物[8]。植物通过2个相互独立的通路合成IPP和烯丙基二磷酸:甲羟戊酸(mevalonic acid,MVA)途径,定位于细胞质基质[9,10];和甲基赤磷酸盐途径,定位于质体[11,12]。MVA代谢途径是萜类化合物生物合成的重要途径[13]。通过该途径形成了植物中的倍半萜、三萜和甾醇等。在MVA代谢途径中,先由3分子乙酰辅酶A(acetyl-coenzyme A,CoA)缩合成3-羟基-3-甲基戊二酰辅酶A(3-hydroxy-3-methyl-glutaryl-CoA,HMG-CoA),该反应在Fe2+和质体醌(quinone)的辅助下由乙酰辅酶A酰基转移酶(acetyl-CoAacetyltransferase,AACT)和HMG-CoA合成酶(3-hydroxy-3-methy-lglutarylCoAsynthase,HMGS)共同催化完成[14];随后,HMG-CoA还原酶(3-hydroxy-3-methyl-glutaryl CoA reductase,HMGR)催化HMG-CoA不可逆地形成具有6个碳原子的中间体甲羟戊酸(MVA),NADPH为该反应的供氢体;在ATP和二价金属离子的参与下,甲羟戊酸激酶(mevalonatekinase,MK)和磷酸甲羟戊酸激酶(phosphomevalonatekinase,MPK)将MVA磷酸化,形成MVAP和MVAPP;最后,MVAPP在焦磷酸甲羟戊酸脱羧酶(pyrophosphomevalonatedecarboxylase,MDC)的作用下脱羧形成IPP[15]。目前已经明确的是HMGR是催化甲羟戊酸(MVA)通路的主要限速酶,对调控MVA途径至关重要[16]。
前期研究表明,白桦脂醇和齐墩果酸等三萜类物质可以在白桦愈伤组织中积累[17]。但是白桦产生萜类化合物的MVA代谢途径中白桦HMGR(BpHMGR)基因的表达模式尚不清楚。本实验重点研究MVA代谢途径中关键基因BpHMGR基因及其编码蛋白的特性,以及BpHMGR基因的节律表达模式,同时揭示其对外源信号分子NO的响应,为进一步研究BpHMGR基因的调控模式及其在MVA途径中的作用奠定基础。
1 材料与方法 1.1 材料和处理材料经笔者鉴定为长势良好的2年生白桦Betula platyphylla Suk.的幼苗。将白桦苗置于气候培养箱中培养,设置12 h光照和12h黑暗的光周期变化,连续培养3 d后取样。选取有代表性的叶片,液氮速冻,每隔3 h为一个时间点,连续24 h。
外源NO处理以白桦悬浮细胞为实验材料,按50 g/L接种量接种于100 mL B5液体培养基中,培养条件为120 r/min,25 ℃,光照强度2 000 lx,光周期16 h,湿度40%~50%,培养周期10 d。培养基为B5基本培养基,附加激素及质量浓度为0.2 mg/L 6-BA和0.6 mg/L TDZ,蔗糖20 g/L,酸水解酪蛋白1 g/L,pH 5.5~6.0,121 ℃高温高压灭菌20 min。
硝普钠(SodiumNitroprusside,SNP)购自美国Sigma公司,溶于灭菌蒸馏水中后经0.22 μm微孔滤膜滤过,在白桦悬浮细胞培养至第8天时添加至培养基中,终浓度1 mmol/L,添加后6、12、24、48、72 h,收获细胞,以同时期添加等体积灭菌水的材料为对照。每个处理重复3次。NO清除剂cPTIO[2-(4-carboxyphenyl)-4,4,5,5-tetramethylimi-dazoline-1-oxyl-3-oxidepotassium salt]为NO专一性猝灭剂,亚铁氰化钾(potassiumferrocyanide,KFeCN)为SNP类似物,但不提供NO,二者均购自美国Sigma公司,分别溶于灭菌水中并经0.22 μm微孔滤膜滤过后添加入液体培养基中,其中cPTIO终质量浓度为150 μmol/L,于添加SNP前30 min加入,KFeCN终浓度与最佳SNP终浓度相同。
1.2 BpHMGR基因编码区全长的克隆以白桦叶片作为材料,应用CTAB法提取总RNA[18,19]。应用反转录试剂盒将RNA反转录为cDNA。应用Clontech公司SMARTerTM RACE cDNAAmplification试剂盒获得BpHMGR基因全长序列(RACE引物见表 1)。应用凝胶回收试剂盒对特异性扩增产物回收纯化,纯化产物与pMD18-T载体连接,转化克隆进入JM109感受态细胞,送生工生物工程(上海)股份有限公司测序。
| 表 1 实时荧光定量PCR采用的引物序列 Table 1 Primer sequences for quantitative real-time PCR |
利用DNAMAN软件对获得的BpHMGR基因的核苷酸序列进行分析,并推测其蛋白质编码氨基酸序列[20]。使用Protparam在线分析软件分析BpHMGR基因编码蛋白的理化性质[21]。利用ProtScale在线分析软件中的Kyte and Doolittle算法分析BpHMGR蛋白的亲水/疏水性[22]。使用SignalP在线分析工具的神经网络算的预测并分析BpHMGR蛋白的信号肽[23]。使用TMPred在线工具预测和分析BpHMGR蛋白的跨膜结构[24]。使用COILS在线分析工具中预测和分析BpHMGR蛋白的卷曲螺旋[25]。使用wolfpsort在线工具对BpHMGR蛋白进行亚细胞定位分析[26]。应用GOR4软件分析蛋白的二级结构[27]。利用NCBI数据库Conserved Domain Search Service(CD Search)在线分析软件分析BpHMGR的保守结构域[28]。应用NCBI数据库Blast对BpHMGR氨基酸序列进行同源序列比对,搜索不同物种中的同源基因及蛋白。先采用ClustalX将氨基酸序列进行多序列比对分析,然后利用MEGA 5.0软件,算法为Neighbor-Joining,自检举1 000次,构建系统发育进化树[29,30,31]。
1.4 BpHMGR基因的实时荧光定量PCR表达特性分析以白桦微管蛋白基因Tu作为内参基因(Genbank:FG067376),引物分别为Tu-sense和Tu-anti,BpHMGR实时定量引物分别为BpHMGR-sense和BpHMGR-anti(表 1)。实时荧光定量PCR所用试剂盒为SYBR Green Realtime PCRMaster Mix-Plus-(codeNo.QPK-212T)购自于TOYOBO公司。PCR反应体系:模板cDNA 2 μL,Master Mix 10 μL,正反向引物(10 μmol/L)各1 μL,用ddH2O补足至总体积20 μL。反应程序:95 ℃预变性1 min,30 s、95 ℃,30 s、58 ℃,30 s、72 ℃,30 s、82 ℃,1 s用于读板,共45个循环。目标基因表达水平按照阈值2ΔΔCt方法计算,相对表达水平通过SNP处理与相应对照的比值来计算。以对照为1计算相对表达量。
1.5 齐墩果酸HPLC测定方法将SNP及cPTIO处理后的材料置于60 ℃烘箱中烘干24 h至恒定质量后称量得干质量进行齐墩果酸的提取与测定。在5 mL量瓶中,放入5.0 mg齐墩果酸对照品(生产批号110709-200505,购自中国食品药品检定研究院),用95%乙醇溶解并定容,用0.45 μm微孔有机滤膜过滤,即得到质量浓度为1 mg/mL的齐墩果酸对照品溶液,置于4 ℃冰箱冷藏保存。取上述对照品混合溶液,配成浓度为0.1、0.2、0.4、0.6、0.8、1.0 mg/mL的溶液进行进样分析。根据流动相乙腈-水(90∶10),体积流量1.0 mL/min,柱温为室温,进样量10 μL,检测波长210 nm的条件测定各质量浓度对照品峰面积,纵坐标为峰面积,横坐标为进样质量浓度(mg/mL),测定标准曲线,建立回归方程为Y=106 X+6903.1,r2=0.999 9,在测定范围内,峰面积与质量浓度成线性相关。精密秤取样品粉末0.5 g,以及少许适量沸石倒入100 mL圆底烧瓶中,加入盐酸-乙醇溶液(2∶8)25 mL,90 ℃水浴锅中加热回流3 h,放冷,滤过,量取滤液15 mL,再加入15 mL蒸馏水,置于80 ℃水浴上蒸去乙醇,所剩溶液用乙醚萃取3次,每次20 mL,低温40 ℃蒸干获得的乙醚提取液,用1 mL甲醇溶解所得残渣,用0.45 μm微孔有机滤膜滤过,测定样品中齐墩果酸的量。
2 结果及分析 2.1 BpHMGR基因的氨基酸序列理化性质分析HMGR是MVA代谢途径中的第一个重要限速酶,是MVA途径中的重要调节点。利用CTAB法提取白桦总RNA,以白桦cDNA为模板,经过RACE-PCR方法,获得了一条1 764 bp全长序列,编码蛋白含有587个氨基酸。序列包含完整开放阅读框,命名为BpHMGR,并已上传至NCBI,登录号为KJ452334。对BpHMGR的氨基酸序列的理化性质进行分析,BpHMGR蛋白的等电点(pI):6.76;不稳定系数:40.85,为不稳定蛋白(不稳定系数小于40时,预测蛋白质稳定,反之则不稳定)。总平均疏水性为0.011,说明该蛋白为疏水性蛋白。蛋白质亲疏水性氨基酸的组成是蛋白质折叠的主要驱动力,通过亲水性预测可以反映蛋白质的折叠情况。对BpHMGR蛋白进行亲水/疏水性分析(>0.5的区域为疏水区,<−0.5的区域为亲水区,介于0.5~−0.5主要为两性区域),结果表明BpHMGR蛋白共有36个疏水区和28个亲水区。
2.2 信号肽、亚细胞定位及跨膜结构的预测和分析信号肽是N端的一段氨基酸序列,指导分泌性蛋白到内质网膜上合成,在蛋白质合成结束之前被切除,信号肽位于蛋白质的N端,一般由16~26个氨基酸残基组成,其中包括疏水核心区、信号肽的C端和N端。对BpHMGR蛋白进行预测。结果表明,BpHMGR蛋白可能不存在信号肽。分析BpHMGR蛋白的亚细胞定位位置,BpHMGR蛋白在细胞膜和内质网的得分较高,都为6,因此,BpHMGR蛋白可能在细胞膜和内质网上起作用。
跨膜结构是蛋白质与膜内在蛋白的静电相互作用和氢键键合相互作用与膜结合的一段氨基酸片段,一般由20个左右的疏水性氨基酸残基组成,主要形成α-螺旋。对BpHMGR蛋白进行跨膜结构分析。结果表明,BpHMGR蛋白有12个跨膜结构域。其中,由内到外的跨膜结构域有6个,分别位于58~77位、98~116位、237~256位、443~472位、511~529位和543~566位,分数分别为2 032、2 241、608、616、291和1 438;由外到内的跨膜结构域有4个,分别位于58~77位、98~115位、238~259位、443~467位、509~532位和543~565位,分数分别为2 223、2 169、1 003、770、16和1 546(分数>500为显著跨膜结构)。经N-terminus outside模型分析表明,其中由内到外的98~116位、443~472位,和由外到内的58~77位、238~259位和543~565位具有强烈的跨膜螺旋结构。据此分析,BpHMGR蛋白既有由内到外的跨膜能力,又有由外到内的跨膜能力(图 1)。
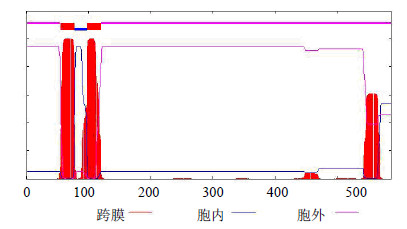 | 图 1 BpHMGR蛋白跨膜结构域预测和分析 Fig.1 Transmembrane domain prediction and analysis of BpHMGR protein |
对BpHMGR蛋白的二级结构进行分析。BpHMGR蛋白都是由α螺旋(Alpha helix)、延伸链(Extended strand)、无规卷曲(Random coil)所组成,并且分布于整个蛋白。其中,α螺旋占30.49%,延伸链占18.74%,无规卷曲占50.77%(图 2)。
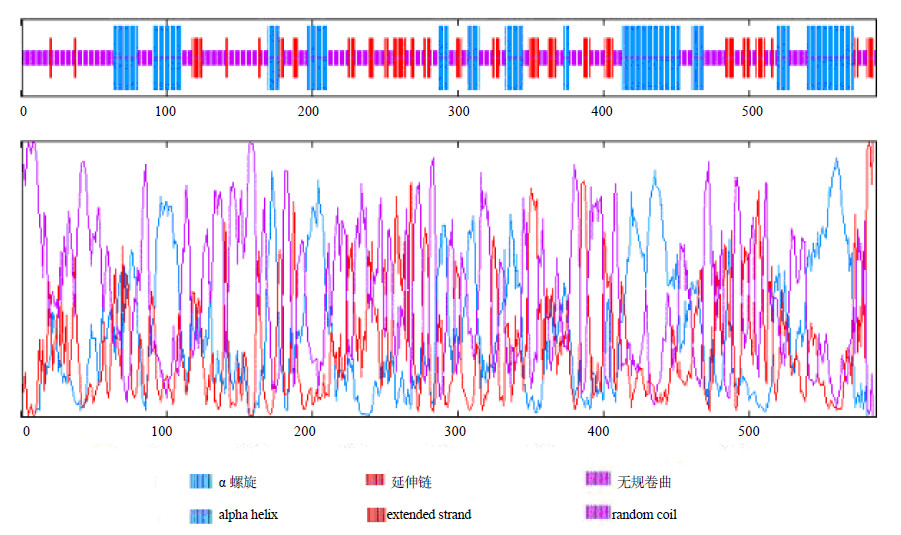 | 图 2 BpHMGR蛋白二级结构预测Fig.2 Secondary structure prediction of BpHMGR protein |
对BpHMGR蛋白进行三级结构同源建模,用Swiss-PdbViewer软件对建模结果进行处理。BpHMGR蛋白同源建模结果如图 3所示。同时,利用软件的α-碳与酰胺平面交角图功能,可以找到1个残基与1个特定平面的构象角(二面角),通过分析二面角,可以判断一个模型的质量。蛋白的二面角分析结果表明,BpHMGR蛋白结构不稳定。
 | 图 3 BpHMGR蛋白三级结构预测Fig.3 Tertiary structure prediction of BpHMGRprotein |
根据催化残基、辅酶结合位点、底物结合袋、抑制剂结合位点、四聚体接口对BpHMGR蛋白的保守结构域进行预测。发现其含有植物HMGR基因具有的保守序列:NADPH结合位点“DAMGMNM”和“GTVGGGT”,只显示其重要功能的结合基序。同时包括2个底物HMG-CoA的结合位点“ELPIGYVQLP”和“TTEGCLVA”,其中,TTEGCLVA中的谷氨酸(Glu)在HMGR催化中起着重要作用[32]。初步证明BpHMGR具有酶促活性,判断该编码蛋白的结构域应当都属于HMG-CoAreductase超家族(图 4)。
 | 图 4 BpHMGR保守结构域预测Fig.4 Prediction of conserved domain of BpHMGR |
对BpHMGR氨基酸序列进行同源序列比对并构建系统进化树。Blastp比对结果如表 2所示,BpHMGR蛋白与荔枝、蓖麻、橡胶树、毛果杨、大豆、苜蓿的蛋白一致性较高,说明同源性较高。
| 表 2 BpHMGR基因序列与其他物种同源比对 Table 2 Homology comparisonon BpHMGR gene sequence with other species |
将BpHMGR氨基酸序列与之构建系统进化树(图 5)。分析系统进化树结果表明,HMGR蛋白主要聚为3类,其中,BpHMGR与大豆、苜蓿亲缘关系较近,聚为一类;橡胶树、蓖麻、毛果杨、拟南芥、黄瓜和荔枝聚为一类;杜仲、烟草和番茄聚为一类。
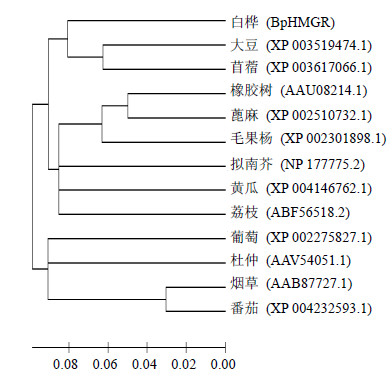 | 图 5 BpHMGR蛋白的系统进化树的构建 Fig.5 Phylogenetic tree of BpHMGR protein |
利用实时荧光定量PCR技术,应用2−ΔΔCt法对BpHMGR基因进行相对定量表达。横坐标表示不同的时间点,纵坐标表示各类关键基因的相对表达量,以零点时的数据为基准点。如图 6表示BpHMGR在24 h的相对表达量情况,0︰00表达量最低,之后开始上升,当6︰00给予光照处理时相对表达量开始降低,12︰00时达到最低点,之后开始上升,18︰00时给予黑暗处理后相对表达趋于缓和,21︰00达到最大值,为12︰00时的近二十倍。
 | 图 6 BpHMGR不同时间的节律表达特征Fig.6 Expression characteristics of BpHMGR at different time points |
白桦三萜合成关键酶HMGR是MVA途径的限速酶。外源NO诱导过程中BpHMGR基因相对对照的表达情况见图 7(注:图中显示的数值为处理数值与对照数值的差值,结果为正说明表达水平上调,为负说明表达水平下调)。SNP处理后BpHMGR基因表达水平表现为先上升后下降的趋势,仅72 h低于对照,48 h表达量最高。KFeCN和cPTIO加SNP处理组12 h虽然导致BpHMGR基因上调表达,但是表达水平均显著低于SNP处理(图 8)。综上可知,外源NO诱导了三萜合成途径中关键酶基因BpHMGR的上调表达。
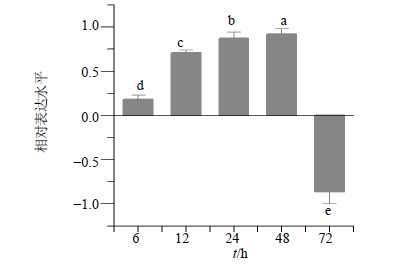 |
不同字母表示0.05显著水平,下同 Different letters indicate differences in the level of0.05, same as below 图 7 外源NO对BpHMGR基因表达的影响Fig.7 Effects of exogenous NO on expression of BpHMGR gene |
 | 图 8 SNP、KFeCN、cPTIO+SNP处理后BpHMGR的表达Fig.8 Expression of BpHMGR after treatment of SNP, KFeCN, and cPTIO + SNP |
以终浓度为1 mmol/L SNP对白桦悬浮细胞进行处理,KFeCN处理12 h细胞内齐墩果酸量并没有升高,表明SNP释放NO后的残余成分对齐墩果酸量没有促进作用(图 9)。cPTIO预处理可清除SNP释放的NO,SNP+cPTIO处理12 h,细胞内齐墩果酸量与对照持平(图 9)。SNP处理12 h齐墩果酸量为1.308 9 mg/g,是对照组5.69倍,这与SNP对白桦中BpHMGR基因的促进表达结果一致,初步说明外源NO影响了白桦细胞内三萜代谢途径的关键酶基因HMGR,进而调控了三萜产物齐墩果酸的合成。
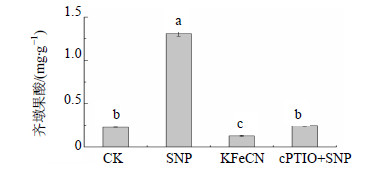 | 图 9 不同处理对白桦细胞齐墩果酸量的影响Fig.9 Changes of oleanolic acid content with treatment time |
HMGR催化类异戊二烯生物合成中MVA途径通路的第一步。在植物中,这条通路为大量的类异戊二烯产物提供了前体,这些类异戊二烯产物参与了多种生物功能,包括参与膜的合成的甾醇类化合物的生成,参与植物防御反应的倍半萜类植保素和类固醇配糖生物碱的合成,调控生长和发育的油菜素内酯和细胞分裂素的合成,蛋白法尼基和香叶基的异戊二烯化,多萜醇的蛋白糖基化,以及泛醌的呼吸作用等[33,34]。已知的所有HMGR蛋白及其同源蛋白都锚定在内质网上,并具有相同的膜拓扑结构[35]。其N端区域以及保守催化结构域定位于细胞质,而具有一个短的可伸展的氨基酸连接着细胞腔的2个跨膜区。这个与植物内细胞溶质中MVA的生物合过程完全相一致[35,36]。
植物的HMGR在MVA通路中起到了关键的调控作用,不仅仅是对于植物的正常生长发育,同时也是植物适应环境条件所必须的。与此一致的,植物HMGR受到大量内源信号以及外援刺激的调控,例如,植物激素、钙、钙调蛋白、光、类异戊二烯生物合成的抑制、化学刺激、伤口、诱导子处理以及病原体攻击[12,33]。研究表明,HMGR活力的调控应该决定在转录水平,然而有人提出,HMGR活力如果是转录后水平的调控,调控的效果会更快更好[34]。近期的研究表明,拟南芥中蛋白磷酸激酶2A(PP2A)通过5个β蛋白家族成员在包括转录后等多水平调控HMGR的活力。但是这种调控方式并不常见[7]。
本研究中,获得了BpHMGR基因全长1 764 bp,推测其编码蛋白含有587个氨基酸,为疏水性不稳定蛋白,BpHMGR基因具有跨膜结构域。不存在信号肽。BpHMGR蛋白可能在细胞膜和内质网上起作用,属于HMG-CoA reductase超家族。Green等[37]的结果表明8︰00到中午期间,花卉中HMGR基因表达减少。其他的MVA基因如HMGS、PMK、IDPI在20︰00时的表达量最高。这与本实验结果类似(图 6),当6︰00给予光照处理后BpHMGR相对表达量开始降低,12︰00时达到最低点,18︰00时给予黑暗处理后,BpHMGR基因的表达量开始增加,在21︰00达到最大值。这是因为光负调控MVA途径,MVA途径中的限速酶HMGR在光下迅速下调。同时HMGR酶活性在光下迅速降低[38]。当黑暗处理后BpHMGR基因开始积累,在21︰00时达到最大值。本研究结果表明BpHMGR的表达呈现出昼夜周期性节律变化,在21时达到峰值。
Wu等[39]实时实时荧光定量PCR分析表明,NO可以显着上调HMGR的表达。同时,SNP处理6 h后,HMGR的表达量为对照的2.5倍;处理12 h后为对照的3.4倍。表现为随着处理时间的延长,HMGE的表达量逐渐升高。这与本实验结果类似(图 7),在用SNP处理后BpHMGR的表达量逐渐上升,在48 h达到最大值。该结果表明,外源NO能够促进BpHMGR上调表达。本实验研究了生物节律及外源NO诱导对白桦BpHMGR表达模式和三萜量的影响,为进一步研究BpHMGR基因的调控模式,以及其在MVA途径中的作用奠定了基础。
| [1] | 田丽婷. 齐墩果酸药理作用研究进展[J]. 中国中药杂志, 2002, 27(12):884-886. |
| [2] | Hess S C, Brum R L, Honda N K, et al. Antibacterial activity and phytochemical analysis of Vochysia Divergens (Vochysiaceas)[J]. J Ethnopharmacol, 1995, 47(1):97-100. |
| [3] | Fujioka T, Kashiwada Y, Kilkuskie R E, et al. Anti-AID Sagents11 betulinic acid and platonic acids as anti HIV priciples from Syzigium claviflorum and the anti-HIV activity of structurally related triterpenoids[J]. J Nat Prod, 1994, 57(2):243-247. |
| [4] | Pisha E, Chai H, Lee I S, et al. Discovery of betulinic acid as a selective inhibiter of human melanoma that function by induction of apoptosis[J]. Nat Med, 1995, 1(10):1046-1051. |
| [5] | 刘志芹, 张庆云, 宋丽杨, 等. 白桦皮和白桦叶体外抗真菌作用研究[J]. 天津药学, 2004, 16(3):3-4. |
| [6] | Doblas V G, Amorim-Silva V, Posé D, et al. The SUD1 gene encodes a putative E3 ubiquitin ligase and is a positive regulator of 3-hydroxy-3-methylglutaryl coenzyme a reductase activity in Arabidopsis[J]. Plant Cell, 2013, 25(2):728-743. |
| [7] | Leivar P, Antolín-Llovera M, Ferrero S, et al. Multilevel control of Arabidopsis 3-hydroxy-3-methylglutaryl coenzyme A reductase by protein phosphatase 2A[J]. Plant Cell Online, 2011, 23(4):1494-1511. |
| [8] | Bouvier F, Rahier A, Camara B. Biogenesis, molecular regulation and function of plant isoprenoids[J]. Prog Lipid Res, 2005, 44(6):357-429. |
| [9] | McGarvey D J, Croteau R. Terpenoid metabolism[J]. Plant Cell, 1995, 7(7):1015-1019. |
| [10] | Newman J D, Chappell J. Isoprenoid biosynthesis in plants:carbon partitioning within the cytoplasmic pathway[J]. Critl Rev Biochem Mol Biol, 1999, 34(2):95-106. |
| [11] | Eisenreich W, Rohdich F, Bacher A. Deoxyxylulose phosphate pathway to terpenoids[J]. Trends Plant Sci, 2001, 6(2):78-84. |
| [12] | Rodríguez-Concepción M, Boronat A. Elucidation of the methylerythritol phosphate pathway for isoprenoid biosynthesis in bacteria and plastids. A metabolic milestone achieved through genomics[J]. Plant Physiol, 2002, 130(3):1079-1089. |
| [13] | Hey S J, Powers S J, Beale M H, et al. Enhanced seed phytosterol accumulation through expression of a modified HMG-CoA reductase[J]. Plant Niotechnol J, 2006, 4(2):219-229. |
| [14] | Bach T J, Boronat A, Caelles C, et al. Aspects related to mevalonate biosynthesis in plants[J]. Lipids, 1991, 26(8):637-648. |
| [15] | McGarvey D J, Croteau R. Terpenoid metabolism[J]. Plant Cell, 1995, 7(7):1015. |
| [16] | Hemmerlin A, Harwood J L, Bach T J. A raison d'être for two distinct pathways in the early steps of plant isoprenoid biosynthesis?[J]. Prog Lipid Res, 2012, 51(2):95-148. |
| [17] | 尹静, 詹亚光, 李新宇, 等. 不同树龄白桦的不同器官及其组培苗诱导的愈伤组织中白桦酯醇和齐墩果酸的分布和量变化[J]. 植物生理学通讯, 2009(6):610-614. |
| [18] | 曾凡锁, 南楠, 詹亚光. 富含多糖和次生代谢产物的白桦成熟叶中总RNA的提取[J]. 植物生理学通讯, 2007, 43(5):913-916. |
| [19] | 梁楠松, 周姗, 李蕾蕾, 等. 小黑杨bHLH转录因子基因和启动子的克隆与分析[J]. 经济林研究, 2013(4):58-66. |
| [20] | Huang Y, Zhang L. Rapid and sensitive dot-matrix methods for genome analysis[J]. Bioinformatics, 2004, 20(4):460-466. |
| [21] | Burge C B, Karlin S. Finding the genes in genomic DNA[J]. Curr Opin Struct Biol, 1998, 8(3):346-354. |
| [22] | Walker J M. The Proteomics Protocols Handbook[M]. Totowa:Humana Press, 2005. |
| [23] | Petersen T N, Brunak S, von Heijne G, et al. Signal P 4. 0:discriminating signal peptides from transmembrane regions[J]. Nat Methods, 2011, 8(10):785-786. |
| [24] | Hofmann K, Stoffel W. TMbase-A database of membrane spanning proteins segments[J]. Biol Chem Hoppe-Seyler, 1993, 374:166-169. |
| [25] | Lupas A, Van Dyke M, Stock J. Predicting coiled coils from protein sequences[J]. Science, 1991, 5009(252):1162-1164. |
| [26] | Horton P, Park K J, Obayashi T, et al. WoLF PSORT:protein localization predictor[J]. Nucl Acids Res, 2007, 35(Suppl 2):W585-W587. |
| [27] | Sen T Z, Jernigan R L, Garnier J, et al. GOR V server for protein secondary structure prediction[J]. Bioinformatics, 2005, 21(11):2787-2788. |
| [28] | Marchler-Bauer A, Lu S, Anderson J B, et al. CDD:a Conserved Domain Database for the functional annotation of proteins[J]. Nucl Acids Res, 2011, 39(Suppl 1):D225-D229. |
| [29] | 李军, 张莉娜, 温珍昌. 生物软件选择与使用指南[M]. 北京:化学工业出版社, 2008. |
| [30] | Tamura K, Dudley J, Nei M, et al. MEGA4:molecular evolutionary genetics analysis (MEGA) software version 4. 0[J]. Mol Biol Evolut, 2007, 24(8):1596-1599. |
| [31] | 梁楠松, 曾凡锁, 李博, 等. 硝普钠(SNP)和茉莉酸甲酯(MeJA)诱导的白桦Ty1-copia类逆转座子的克隆及分析[J]. 东北林业大学学报, 2013(10):69-74. |
| [32] | 蒋春, 张华玲, 彭江. 尾巨桉HMGR基因的克隆及表达分析[J]. 广西植物, 2012, 32(1):113-117. |
| [33] | Stermer B A, Bianchini G M, Korth K L. Regulation of HMG-CoA reductase activity in plants[J]. J Lipid Res, 1994, 35(7):1133-1140. |
| [34] | Chappell J. Biochemistry and molecular biology of the isoprenoid biosynthetic pathway in plants[J]. Annl Rev Plant Biol, 1995, 46(1):521-547. |
| [35] | Campos N, Boronat A. Targeting and topology in the membrane of plant 3-hydroxy-3-methylglutaryl coenzyme A reductase[J]. Plant Cell Online, 1995, 7(12):2163-2174. |
| [36] | Harpaz-Saad S, Azoulay T, Arazi T, et al. Chlorophyllase is a rate-limiting enzyme in chlorophyll catabolism and is posttranslationally regulated[J]. Plant Cell, 2007, 19(3):1007-1022. |
| [37] | Green S A, Chen X, Nieuwenhuizen N J, et al. Identification, functional characterization, and regulation of the enzyme responsible for floral (E)-nerolidol biosynthesis in kiwifruit (Actinidia chinensis)[J]. J Expl Bot, 2011, 63(5):1951-1967. |
| [38] | Vranová E, Coman D, Gruissem W. Structure and dynamics of the isoprenoid pathway network[J]. Mol Plant, 2012, 5(2):318-333. |
| [39] | Wu S J, Qi J L, Zhang W J, et al. Nitric oxide regulates shikonin formation in suspension-cultured Onosma paniculatum cells[J]. Plant Cell Physiol, 2009, 50(1):118-128. |
 2016, Vol. 47
2016, Vol. 47


