玄参科(Scrophulariaceae)地黄属Rehmannia Libosch. ex Fisch. et Mey. 植物在我国有6种,全部产于我国[1]。其中,中药材地黄来源于该属植物地黄Rehmannia glutinosa (Gaert.) Libosch. ex Fisch,全国各地均有分布,在河南焦作地区广为栽培,是著名的“四大怀药”之一[2]。《中国药典》2010年版中地黄以块根入药,药材分为鲜地黄、生地黄和熟地黄,具有清热生津、凉血、止血;补血滋阴、益精填髓等功效。用于热病伤阴、舌绛烦渴、温毒发斑、吐血、衄血、咽喉肿痛、骨蒸劳热、肝肾阴虚,腰膝酸软等症的治疗[3]。植物化学和药理学的研究表明地黄属植物活性成分为环烯醚萜苷类化合物,主要包括梓醇和毛蕊花糖苷等[3, 4],具有降血糖、抗炎、保肝、利尿和缓泻、调节免疫功能的作用[5],临床应用十分广泛。作为河南栽培的主要药材品种,野生和栽培的中药材地黄种内遗传变异显著,形态上有较大差异,与同属近缘种类很难区分,从而影响鉴定的准确性,造成传统鉴定困难[6]。此外,除正品地黄外,同属的一些植物(如湖北地黄等)在地方也作为中药材地黄入药[7],常有混淆现象,直接影响临床用药的安全。而目前,对该属的鉴定研究主要集中在形态性状和RAPD(random amplified phlymorphic DNA)分析等[8]。DNA条形码鉴定仅局限于对ITS2单条序列研究[9],缺乏对其他几个常用DNA条形码候选序列 [intemal transcribed spacer(ITS)、psbA-trnH、rbcL和matK] 在地黄属鉴定作用的评价和分析,且没有在整个属的水平取样,进行系统的鉴别分析。而在地黄属内,中药材地黄和茄叶地黄均为四倍体(2n=56),而其他种类均为二倍体(2n=28)[6]。目前,对于四倍体地黄是否来源于该属其他二倍体种类的异源或同源加倍后形成,尚不明确,且栽培地黄品种起源也不清楚。
近年来,随着分子生物学的快速发展,DNA条形码技术(DNA barcoding)为中药材物种鉴定及中药材物种的系统进化研究提供了方便快捷的方法[10]。DNA条形码技术是利用标准的、有足够变异的、易扩增且相对较短的DNA片段对物种进行快速、准确的自动鉴定,并通过构建中药材物种及其近缘种类的亲缘关系,探讨中药材物种的系统演化及栽培品种的起源[11]。目前,DNA条形码鉴定研究广泛应用于多个科属的药用植物及中药材鉴定[12, 13, 14, 15]。因此,本研究通过实验并结合GenBank数据库数据,分析ITS、psbA-trnH、rbcL和matK 4条常用条形码序列对地黄属物种的鉴别能力,以筛选能鉴别地黄属药用植物的优选序列及为地黄属植物的分子鉴定提供依据,并通过上述序列构建中药材地黄及其近缘种类的亲缘关系,探讨中药材地黄的栽培起源。
1 材料与方法 1.1 材料实验材料的收集包括地黄属5个种共13个样品,其中地黄栽培品种3个样品,地黄野生材料6个样品,地黄属其他种4个样品,实验材料由中国科学院植物研究所系统与进化植物学国家重点实验室王印政研究员鉴定,凭证标本保存于中国科学院植物研究所标本馆(PE)和河南农业大学。实验材料来源于植物新鲜叶片,经硅胶快速干燥后保存于−80 ℃冰箱,实验材料详细信息及GenBank登录号见表 1。
|
|
表 1 植物样品来源 Table 1 Origins of plantsamples |
采用北京天根生化植物DNA提取试剂盒(Tiangen Biotech Co.,China)提取样品DNA,ITS序列扩增使用Wendel等[16]设计的引物ITS1(5’-AGAAGTCGTA-ACAAGGTTTCCGTA-3’)和ITS4(5’-TCCTCCG-CTTATTGATATGC-3’);psbA-trnH、rbcL和matK序列扩增使用DNA条形码通用引物[17],引物由上海生工生物技术服务有限公司合成。PCR反应条件及扩增程序参考Xia等[18]和陈士林等[17]的研究。PCR产物纯化后,用ABI 3730X测序仪(Applied Biosystems Co.,美国)进行双向测序,测序结果在GenBank注册序列号(表 2)。
|
|
表 2 GenBank 序列来源序列号 Table 2 Accession numbers of GenBank database |
测序所得的峰图采用CodonCode Aligner V3.0软件(CodonCode Co.,美国)对序列峰图进行校对拼接,去除引物区和低质量的序列,同时,本研究从GenBank数据库中搜集并下载地黄属ITS、rbcL和matK 序列49条(表 2),整合处理后结合样品序列分析。用PAUP* version 4.0b10(Florida State University,美国)计算K2P(Kimura-2-Parameter)距离,基于K2P模型进行遗传距离分析,对不同序列种间、种内变异大小进行比较。统计不同序列种间、种内遗传距离的分布频度。利用软件PAUP* version 4.0b10构建邻接(Neighbor-Joining,NJ)系统发育树,利用Bootstrap(BS,1 000次重复)检验各分支的支持率。利用相似性搜索(BLAST1)、最近距离(Nearest Distance)、构建NJ系统发育树等方法评价各序列对地黄属的鉴定作用,并构建中药材地黄及其近缘种类的系统发育关系。
2 结果与分析 2.1 不同DNA条形码的序列信息、种内种间差异分析本研究中地黄属叶绿体基因matK序列有6个种,中药材地黄取样个体为10个,3个栽培品种,7个野生种(包括来源于GenBank 1个样品),共计15条序列,matK序列的扩增和测序成功率均为100%。序列对比发现,matK序列在地黄属内非常保守,序列同源性为99.9%~100%。所有序列长度为820 bp,6个种15条序列,仅出现2个位点的差异,分别为湖北地黄在572个位点发生T到G的颠换,和高地黄在660个位点发生G到T的颠换,其余种类茄叶地黄、裂叶地黄、天 目地黄与地黄的野生和栽培种类的matK序列同源性为100%。同样,叶绿体基因rbcL在地黄属内也极为保守,本实验取样的6个物种,中药材地黄取样个体为10个,包括3个栽培品种,7个野生种(包括来源于GenBank 1个样品),共计15条序列,rbcL序列的扩增和测序成功率均为100%。所有序列的同源性均为100%。这表明,进化速率较慢的这2个国际通用叶绿体基因的DNA条形码序列不适合地黄属内的种间鉴定,因此,在后面分析直接舍弃这2条序列。
地黄属ITS序列有6个种,取样的中药材地黄取样个体为9个,3个栽培品种,6个野生种,GenBank下载地黄物种40条序列,共计49条序列,高地黄1个样品,其余4个种均为2个样品。中药材地黄及其近缘种类的6个物种的ITS序列长度变异范围为613~615 bp。其中ITS1序列长度变异范围为223~224 bp,5.8 S区域长度为164 bp,6个物种序列完全一致。ITS2序列长度变异范围为224~227 bp。在ITS序列的矩阵中,本研究取样的地黄实验样品的3个栽培品种,6个野生种的ITS序列比对发现,仅河南林州采集的样品在162位点处出现一个由C到A的颠换,结合GenBank的序列,发现地黄属内各种间ITS序列具有丰富的变异。地黄属叶绿体基因psbA-trnH序列有5个种,中药材地黄9个样品包括3个栽培品种,6个野生种,其余4个种均为1个样品。在psbA-trnH序列的矩阵中,裂叶地黄和天目地黄的psbA-trnH序列最长为516 bp,湖北地黄最短为438 bp。湖北地黄对比其他种序列,在79个位点有1个T到G的转换,165位点处有1个G到T的转换,在200和459位点各有1个碱基的缺失,在223~299位点处有77个碱基的大范围缺失。茄叶地黄对比其他种序列,在第200位点处有1个碱基的缺失,在232~236位点处、286~290位点处,各有5个碱基的缺失。中药材地黄种内,psbA-trnH序列共有4个单倍型,河南温县栽培品种(金九、北京3号、85-5)3个样品与来源于河南温县、郑州和南阳野生地黄的psbA-trnH序列同源性为100%,序列长度为502 bp,共享1个单倍型。其余3个单倍型来源于北京、洛阳和林州的野生地黄样品。
种内种间差异是分析DNA条形码序列的重要指标。理想的条形码序列具有较大的种间变异、较小的种内变异,以便准确鉴别同属不同种的物种。本研究从平均种间变异、种间最小变异、平均种内变异、种内最大变异分析ITS和psbA-trnH序列的种内种间差异(表 3)。结果表明,ITS和psbA-trnH序列的平均种间变异(0.017 96和0.005 15)均大于平均种内变异(0.006 12和0.001 24),且2条序列的种间最小变异(0.004 93和0.001 71)均大于种内最大变异(0.003 64和0.000 81)。因此,ITS和psbA-trnH序列是鉴别中药材地黄及同属近缘种比较理想的条形码序列。
|
|
表 3 不同DNA条形码序列的种内和种间的变异分析 Table 3 Analysis on intra- and inter-specific variation of different DNA barcodes |
应用BLAST1对地黄属种类进行鉴定分析,结果表明ITS和psbA-trnH序列可以准确鉴别中药材地黄与其同属近缘种类。应用PAUP* version 4.0b10软件构建中药材地黄与其同属近缘种类的ITS和psbA-trnH序列的NJ系统发育树(图 1、2),外类群选择与地黄属近缘的崖白菜属。在ITS序列构建的分子系统树上(图 1),中药材地黄的49个样品与茄叶地黄的2个样品聚为一单系分支,支持率96%,其中,茄叶地黄的2个样品聚为一单系分支,支持率为63%,这表明地黄与茄叶地黄亲缘关系较近。高地黄与裂叶地黄聚为一单系分支,支持率为100%,湖北地黄和天目地黄的2个个体均各自聚为一单系分支,支持率为100%和69%。ITS序列构建的NJ树可以将中药材地黄与除茄叶地黄外的其他地黄属种类很好区分开。

|
图 1 基于NJ 法 (ITS序列) 构建系统发育树 Fig. 1 Phylogenetic tree constructed based on NJ methods (ITS data) |
|
图 2 基于NJ法 (psbA-trnH数据) 构建系统发育树 Fig. 2 Phylogenetic tree constructed based on NJ methods (psbA-trnH data) |
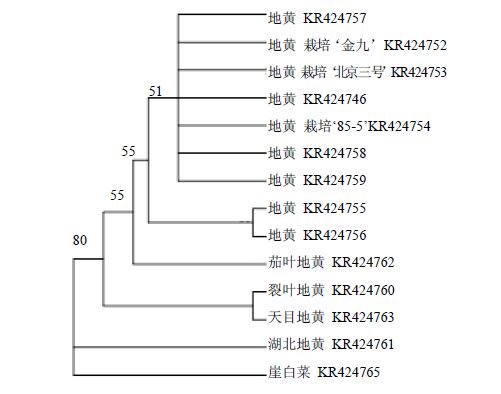
在psbA-trnH序列构建的分子系统树上(图 2),中药材地黄的9个样本单独聚为一单系,支持率为55%,可以很好地与地黄属其他种类区分开,其中来源于洛阳和林州2个野生居群聚为一单系分支,支持率为66%,其余3个栽培品种与来源于河南温县、河南郑州、南阳和北京野生地黄居群聚在一支,支持率为51%。与ITS系统树一致的是茄叶地黄与地黄聚在一起,支持率为55%,支持二者亲缘关系较近。与ITS系统树不同的是在ITS系统树上,地黄与茄叶地黄二者构成的单系分支,与裂叶地黄和高地黄构成的单系分支聚在一起,支持率为55%,但在psbA-trnH系统树上,地黄、茄叶地黄二者构成的单系分支,与裂叶地黄和天目地黄构成的单系分支聚在一起,支持率为80%。依据ITS和psbA-trnH序列构建的分子系统树可将中药材地黄与其同属近缘种类明显地区分开。
在ITS和psbA-trnH序列联合构建的分子系统树上(图 3),中药材地黄的9个样本单独聚为一单系,支持率为58%,其中来源于河南洛阳和林州2个野生居群聚为一单系分支,支持率为52%,其余3个栽培品种与来源于河南温县、郑州、南阳和北京野生地黄居群聚在一支,支持率为69%。与ITS和psbA-trnH系统树一致的是,茄叶地黄与地黄聚在一起,支持率为99%,支持二者亲缘关系较近。ITS和psbA-trnH序列联合构建的分子系统树支持地黄,茄叶地黄二者构成的单系分支,与裂叶地黄构成的单系分支聚在一起,支持率为68%,随后与天目地黄聚在一起,支持率为82%。

|
图 3 基于NJ法 (ITS和psbA-trnH联合数据) 构建系统发育树 Fig. 3 Phylogenetic tree constructed based on NJ method (combined ITS and psbA-trnH data) |
本研究通过对国际通用的DNA条形码候选序列对比分析发现,虽然叶绿体基因matK和rbcL在地黄属扩增和测序成功率均能达到100%,但这2个基因序列在该属内极度保守,进化速率较慢,种间差异极小或没有差异,不适合作为DNA条形码序列对该属属下种类进行分子鉴定研究。本研究对地黄属进行全面取样,研究结果发现核基因片段ITS(包括ITS2)和叶绿体基因psbA-trnH在该属内不仅扩增和测序成功率均能达到100%,且这2条序列在地黄属内存在丰富的变异,支持陈士林等[19]认为核基因ITS2的片段可以作为《中国药典》2015年版中药材地黄种内部分居群间的分子鉴定的DNA条形码序列。基于ITS和叶绿体基因psbA-trnH序列对地黄属种间和种内的变异分析表明,ITS和psbA-trnH序列的平均种间变异均大于平均种内变异,且2条序列的种间最小变异均大于种内最大变异。NJ分子系统树分析显示,核基因片段ITS和叶绿体基因psbA-trnH联合可以准确区分中药材地黄及其近缘种。因此,ITS和psbA-trnH序列可以作为地黄属内中药材地黄及其与同属近缘种进行鉴定的条形码序列。
3.2 中药材地黄物种形成及其栽培起源基于DNA条形码技术的分子鉴定不仅能准确鉴定中药材正品与伪品,还能准确构建药用植物与其近缘种类的系统发育关系,是探讨药用植物物种及其栽培品种起源的基础[11]。本研究对地黄属内种间的系统关系的研究结果表明,茄叶地黄与地黄的亲缘关系最近,与Xia等[18]和Albach等[20]的结果完全一致。与中药材地黄和茄叶地黄的四倍体不同,地黄属内其他4个种染色体2n=28,均为二倍体[6, 21]。Albach等[20]认为地黄种内ITS基因存在高度变异,加上形态特征的多样性,推断中药材地黄为异源四倍体[22]。本研究核基因ITS分子系统树显示,中药材地黄与茄叶地黄构成的分支,与裂叶地黄和高地黄构成的分支聚在一起,支持率为55%,表明地黄、茄叶地黄与裂叶地黄、高地黄亲缘关系较近。李宏庆等[23]和夏至等[6, 7]研究认为裂叶地黄与高地黄为同一种,高地黄为裂叶地黄的异名,因此,ITS分子系统树支持地黄、茄叶地黄与裂叶地黄亲缘关系最近。叶绿体基因psbA-trnH的分子系统树结果显示,中药材地黄与茄叶地黄构成的分支,与裂叶地黄和天目地黄构成的分支聚在一起,支持率为55%。相对于核基因的双亲遗传,叶绿体基因在被子植物中是母系遗传[24],因此,推测四倍体地黄的物种形成一定与二倍体裂叶地黄存在密切的联系,天目地黄可能是四倍体地黄物种形成的另一个亲本来源。
中药材地黄的栽培具有悠久的历史,新老产区均存在栽培品种混杂且来源并不清楚[2]。本研究核基因ITS和基因psbA-trnH的取样既包括地黄的野生类群,同时也包括部分栽培品种。在核基因ITS和叶绿体基因psbA-trnH联合的分子系统树上,发现河南温县取样3个栽培品种与河南温县、郑州、南阳和北京取样的野生群体聚在一起,支持率为69%,而同样这一分支在叶绿体基因psbA-trnH的分子系统树上也得到支持。对叶绿体基因psbA-trnH的单倍型分析结果表明,地黄野生和栽培品种有4个单倍型,河南温县取样3个栽培品种与河南温县、郑州和南阳取样的野生群体共享1个单倍型,而其余3个单倍型分别为河南洛阳、林州和北京的3个野生群体。这表明对比野生地黄,栽培地黄的遗传多样性已明显降低。遗传多样性降低的程度取决于人类栽培驯化过程中参与栽培起源的野生群体的大小,以及栽培驯化时间的长短,中药的栽培起源不像农作物那样规模庞大,时间长,人们只是零星地种植从本区域野外收集的药用植物,所以只有有限的野生群体参与了中药的栽培起源[25]。因此推测,取样的栽培地黄品种可能起源于分布于河南温县区域的野生群体。本研究对地黄属的DNA条形码鉴定及中药材地黄栽培起源研究进行初步探讨,但鉴于中药材地黄的栽培品种众多,且中药材地黄又是一个广布种,因此进一步在该属扩大取样范围(特别是扩大野生地黄不同区域分布的群体及栽培品种的数量),选取其他多基因片段开展分子谱系地理学研究,方能全面探讨中药材地黄的物种形成及其栽培起源,这将为地黄属的药用植物的资源开发与利用、品种选育、引种栽培提供可靠的理论依据。
志谢:中国科学院植物研究所系统与进化植物学国家重点实验室提供本研究分子实验平台。
| [1] | 中国科学院中国植物志编辑委员会. 中国植物志[第67(2)卷] [M]. 北京:科学出版社, 1979. |
| [2] | 温学森, 杨世林, 魏建和, 等. 地黄栽培历史及其品种考证[J]. 中草药, 2002, 33(10):946-949. |
| [3] | 中国药典[S]. 一部. 2010. |
| [4] | 王太霞, 李景原, 胡正海. 地黄的形态结构与化学成分研究进展[J]. 中草药, 2004, 35(5):585-587. |
| [5] | 邢洁, 徐为人, 刘鹏, 等. 栀子和地黄环烯醚萜类成分抗炎作用的虚拟评价[J]. 中草药, 2009, 40(6):930-935. |
| [6] | 夏至. 地黄属和崖白菜属的分子系统学研究-兼论地黄属和崖白菜属的科级系统位置[D]. 北京:中国科学院植物研究所, 2009. |
| [7] | 夏至, 李家美. 地黄属及其近缘属的药用植物资源调查研究[J]. 商丘师范学院学报, 2009, 25(12):96-98. |
| [8] | 陈京荔, 黄璐琦, 邵爱娟, 等. 地黄不同品种的RAPD分析[J]. 中国中药杂志, 2002, 27(7):505-508. |
| [9] | 侯典云, 辛天怡, 杨培, 等. 应用ITS2条形码鉴定中药材地黄[J]. 世界科学技术-中医药现代化, 2013, 15(3):441-445. |
| [10] | Hebert P D N, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes[J]. Proc Biol Sci, 2003, 270:313-321. |
| [11] | 韩建萍, 宋经元, 姚辉, 等. 中药材DNA条形码鉴定的基因序列比较[J]. 中国中药杂志, 2012, 37(8):1056-1061. |
| [12] | 夏至, 张红瑞, 李贺敏, 等. 碎米桠及其近缘种的分子鉴定和亲缘关系研究[J]. 中草药, 2013, 44(20):2904-2909. |
| [13] | 夏至, 李贺敏, 张红瑞, 等. 紫苏及其变种的分子鉴定和亲缘关系研究[J]. 中草药, 2013, 44(8):1027-1032. |
| [14] | 夏至, 高致明, 李贺敏, 等. 鬼针草及其近缘种的分子鉴定和亲缘关系研究[J]. 中草药, 2014, 45(6):828-834. |
| [15] | 夏至, 冯翠元, 高致明, 等. 黄芩及其同属近缘种的DNA条形码鉴定研究[J]. 中草药, 2014, 45(1):107-112. |
| [16] | Wendel J F, Schnabel A S, Seelanan T. Bidirectional inter locus concerted evolution following allopolyploid speciation in cotton (Gossypium)[J]. Proc Natl Acad Sci USA, 1995, 92:280-284. |
| [17] | Chen S L, Yao H, Han J P, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS One, 2010, 5:e8613. |
| [18] | Xia Z, Wang Y Z, Smith J F. Familial placement and relationships of R. and Triaenophora (Scrophulariaceae sensu lato) inferred from five different gene regions[J]. Am J Bot, 2009, 96(2):519-530. |
| [19] | 陈士林. 中国药典中药材DNA条形码标准序列[M]. 北京:科学出版社, 2015. |
| [20] | Albach D, Yan K, Jensen S, et al. Phylogenetic placement of Triaenophora (formerly Scrophulariaceae) with some implications for the phylogeny of Lamiales[J]. Taxon, 2009, 58:749-756. |
| [21] | 温学森, 李先恩, 赵华英, 等. 地黄常见种质的染色体观察[J]. 中草药, 2005, 36(1):124-125. |
| [22] | 刘占林, 李建芳. 地黄属分子系统学分析[J]. 西北植物学报, 2014, 34(1):77-82. |
| [23] | 李宏庆, 刘国丽. 地黄属的一个新异名[J]. 广西植物, 2012, 32(1):23-26. |
| [24] | Soltis D E, Kuzof R K. Discordante between nuclear andchloroplast phylogenies in the Heuchera Group (Saxifragaceae)[J]. Evolution, 1995, 49:727. |
| [25] | 陈川. 药用植物玄参的栽培起源、亲缘地理及东亚玄参系统发育研究[D]. 杭州:浙江大学, 2011. |
 2016, Vol. 47
2016, Vol. 47


