2. 济南市食品药品检验检测中心, 山东 济南 250001;
3. 山东亚特生态技术有限公司, 山东 临沂 266071
2. Jinan Food and Drug Control/Testing Center, Jinan 250001, China;
3. Shandong Yate Eco-tech Co. Ltd., Linyi 266071, China
木瓜属Chaenomeles Lindl. 植物属于蔷薇科,全世界共有5种,包括木瓜Chaenomeles sinensis Koehne、皱皮木瓜C.speciosa (Sweet) Nakai.、毛叶木瓜C. cathayensis (Hemsl.) Schneid.、西藏木瓜C. thibetica Yü及日本木瓜C. japonica (Thunb.) Lindl. ex Spach。木瓜为一味传统中药,《中国药典》2010年版规定:木瓜为蔷薇科植物贴梗海棠(皱皮木瓜)Chaenomeles speciosa (Sweet) Nakai. 的干燥近成熟果实。我国是木瓜属植物起源中心和分布中心,变异类型和品种资源十分丰富。木瓜属植物形态变异较大,种间过渡性状多,在亲缘关系和遗传多样性的研究中存在诸多分歧,因此需要寻求一种快速且准确的方法进行鉴别。
DNA条形码鉴定技术是通过比较一段通用DNA片段,对物种进行快速、准确的识别和鉴定[1]。目前研究者们已经提出了10多条植物候选DNA条形码序列[2],其中核糖体DNA(nrDNA)中的ITS2区具有进化速率快、高度重复、长度适中等优点,成为药用植物DNA条形码研究中最为重要候选序列之一[3]。Chen等[4]分析比较了7个候选DNA条形码,建议将ITS2作为药用植物标准 DNA 条形码,psbA-trnH作为ITS2的辅助序列对药用植物进行鉴定。对于木瓜属物种在分类学中难以区分的类群,采用DNA条形码可从基因水平上提供一种分类依据。本研究选用核基因ITS2片段和叶绿体基因psbA-trnH片段对木瓜属植物进行DNA分子鉴定,为解决分类学争议提供分子依据。
1 材料与方法 1.1 材料、试剂及仪器 1.1.1 材料2014年4月在山东亚特生物有限公司木瓜种植基地采集,采摘各植株的幼嫩叶片,硅胶干燥保存。原植物经山东中医药大学周凤琴教授鉴定为毛叶木瓜C. cathayensis (Hemsel) Schneid.、皱皮木瓜C. speciosa (Sweet) Nakai、日本木瓜C. japonica (Thunb.) Lindl ex Spach、西藏木瓜C. thibetica Y和光皮木瓜C. sinensis (Thouin) Koehne,数字影像信息及凭证标本保存于山东中医药大学。实验样品材料见表 1。
| 表 1 样品信息 Table 1 Samples information |
植物基因组DNA提取试剂盒和琼脂糖凝胶DNA回收试剂盒购自北京天根生物技术有限公司;2×Master Mix(Taq DNA Polymerase)(recombinant);D2000 plus DNA Ladder(100~5 000)(Solarbio);氯仿、β-巯基乙醇、无水乙醇(山东禹王实业有限公司);引物由上海生工有限公司合成。
1.1.3 仪器Takara PCR扩增仪(GJD35-TP600);DYY-8C型电泳仪和电泳槽(北京六一仪器厂);微波炉(美的);ESJ系列电子分析天平;数显恒温水浴锅HH-4(国华电器有限公司);离心机(德国Eppendorf);KEDA成像仪及操作系统(美国柯达公司);紫外分析仪UV-2000(上海天能科技有限公司); DNA测序仪3730XL(Applied Biosystems公司)。
1.2 方法 1.2.1 DNA提取实验材料均为硅胶干燥叶片,取样约20 mg,液氮充分研磨,利用DNA提取试剂盒提取DNA。
1.2.2 PCR扩增及测序利用ITS2、psbA-trnH 2个序列的通用引物分别对所提取的DNA中靶序列进行PCR扩增。反向PCR反应体积为20 μL,体系内含:2×Taq master mix 12.5 μL、ddH2O 9.5 μL、引物各1 μL(2.5 μmol/L)、DNA 模板1 μL(30 ng)。扩增程序为94 ℃,5 min;94 ℃、30 s,56 ℃、30 s,72 ℃、45 s,40个循环;72 ℃、10 min。PCR扩增产物电泳检测后,切胶纯化,使用ABI 3730XL测序仪双向测序。ITS、psbA-trnH 2个序列的引物序列见表 2。
| 表 2 DNA条形码引物 Table 2 DNA barcode primers information |
测序峰图利用CodonCode Aligner V 2.06(CodonCode Co.,美国)校对拼接,去除引物区,ITS2序列使用基于隐马尔可夫模型的HMMer注释方法除去两端的5.8S和28S区段获得ITS2间隔区序列[5]。psbA-trnH序列切除两端引物获得完整序列。然后,将所有序列用软件MEGA(Molecular Evolutionary Genetics Analysis)6.0分析比对并计算Kimura-2-parameter(K2P)距离,用NJ邻接法(neighbor-2-joining method)构建系统发育树。利用相似性搜索法(BLAST1)、最近距离法(Nearest distance)等方法进行鉴定分析。利用bootstrap(1 000次重复)检验各分支的支持率。利用Schultz等[6]建立的ITS2数据库及其网站(http://its2.bioapps.ibiozentrum.uni-wuerzburg.de)预测ITS2二级结构。
2 结果与分析 2.1 相似性搜索法鉴定结果进入NCBI对实验样品的ITS2、psbA-trnH序列使用BLAST方法进行鉴定和序列验证[7],结果显示实验样品与木瓜属查询序列的相似度达96%以上,此鉴定结果说明实验药材样本来源于蔷薇科木瓜属。
2.2 ITS2序列种内种间比较分析木瓜属5种不同种质植物(皱皮木瓜、光皮木瓜、西藏木瓜、日本木瓜、毛叶木瓜)序列比对后长度为225 bp,G+C量在69.6%(表 3)。对比实验中木瓜属19个样本的种间ITS2序列(图 1)发现共有40处位点变异,其中光皮木瓜的总变异率最低(0.88%),毛叶木瓜的总变异率最高(4.94%)。根据K2P参数遗传距离模型计算得到种间最大遗传距离为0.124,在光皮木瓜与皱皮木瓜之间。不同种间最小遗传距离0.005,在皱皮木瓜与日本木瓜之间,说明这2种亲缘关系较近。种内遗传距离分布于0~0.015。由此可知ITS2序列可以用于木瓜属植物的分子鉴定,但部分种间差异则不能通过ITS2序列的位点碱基差异区分开。
| 表 3 ITS2序列信息 Table 3 Main information of ITS2 sequences |
 |
“−”表示该位点碱基缺失,下同 “−” means the site base deletion, same as below 图 1 木瓜属植物的ITS2变异位点图 Fig.1 Variable sites of ITS2 sequence of plants in Chaenomeles Lindl. |
对比过程中发现各物种的psbA-trnH的长度不一,序列长度变异从260~313 bp波动,G+C量25.1%~28.5%,见表 4。对序列进行分析,结果日本木瓜和皱皮木瓜总变异率最低(0.64%),毛叶木瓜的总变异率最高(17.20%)。序列比对结果显示(图 2),把空位当作缺失发现,西藏木瓜和毛叶木瓜2个种在162 bp有一大段缺失53个碱基,与皱皮木瓜相比,日本木瓜、毛叶木瓜分别缺失1个碱基,光皮木瓜缺失4个碱基,种间的位点变异较少。木瓜属psbA-trnH序列K2P遗传距离分布于0~0.036,其中光皮木瓜与皱皮木瓜的K2P遗传距离最大,毛叶木瓜和西藏木瓜种间遗传距离为0,亲缘关系很近,与ITS2结果一致。psbA-trnH种间平均K2P遗传距离0.007,种内最大遗传距离为0.007。
| 表 4 psbA-trnH序列信息 Table 4 Main information of psbA-trnH sequences |
 | 图 2 木瓜属植物的psbA-trnH变异位点图 Fig.2 Variable sites of psbA-trnH sequence of plants in Chaenomeles Lindl. |
从木瓜属5个种的二级结构比较可以看出不同基原植物间ITS2二级结构非常相似,均由一个中心环及4个螺旋(Helix)区构成(图 3),比较木瓜属二级结构发现,其中IV区较为保守,主要差异在螺旋I、II、III区,其中光皮木瓜无论是从茎环(Loop)的数目、大小、角度和长度等方面,都与其他4种有很大区别。毛叶木瓜在螺旋II区较其他种缺少一个茎环,日本木瓜在螺旋I区多一个茎环,不同物种间茎环大小、位置有或多或少的区别,见图中方框所示。因此,通过ITS2二级结构,可以将木瓜属5个近缘种直观地鉴别出来。
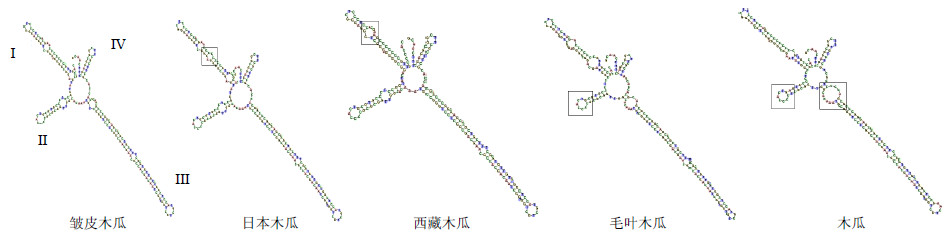 | 图 3 ITS2二级结构比较 Fig.3 Comparison onsecondary structure of ITS2 |
木瓜属植物分布广泛,种间性状交叉普遍,种间关系不清晰,导致木瓜属植物在分类处理上存在较大争议,而基于DNA条形码序列构建木瓜属系统发育树,不仅能通过树形图分支结果判断物种,还能在一定程度上还原其系统进化关系。
从ITS2序列聚类分析结果来看(图 4-A),木瓜属5个种种内的不同样本分别聚为一支,其中,样本中皱皮木瓜聚为一支,后与日本木瓜分支聚为一支,呈现较近的亲缘关系。西藏木瓜与毛叶木瓜分支聚为一支持率较高的分支,亲缘关系较高。而光皮木瓜形成支持率为100%单独分支,表现出单系性,与其他同属能够很明显区分开。依据ITS2序列构建的分子系统树可将木瓜属5个种很好地区分开。
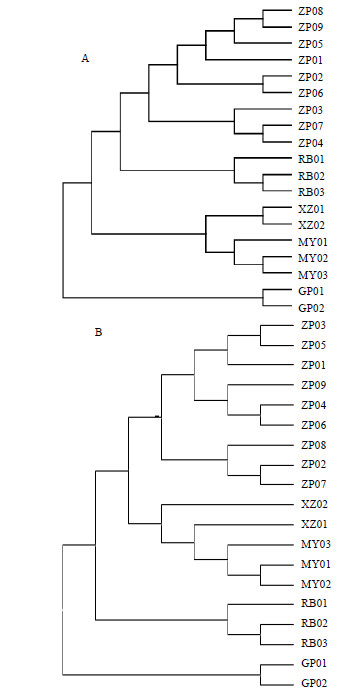 |
Bootsrap 1 000次重复,仅显示支持率≥50%的分支 Bootsrap 1 000 repeated, the support rate ≥ 50% were shown above branches 图 4 基于ITS2 (A) 和psbA-trnH (B) 序列构建NJ树 Fig.4 NJ tree based on ITS2 (A) and psbA-trnH (B) sequences |
psbA-trnH序列的NJ树(图 4-B)中,光皮木瓜与其他种间呈现明显的单系性;日本木瓜、毛叶木瓜、皱皮木瓜种质品种内优选聚类;西藏木瓜与毛叶木瓜种质共聚为一个类群,表明2个物种间亲缘关系密切。日本木瓜种质与皱皮木瓜种质亲缘关系较远。皱皮木瓜种质聚为一个类群,但与ITS2序列相比较,种内样品在树状图中位置不稳定。
3 讨论木瓜属植物变异类型多、变异幅度大,种间杂交容易,传统的形态分类鉴别法并不能完全区分各种质资源。近年来随着分子生物学的不断发展,已有研究通过RAPD、AFLP及SRAP等方法对木瓜种质的遗传多样性进行了分析[8, 9, 10, 11, 12, 13, 14],但在种间、种内显示出的多态性程度并不完全一致,且各种分子标记技术之间缺乏一定的相互印证。基于遗传物质DNA的条形码鉴定技术,具有稳定、可靠、不受外界影响的特点,是一种新的分子生物鉴定技术。
本实验应用DNA 条形码技术测定了木瓜属的ITS2和psbA-trnH序列,从分子水平证明了5个种间的亲缘关系。研究结果表明,ITS2序列在木瓜不同种质个体间存在丰富的变异,psbA-trnH序列的种内种间变异位点较少。ITS2序列种内种间的K2P遗传距离接近,说明遗传相似度高,亲缘关系较近。psbA-trnH序列K2P遗传距离显示光皮木瓜与皱皮木瓜之间遗传距离最远,而毛叶木瓜和西藏木瓜种间遗传距离较小,亲缘关系紧密,与ITS2结果一致。木瓜属5个种的ITS2二级结构存在明显不同,说明ITS2二级结构有助于更直观地鉴定物种。
本研究中ITS2序列信息聚类结果与psbA-trnH基本一致,5个不同种质分别聚为一支,可以很好地区分开,其中4个种的亲缘关系较近,仅有光皮木瓜表现出显著的单系性;2个聚类图中西藏木瓜与毛叶木瓜均聚为一支,显示密切的亲缘关系,本研究支持王明明等[15]对木瓜属品种亲缘关系的SRAP分析的研究结果。同时,王明明等[16]的数量分类学研究表明日本木瓜种系较为独立,与psbA-trnH聚类结果基本一致。ITS2序列各分支的支持率较高,在物种水平的鉴定成功率高,但psbA-trnH序列各分支的支持率不高,主要原因在于种内种间变异位点较少,各品种区分度不高。以上结论说明采用以ITS2序列为主,psbA-trnH序列为辅的鉴定方法能够从分子角度区分木瓜属的种间遗传性,对于探讨木瓜属不同物种间的分类学关系以及药材资源的合理开发利用亦具有重要的参考价值。
本研究结果验证了ITS2、psbA-trnH序列在木瓜属的鉴别能力,对于实现木瓜分子水平的快速准确鉴定具有重要意义,可为研究木瓜种质资源的起源和进化提供参考。本研究中DNA条形码候选序列ITS2与psbA-trnH片段表现了较好的物种鉴定能力,从分子水平揭示木瓜属各种间的亲缘关系,适用于木瓜属植物物种鉴定。
志谢:山东省农业科学研究院生物信息中心王兴军教授的课题组提供本研究分子实验平台。
| [1] | Hebert P D, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes[J]. Proc Biol Sci, 2003, 270(1512): 313-321. |
| [2] | Ning S P, Yan H F, Gang H, et al. Current advances of DNA barcoding study in plants[J]. Biodiv Sci, 2008, 16: 417-425. |
| [3] | 陈士林, 姚 辉, 宋经元, 等. 基于DNA barcoding (条形码) 技术的中药材鉴定[J]. 世界科学技术—中医药现代化, 2007, 9(3): 7-12. |
| [4] | Chen S L, Yao H, Han J P, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS ONE, 2010, 5: e8613. |
| [5] | Keller A, Schleicher T, Schultz J, et al. 5.8S-28S rRNA interaction and HMM-based ITS2 annotation[J]. Gene, 2009, 430: 50-57. |
| [6] | Schultz J, Muller T, Achtziger M, et al. The internal transcribed spacer2 database-a web server for (not only) low level phylogenetic analyses[J]. Nucl Acids Res, 2006, 34: W704-W707. |
| [7] | 陈士林, 庞晓慧, 姚 辉, 等. 中药DNA条形码鉴定体系及研究方向[J]. 世界科学技术—中医药现代化, 2011, 13(5): 747-754. |
| [8] | 陈 红, 张 雷, 吕晓贞, 等. 适于木瓜属植物AFLP分析用DNA提取方法研究[J]. 北方园艺, 2008(12): 161-163. |
| [9] | 尹长虹, 马 燕, 臧德奎. 木瓜属植物AFLP分析体系的建立与应用[J]. 北方园艺, 2010(13): 116-119. |
| [10] | 张亚利. 形态学性状及AFLP标记在木瓜海棠新种质鉴别和分类中的应用[J]. 江苏农科学, 2010(4): 145-147. |
| [11] | 陈 红. 木瓜属种质资源的RAPD、AFLP亲缘关系鉴定及遗传多样性分析[D]. 济南: 山东农业大学, 2008. |
| [12] | 臧德奎, 陈 红, 郑 林, 等. 木瓜属优良品种亲缘关系的AFLP分析[J]. 林业科学, 2009(8): 39-43. |
| [13] | 夏永秀, 曾秀丽, 廖明安, 等. SRAP 在西藏木瓜属种质资源研究中的应用[J]. 果树学报, 2010, 27(6): 1014-1018. |
| [14] | Li G, Quiros C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J]. Theor Appl Genet, 2001, 103: 455-461. |
| [15] | 王明明, 陈化榜, 王建华, 等. 木瓜属品种亲缘关系的SRAP分析[J]. 中国农业科学, 2010(3): 542-551. |
| [16] | 王明明, 王建华, 宋振巧, 等. 木瓜属品种资源的数量分类研究[J]. 园艺学报, 2009(5): 701-710. |
 2016, Vol. 47
2016, Vol. 47


