2. 平凉医学高等专科学校, 甘肃 平凉 744000;
3. 成都中医药大学, 四川 成都 610075;
4. 上海伯豪生物技术有限公司, 上海 201203
2. Pingliang Medical College of Gansu Province, Pingliang 744000, China;
3. Chengdu University of TCM, Chengdu 610075, China;
4. Shanghai Bohao Biotechnology Co., Ltd., Shanghai 201203, China
当归Angelica sinensis (Oliv.) Diels属伞形科(Umbelliferae)多年生草本植物,主产于甘肃、云南和四川等地。其中,甘肃岷县所产当归,产量大,质量好,畅销国内,驰名海外,历来被认为是中医用药的道地药材。岷县古称“秦州”,岷县所产当归,又有“秦归”之名。当归被视为补血、活血要药,临床应用十分讲究。当归根中含有大量具有药用活性的次级代谢产物,如当归多糖、阿魏酸、当归总黄酮和叶酸等[ 1 ]。当归多糖由鼠李糖、阿拉伯糖、甘露糖、葡萄糖、半乳糖组成,具有保肝、调节免疫、抗氧化、抗肿瘤、抗血小板聚集和促进骨髓造血等药理作用,与当归补血、活血功效相关[ 2,3 ]。阿魏酸可减少血栓素A2(TXA2)的生成,抑制血小板聚集,具有抗血栓等药理作用,为当归活血的物质基础,常常作为当归质量评价的标志性化合物[ 1,4,5,6,7 ]。
当归具有重要的药用价值和经济价值,但其基因组研究十分缺乏。在当归早期的基因研究中,张西玲等[ 8 ]用DNA测序技术建立岷归的rRNA基因图谱;Yu等[ 9 ]用cDNA-AFLP分析技术阐释岷归抽薹的分子调控机制,均有助于当归品种品质鉴定,却难以阐明当归次生代谢产物生物合成的基因表达调控机制。近年来,RNA高通量测序技术的快速发展促进了植物基因表达研究,并应用于西洋参、丹参、虎杖和甘草等多种药用植物[ 10,11 ]。Illumina HiSeq 2000第2代高通量测序技术平台以高通量、高分辨率、高精度和价格低廉等优势发挥着巨大作用,已应用于人类基因组测序[ 10,12 ]。本研究首次用Illumina HiSeq 2000技术测序采收期当归根转录组,分析其转录组特性。本研究还发现了已注释当归基因序列主要分布于目前研究较深入的7种重要经济作物,并识别了与当归重要药效物质(阿魏酸、当归多糖、当归总黄酮等)生物合成相关的基因序列,阐释了当归补血、活血功效的分子生物学基础。 1 材料与方法 1.1 转录组测序 1.1.1 样本采集
在甘肃岷县清水乡当归主要生产区采收期现场收集5个代表性新鲜当归样品,并经平凉医学高等专科学校药用植物学程亚青教授鉴定为当归Angelica sinensis (Oliv.) Diels,分别取归头、归尾各0.5 g,共10个样本,立即投入液氮中保存,干冰运输。 1.1.2 RNA提取与纯化
从当归头、当归尾分别提取总RNA[ 13 ],所得总RNA经ND-1000分光光度计和Agilent2100系统电泳质检合格后备用。每个样本各取3 μg RNA混合得到1个混合样本,并采用RNeasy Micro kit按其操作流程纯化20 μg总RNA,所得产物用于测序文库构建。 1.1.3 测序文库构建与质检
按照“TruSeq RNA Sample Preparation Guide”对纯化后的总RNA进行mRNA的分离、片段化、第一链cDNA合成、第二链cDNA合成、末端修复、3’末端加A、连接接头、富集等步骤,完成测序样本文库构建。所建测序文库经Qubit®2.0 Fluorometer和Agilent 2100系统检测合格后,在Illumina HiSeq 2000测序仪上完成双端测序。转录组测序实验委托上海伯豪生物有限公司完成。 1.2 数据预处理
将预处理后测序数据的质量信息进行统计,碱基测序质量值范围为27~40。测序错误率用E表示,碱基测序质量值用sQ表示,其中sQ=-10 lgE。
去除总体质量偏低的读序,去除读序中含有的模糊N碱基、去除长度小于20 bp的读序以及含有的接头序列后所得读序为干净读序。 1.3 短读序组装 1.3.1 原始序列组装
将样本测序数据读序,应用CLC Genomics Workbench(version: 5.5)[ 14,15 ]的scaffolding contig算法进行从头合成拼接,此阶段拼接得出的序列称之为初始序列。 1.3.2 最终序列组装
应用CAP3 EST拼接软件对两个样本得到的初始短读序进行混合拼接,得到一个最终序列集合。采用CAP3软件的策略为对初始序列进行2次拼接,第1次使用宽松拼接参数进行初始拼接,第2次使用严格拼接参数进行拼接。 1.4 功能基因序列注释、功能分类和代谢路径分析 1.4.1 功能基因序列注释
将最终序列与蛋白质数据库(UniProt-date:2012.09)进行Blastx比对(将核酸序列翻译为蛋白,再进行比对)[ 16 ]。以trEMBL+swissprot,E<1e-5为筛选条件。UniProt数据库是目前对蛋白功能结构注释最为详细且准确的蛋白数据库,大概包括22 000 000个蛋白。 1.4.2 功能基因序列表达分析
以最终序列作为参考,将样本的读序进行参考映射,从而得到样本在每个功能基因序列中的一个读序的覆盖度,应用RPKM量化标准对读序覆盖度进行表达量计算,公式为RPKM=覆盖整个功能基因序列的读序数目/功能基因序列长度×该样本所有参与拼接的总读序数目×109。 1.4.3 基因本体(gene
ontology,GO)分类 GO是一个国际标准化的基因功能分类体系,提供了一套动态更新的标准词汇表来全面描述生物体中的基因和基因产物的属性。根据UniProt蛋白质数据库的注释结果,应用Blast2 GO算法进行GO功能分类,得到分子功能、细胞成分、生物学过程各个层次所占数目,从宏观上认识当归基因功能分布特征。 1.4.4 代谢路径注释
Kyoto Encyclopedia of Genes and Genomes(KEGG)是系统分析基因产物在细胞中的代谢途径以及这些基因产物的功能数据库。应用KEGG KEGG Automatic Annotation Server(KEGG KAAS)在线pathway比对分析工具对拼接得到的功能基因序列进行KEGG映射分析,进一步得到功能基因序列的代谢路径注释。 2 结果与分析 2.1 转录组测序产出及序列组装
由Illumina HiSeq 2000高通量测序获得66 432 540个原始读序。经去除总体质量偏低和含有接头序列的读序后所得到的干净读序为62 539 584个。由这些短读序组装出110 706个重叠群(N),平均长度556 bp,N50 930 bp。因后续注释分析基于功能基因序列,因此应用CLC Genomics Workbench(version:5.5)的scaffolding contig算法进行从头合成拼接出65 674个功能基因序列(unigene),平均长度1 004 bp,N50 1 544 bp(表 1)。再应用CAP3 EST拼接软件对2个样本得到的功能基因序列进行混合拼接,得到63 585个功能基因序列,平均长度1 035 bp,N50 1 608 bp(表 1)。从图 1可知,约16 000个功能基因序列长度为400 nt。
| 表 1 序列组装统计 Table 1 Sequence assembe statistics |
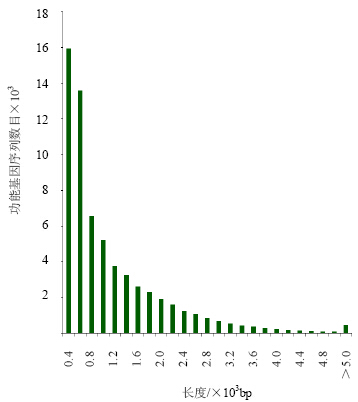 | 图 1 当归功能基因序列长度分析Fig. 1 Analysis on unigene sequence length in A. sinensis |
目前无药用植物当归全基因组测序,因此将最终功能基因序列与UniProt(date:2012.09)库进行Blastx比对,注释上的功能基因序列数目为30 432个,注释比例为47.9%。经Blastx比对,发现当归功能基因序列主要分布于目前研究较深入的7种重要经济作物,如葡萄、蓖麻、毛果杨、大豆、苜蓿、拟南芥和烟草。在映射到7种重要经济作物的当归功能基因序列中,完全匹配程度大于40%的功能基因序列见表 2。其中,RPKM值最高为25.84,最低为0.79,说明Illumina HiSeq 2000高通量测序能检测低水平的同源基因表达。序列分布的同源性以E<1e-5为筛选条件,即E值越小,同源性越可靠。
| 表 2 当归与重要经济作物功能基因序列的匹配程度 Table 2 Unigene identity between A. sinensis and important economic crops |
GO的功能是按照树形结构进行排布的,一般层数越深,功能越详细,但相应的冗余也随之增加,目前第二层功能用的比较广泛。从图 2可知,以生物学过程、细胞过程和细胞成分过程3方面对当归功能基因序列进行阐释,发现参与代谢过程的功能基因序列数目为13 688个(占已注释序列比例的44.98%),参与催化活性的功能基因序列数目为10 684个(占已注释序列比例的35.11%)。研究发现,当归中微量元素量丰富,能激活体内酶的合成与分泌,与酶催化的细胞代谢过程相关。
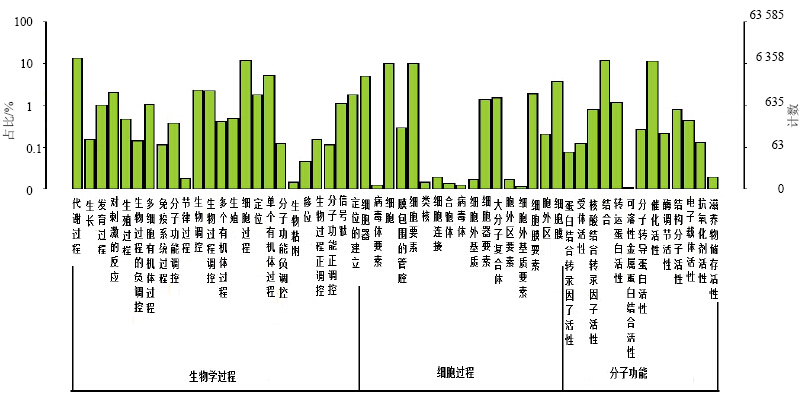 | 图 2 当归功能基因序列的GO功能Fig. 2 GO functional distribution in A. sinensis unigene sequences |
为了识别当归中活性高的代谢路径,将63 585个功能基因序列进行KEGG映射分析,共映射到269条代谢路径。包含功能基因序列最多的是代谢路径(2 242个)、次级代谢产物生物合成(1 159个)、不同环境中的微生物代谢(550个)。类苯基丙烷生物合成、黄酮类化合物生物合成、叶酸生物合成和N-聚糖生物合成等路径参与当归根重要药效物质生物合成,如阿魏酸、当归总黄酮、叶酸和当归多糖(表 3)。阿魏酸源自类苯基丙烷生物合成路径,当归多糖源自N-聚糖生物合成路径,当归总黄酮源自黄酮类化合物生物合成路径,叶酸源自叶酸生物合成路径。从表 3可知,分别有127、69、70、94个功能基因序列映射到上述4种代谢路径。
| 表 3 与当归重要药效物质相关的次生代谢产物生物合成路径 Table 3 Biosynthetic pathway of secondary metabolites involved in major active substances of A. sinensis< |
在类苯基丙烷生物合成路径中,以苯丙氨酸为原料,经苯丙氨酸氨裂解酶、桂皮酸-4-羟化酶等酶合成阿魏酸。阿魏酸可经阿魏酸-5-羟化酶代谢成5-羟阿魏酸。类苯基丙烷生物合成路径的中间代谢产物,如桂皮酸、香豆酸、咖啡酸、阿魏酸可参与黄酮类化合物生物合成路径的代谢调控;如咖啡酰辅酶A经咖啡酰辅酶A邻甲基转移酶转化为阿魏酰辅酶A,然后再经查耳酮合酶合成四羟基-3-甲氧基查耳酮,成为当归总黄酮中的一类药效成分。在叶酸生物合成路径中,叶酰聚谷氨酸合酶和二氢叶酸还原酶/胸苷酸合酶是叶酸合成的2个关键调控酶。
糖基转移酶催化单糖从糖供体转移到糖受体分子,参与天然产物黄酮类、类苯基丙烷生物合成路径。经研究证实,当归多糖在补血与止血方面起双向调节作用,是当归的主要药效活性成分,由鼠李糖、阿拉伯糖、甘露糖、葡萄糖、半乳糖组成。在当归转录组数据中,发现糖基转移酶、糖苷酶参与调控N-聚糖生物合成路径,如β-1,4-甘露糖基糖蛋白β-1,4-N-乙酰葡糖胺基转移酶、甘露糖基-寡聚糖α-1,2-甘露糖苷酶、α-1,6-甘露糖基糖蛋白β-1,2-N-乙酰葡糖胺基转移酶、α-1,3-葡糖苷酶、α-1,3-葡糖基转移酶等。其中,RPKM值最高为91.99,RPKM值最低为0.54。经这些注释提供了有价值的资源,有助于研究特定过程、功能和路径,也有助于识别次级代谢产物生物合成路径的新基因。 3 讨论 3.1 Illumina HiSeq 2000二代高通量测序技术可识别非模式物种功能注释基因
近年来,随着新一代高通量测序技术的广泛应用,植物基因组研究得到快速发展[ 17 ],但是识别当归根转录组特性的基因序列研究还比较少。该类研究有助于开发天然药物,使当归生药研究与生命科学前沿技术深度融合。本研究首次采用Illumina HiSeq 2000高通量测序平台对当归根转录组进行测序,获取了63 585个当归功能基因序列,并与UniProt蛋白数据库进行blastx比对,发现已注释当归基因序列主要分布于目前研究较深入的7种重要经济作物,如葡萄、蓖麻、毛果杨、大豆、苜蓿、拟南芥和烟草。当归与不同物种序列相似性的比较,可绘制系统进化树图谱,为阐明栽培当归的起源物种提供理论依据。本研究提示,Illumina HiSeq 2000第2代高通量测序技术可检测非模式物种大量功能注释基因,可进一步探测基因组研究缺乏物种功能基因变化的网络模式[ 18 ]。 3.2 次级代谢基因
通过Uniprot蛋白质数据库注释并比对了30 432个unigenes,获取基因描述和保守的蛋白质结构域[ 19 ]。应用Blast GO算法对注释序列进行GO分类,发现当归根的转录组特性主要与酶催化的代谢过程相关。进一步对当归功能基因序列进行代谢路径注释,发现参与次生代谢产物生物合成路径的功能基因序列为1 159个,可能参与当归药用活性物质代谢调控。当归视为补血、活血要药,被称为“血中之圣药”,对其补血、活血微观物质基础研究,有助于阐释当归补血、活血的科学内涵。
当归多糖能增加外周血细胞、白细胞、血红蛋白及骨髓有核细胞数,这种作用特别是在外周血细胞减少和骨髓受到抑制时尤为明显,可用于治疗各种原因引起的贫血疾病[ 20 ],提示了当归补血的主要活性成分为当归多糖。当归多糖由鼠李糖、阿拉伯糖、甘露糖、葡萄糖、半乳糖组成,其形成需糖基转移酶和糖苷酶的调控。
糖基转移酶在生物体内催化活化的糖连接到不同的受体分子,如蛋白、核酸、寡糖、脂质和小分子上[ 21 ]。当归多糖可以甘露糖、葡萄糖和乙酰葡糖胺为原料参与N-聚糖生物合成路径(69个功能基因序列参与)的调控。在RPKM值中,以β-1,4-甘露糖基糖蛋白β-1,4-N-乙酰葡糖胺基转移酶为最高。识别糖基转移酶和糖苷酶相关基因序列,对研究当归多糖参与的特定代谢路径有重要意义。
叶酸经人体吸收后,将转化为四氢叶酸,并作为一碳单位的辅酶参与红细胞、血红蛋白DNA的合成,有助于提高机体红细胞、血红蛋白的数量,可用于治疗贫血疾病。生物体内叶酸含量的高低取决于叶酸生物合成路径,受叶酰聚谷氨酸合酶[ 22 ]和二氢叶酸还原酶/胸苷酸合酶[ 23 ]的调控,其中以叶酰聚谷氨酸合酶的RPKM值为最高。识别叶酸生物合成路径相关基因,可佐证当归根中叶酸的量,反映当归补血功效的高低。经上述分析,N-聚糖生物合成路径及叶酸生物合成路径与当归补血功效相关,有助于阐释当归补血的分子科学内涵。
阿魏酸次级代谢基因是当归活血化瘀的物质基础[ 24 ],参与类苯基丙烷生物合成路径。在此代谢路径中,以苯丙氨酸为原料,先后生成桂皮酸和香豆酸,最后合成阿魏酸,阿魏酸又可代谢成5-羟阿魏酸。在当归根中,苯丙氨酸氨裂解酶[ 25 ]和桂皮酸-4-羟化酶[ 26 ]可促进阿魏酸合成,阿魏酸-5-羟化酶[ 27 ]可降低阿魏酸的量。识别上述3个酶的功能基因序列,可佐证当归根中阿魏酸量的高低,反映当归活血化瘀的功效,为当归质量鉴定、分子育种及栽培提供理论依据。
类苯基丙烷合成路径的中间代谢产物,如咖啡酸、阿魏酸,可经黄酮化合物生物合成路径生成四羟基-3-甲氧基查耳酮,属于当归总黄酮的一类药效成分。桂皮酸-4-羟化酶可共同调控类苯基丙烷生物合成路径和黄酮类化合物生物合成路径,影响阿魏酸和当归总黄酮的量。查耳酮合酶[ 28 ]是合成四羟基-3-甲氧基查尔酮的一种关键酶,其活性高低可反映当归总黄酮的量。因此,类苯基丙烷生物合成路径及黄酮类化合物生物合成路径与当归活血功效相关。
本研究首次发现了与5种重要经济作物序列完全匹配程度大于40%的当归功能基因序列。识别了与当归补血、活血物质基础相关的基因序列及代谢路径,阐明了当归视为补血、活血要药的分子生物学基础,为中药生药学研究提供示范。
| [1] | 杨应文, 王亚丽, 萨日娜, 等. 不同生长期当归1H-NMR指纹图谱的研究[J]. 波谱学杂志, 2013, 3(1): 70-79. |
| [2] | 韦 玮, 龚苏晓, 张铁军, 等. 当归多糖类成分及其药理作用研究进展[J]. 药物评价研究, 2009, 32(2): 130-134. |
| [3] | 汪诺舟. 当归多糖的药理作用与研究进展[J]. 中国现代医学杂志, 2012, 22(19): 67-69. |
| [4] | 刘雪东, 李伟东, 蔡宝昌. 当归化学成分及对心脑血管系统作用研究进展[J]. 南京中医药大学学报, 2010, 26(2): 155-157. |
| [5] | 萨日娜, 朱书强, 潘新波, 等. 不同生长期当归根挥发油中Z-藁苯内酯和正丁烯基酞内酯含量的动态变化研究[J]. 中药材, 2012, 35(11): 1738-1742. |
| [6] | 杨英来, 胡 芳, 刘小花, 等. 当归补气活性部位的谱效关系研究[J]. 中草药, 2013, 44(23): 3346-3351. |
| [7] | 张 静, 杨义芳, 吴春珍, 等. 当归-川芎药对超临界提取物配伍红花抗心肌缺血的谱效关系研究[J]. 中草药, 2013, 44(14): 1944-1950. |
| [8] | 张西玲, 姬可平, 李应东, 等. DNA测序技术建立甘肃当归和大黄种子rRNA基因图谱的研究[J]. 中药材, 2003, 26(7): 481-484. |
| [9] | Yu G, Duan J, Yan H, et al. cDNA-AFLP analysis of gene expression differences between the flower bud and sprout-shoot apical meristem of Angelica sinensis[J]. Genet Mol Biol, 2011, 34(2): 274-279. |
| [10] | 郝大程, 马 培, 穆 军, 等. 中药植物虎杖根的高通量转录组测序及转录组特性分析[J]. 中国科学: 生命科学, 2012, 42(5): 398-412. |
| [11] | 吴 琼, 孙 超, 陈士林, 等. 转录组学在药用植物研究中的应用[J]. 世界科学技术—中医药现代化, 2010, 12(3): 457-462. |
| [12] | Joy N, Asha S, Mallika V, et al. De novo transcriptome sequencing reveals a considerable bias in the incidence of simple sequence repeats towards the downstream of ‘Pre-miRNAs' of black pepper[J]. PLoS One, 2013, 8(3): e56694. |
| [13] | 张年辉, 韦振泉, 何军贤, 等. 一种高效经济的高质量植物RNA提取方法[J]. 生物化学与生物物理进展, 2004: 31(10): 947-949. |
| [14] | Su C L, Chao Y T, Chang Y C, et al. De Novo assembly of expressed transcripts and global analysis of the phalaenopsis aphrodite transcriptome[J]. Plant Cell Physiol, 2011, 52(9): 1501-1514. |
| [15] | Bräutigam A, Mullick T, Schliesky S, et al. Critical assessment of assembly strategies for non-model species mRNA-Seq data and application of next-generation sequencing to the comparison of C3 and C4 species[J]. J Exp Bot, 2011, 62(9): 3093-3102. |
| [16] | Altschul S F, Gish W, Miller W, et al. Basic local alignment search tool[J]. J Mol Biol, 1990, 215(3): 403-410. |
| [17] | Zhang L, Jia H, Yin Y, et al. Transcriptome analysis of leaf tissue of raphanus sativus by RNA sequencing[J]. PLoS One, 2013, 8(11): e80350. |
| [18] | Leung R K, Dong Z Q, Sa F, et al. Quick, sensitive and specific detection and evaluation of quantification of minor variants by high-throughput sequencing[J]. Mol Biosyst, 2013, 10(2): 206-214. |
| [19] | Taylor D, Cawley G, Hayward S. Classification of domain movements in proteins using dynamic contact graphs[J]. PLoS One, 2013, 8(11): e81224. |
| [20] | Zhao L, Wang Y, Shen H L, et al. Structural characterization and radioprotection of bone marrow hematopoiesis of two novel polysaccharides from the root of Angelica sinensis[J]. Fitoterapia, 2012, 83(8): 1712-1720. |
| [21] | Albesa-Jové D, Giganti D, Jackson M, et al. Structure-function relationships of membrane associated GT-B glycosyltransferases[J]. Glycobiology, 2013, 24(2): 108-124. |
| [22] | Jiang L, Liu Y, Sun H, et al. The mitochondrial folylpolyglutamate synthetase gene is required for nitrogen utilization during early seedling development in Arabidopsis[J]. Plant Physiol, 2013, 161(2): 971-989. |
| [23] | Francis K, Stojkovic V, Kohen A. Preservation of protein dynamics in dihydrofolate reductase evolution[J]. J Biol Chem, 2013, 288(50): 35961-35968. |
| [24] | Gim S A, Sung J H, Shah F A, et al. Ferulic acid regulates the AKT/GSK-3β/CRMP-2 signaling pathway in a middle cerebral artery occlusion animal model[J]. Lab Anim Res, 2013, 29(2): 63-69. |
| [25] | Jaliani H Z, Farajnia S, Mohammadi S A, et al. Engineering and kinetic stabilization of the therapeutic enzyme anabeana variabilis phenylalanine ammonia lyase[J]. Appl Biochem Biotechnol, 2013, 171(7): 1805-1818. |
| [26] | Singh K, Kumar S, Rani A, et al. Phenylalanine ammonia-lyase (PAL) and cinnamate 4-hydroxylase (C4H) and catechins (flavan-3-ols) accumulation in tea[J]. Funct Integr Genomics, 2009, 9(1): 125-134. |
| [27] | Al-Haddad J M, Kang K Y, Mansfield S D, et al. Chemical responses to modified lignin composition in tension wood of hybrid poplar (Populus tremula × Populus alba)[J]. Tree Physiol, 2013, 33(4): 365-373. |
| [28] | Cho Y B, Jones S I, Vodkin L. The transition from primary siRNAs to amplified secondary siRNAs that regulate chalcone synthase during development of Glycine max seed coats[J]. PLoS One, 2013, 8(10): e76954. |
 2015, Vol. 46
2015, Vol. 46



