2. 广东省微生物研究所 省部共建华南应用微生物国家重点实验室 广东省菌种保藏与应用重点实验室 广东省微生物应用新技术公共实验室, 广东 广州 510070;
3. 内蒙古医科大学基础医学院, 内蒙古 呼和浩特 010110
2. State Key Laboratory of Applied Microbiology Southern China, Guangdong Open Laboratory of Applied Microbiology, Guangdong Provincial Key Laboratory of Microbial Culture Collection and Application, Guangdong Institute of Microbiology, Guangzhou 510070, China;
3. College of Basic Medicine, Inner Mongolia Medical University, Hohhot 010110, China
沉香是我国传统名贵中药,为瑞香科(Thymelaeaceae)植物白木香Aquilaria sinensis (Lour.) Gilg. 含有黑色树脂的木材[1],其味辛、苦,性微温,具行气止痛、温中止呕、纳气平喘等功效[2]。健康的白木香树体不能产生沉香类物质,只有在外界物理、化学伤害或真菌的刺激作用下才能够产生,故沉香资源十分珍稀。
沉香的成分主要包括倍半萜类(挥发性成分)、2-(2-苯乙基) 色酮类、芳香族类、三萜类及脂肪酸类化合物,其中前三者是沉香的特征性成分。倍半萜类化合物为沉香主要药效成分,药理学实验证明该类化合物对神经系统有明显的生理活性[3]。目前大量不同植物种类的倍半萜类合成酶基因已经被成功克隆[4],但沉香倍半萜合酶相关基因的生物合成研究并不多,仅克隆得到主产物为愈创木烯型倍半萜的合成酶基因[5, 6]。因此,研究沉香倍半萜合成酶基因对于阐明倍半萜生物合成途径,进而揭示白木香结香的分子机制具有重要意义。
本课题组前期对化学诱导后的白木香转录组进行了测序和分析[7],获得多条预测为倍半萜合成酶基因的编码序列,并首次成功从白木香总RNA中克隆到可能编码大根香叶烯-D-合成酶的基因(As-SesTPS)[8]。本研究选取了与As-SesTPS同一条倍半萜代谢通路中的另一条编码序列,该序列经生物信息学分析后预测其为一种新的倍半萜合成酶关键基因,与As-SesTPS编码不同结构类型的倍半萜合成酶蛋白。利用RT-PCR扩增,在诱导结香后的白木香cDNA中克隆得到该序列,命名为白木香倍半萜合成酶(As-SesTPS1)基因,将其在基因组进行验证,运用生物信息学的方法对该序列进行同源性分析,预测其编码蛋白及各种理化性质,最后通过实时荧光定量PCR技术对As-SesTPS1基因在诱导结香后的白木香不同部位的表达进行差异对比分析,为后续进一步研究其功能及白木香倍半萜生物合成途径奠定基础。
1 材料沉香样品(结香部位)、白木样品(白木部位)和过渡样品(结香与白木过渡部位)取自经化学诱导后已结香的白木香植株[7],经广东药学院中药学院严寒静副教授鉴定为白木香Aquilaria sinensis (Lour.) Gilg.。参照吴宏清等[8]方法配制改良异硫氰酸胍-CTAB提取液;DNase I,RNase-free(Thermo公司);植物基因组提取试剂盒(天根生化科技有限公司);Promega第一链合成试剂盒;Ex Taq DNA聚合酶,Ex Taq HS DNA聚合酶(Takara公司);零背景快速克隆试剂盒(天根生化科技有限公司);超纯dNTP(Takara公司);SYBR Green qPCR Mix(东盛生物公司);主要化学试剂为国产分析纯,引物委托华大基因(广州)有限公司合成(表 1)。
| 表 1 引物序列 Table 1 Primer sequences |
参照改良异硫氰酸胍-CTAB提取方法[9],以1%的非变性琼脂糖凝胶电泳检测RNA的完整性,采用分光光度法检测RNA的量和纯度。以提取的RNA样品为模板,使用Promega公司的cDNA第一链合成试剂盒合成第一条链,获得的cDNA样品于-20 ℃贮存备用。
2.2 白木香As-SesTPS1的克隆根据前期白木香转录组测序获得的长度为1853 bp的CL5155.Contig3_B1序列(KM881473),设计特异性引物,上游引物C5155.1,下游引物C5155.2。以“2.1”项方法获得的cDNA为模板进行PCR扩增,反应体系50 μL,扩增程序:94 ℃预变性3 min,94 ℃变性45 s,56 ℃退火45 s,72 ℃延伸110 s,30个循环后72 ℃延伸10 min。PCR产物经1%琼脂糖凝胶电泳后,回收目的条带,使用零背景快速克隆试剂盒进行克隆,挑取单克隆转化至大肠杆菌TOP10后,菌落PCR验证阳性克隆,送菌液至华大基因进行测序,将测序结果上传至ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)分析以确定是否获得全长。
2.3 白木香As-SesTPS1的扩增取沉香样品约100 mg加入液氮充分研磨;使用植物基因组提取试剂盒提取沉香样品基因组,引物同“2.2”项方法,以提取的基因组为模板进行PCR扩增,反应体系50 μL。反应条件为94 ℃预变性5 min,94 ℃变性45 s,56 ℃退火45 s,72 ℃延伸2 min,30个循环后72 ℃延伸10 min。PCR产物经1%琼脂糖凝胶电泳后,回收测序。
2.4 As-SesTPS1基因的生物信息学分析使用Lasergene 7.1的EditSeq软件将As-SesTPS1基因翻译成氨基酸序列,并用NCBI的Nr数据库进行BLASTp分析,获得相似度最高的序列,预测编码蛋白质;使用GENEDOC软件将预测的氨基酸序列与在NCBI比对相似度最高的5条序列以及本课题组先前获得的一条倍半萜合成酶基因As-SesTPS一并进行保守序列分析;使用MEGA5软件将预测的氨基酸序列与GenBank上的其他序列进行系统进化分析,采用邻接法(neighbor-joining,NJ)法构建系统进化树;ExPASy在线服务器的ProtParam(http://web.expasy.org/protparam/)预测蛋白质的基本理化性质;PSORT服务器(http://wolfpsort.org/)预测蛋白质的亚细胞定位;Predictprotein在线网站(http://www.predict protein.org/)进行蛋白质二级结构分析;使用Swiss-model(http://swissmodel.expasy.org/)进行三维结构预测;使用Discovery Studio软件进行蛋白三级结构建模。
2.5 As-SesTPS1基因的表达水平分析按“2.1”项方法从沉香样品、过渡样品和白木样品中提取RNA并逆转录合成cDNA样品。选取Histone基因为内参基因[10],以上述3个不同样品的cDNA为模板,进行实时荧光定量PCR扩增。内参Histone基因上游引物HF,下游引物HR,预测扩增大小200 bp左右;目的基因As-SesTPS1上游引物Q4.1,下游引物Q4.2,预计扩增大小200 bp左右。首先用常规PCR进行扩增,检测引物特异性,PCR条件同“2.3”项方法,如产物通过琼脂糖凝胶电泳检测可见符合预测大小的清晰条带,再采用“三步法”,反应体系20 μL,其中上游引物和下游引物各0.4 μL,样品cDNA模板0.5 μL,2×SYBR Green qPCR Mix 10 μL,补超纯水至20 μL。反应条件为94 ℃、4 min,94 ℃、30 s,56 ℃退火30 s,72 ℃、40 s,35个循环,20 min内从60 ℃升温到95 ℃,每个样品重复3次,空白对照以超纯水代替cDNA作为模板,qPT-PCR以Histone为内参基因。数据处理采用相对定量法,参照2-ΔΔCt方法进行计算分析[11]。
3 结果与分析 3.1 白木香总RNA的提取按照“2.1”项方法提取沉香样品、过渡样品和白木样品的RNA,1%琼脂糖凝胶电泳检测结果见图 1。结果显示所提RNA的28 S与18 S条带明亮清晰,RNA 28 S亮度约为18 S亮度的2倍,且两条带均无明显拖尾。经紫外分光光度计检测A260/A280大小在1.8~2.0,RNA完整性较好,未发生降解,可满足后续实验要求。
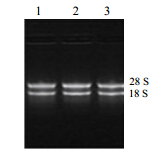 | 1-白木样品 2-过渡样品 3-沉香样品 1-whitewood sample 2-transition sample 3-agarwood sample图 1 白木香总RNA电泳图Fig. 1 Electrophoretogram of total RNA from A. sinensis |
根据前期白木香转录组倍半萜代谢通路差异表达基因测序结果分析,选取长度为1 853 bp的CL5155.Contig3_B1序列,将该序列进行CD-search分析,发现其可能编码倍半萜合成酶。将其进行ORF Finder分析,发现该序列包含完整的开放阅读框(ORF)。根据完整开放阅读框设计特异性引物,以沉香样品逆转录cDNA为模板进行扩增,在1 700 bp左右有明显特异性条带,初步认为得到目的基因(图 2)。将PCR产物纯化后克隆,挑取阳性菌落进行菌液PCR验证并测序,测序结果经Lasergene 7.1的SeqMan软件拼接,对该序列进行ORF查找,得到完整的编码序列,长1 671 bp,编码556个氨基酸,命名为As-SesTPS1基因(KM881473)。
 | 1-以cDNA为模板的As-SesTPS1扩增 2-以基因组DNA为模板的As-SesTPS1扩增 3-阴性对照 M-Marker 1-As-SesTPS1 amplification from cDNA 2-As-SesTPS1 amplification from genomic DNA 3-negative control M-Marker图 2 As-SesTPS1扩增Fig. 2 As-SesTPS1 amplifications |
以沉香样品的基因组为模板进行扩增,可得到1条约2 200 bp条带(图 2)。将此条带测序后的拼接结果上传至NCBI的Spidey(http://www.ncbi.nlm.nih.gov/IEB/Research/Ostell/Spidey/)进行分析,发现该序列的内含子核苷酸序列符合典型的TG-AG拼接点序列特征,且整个序列中分散着富含碱基AT或T的区段;将As-SesTPS1基因与其进行Spidey比对,发现As-SesTPS1与该序列外显子的同源性为100%。该结果从基因组验证了扩增得到As-SesTPS1基因的正确性。
3.4 白木香As-SesTPS1相似性及同源性分析将As-SesTPS1基因编码的氨基酸序列在NCBI上进行BLASTp分析,发现其与多条倍半萜合成酶基因具有较高相似性,其中相似性最高的为白木香δ-愈创木烯合成酶(delta-guaiene synthase,AFV99466.1)基因,达54%,与越南沉香δ-愈创木烯合成酶(delta-guaiene synthase,AEG77020.1)基因,可可δ-杜松烯合成酶(delta-cadinene synthase,XP_007021123.1)基因,刺五加α-古巴烯合成酶(alpha-copaene synthase,ADK94034.1)基因,葡萄大根香叶烯D合成酶 [(-)-germacrene D synthase,NP_001268213.1]基因也有较高的同源性,分别为53%、48%、48%、45%;与课题组之前克隆得到的倍半萜合成酶基因As-SesTPS的相似性为41%。运用GENEDOC 软件将As-SesTPS1基因编码的氨基酸序列与相似基因编码的6条氨基酸序列共同比对,结果显示As-SesTPS1基因具有植物倍半萜合成酶家族典型的保守序列RRx8W和DDxxD(图 3),推测其可能编码植物倍半萜合成酶类蛋白。
 | 黑色表示完全相同,灰色表示部分相同 Black represent identical and gray represent partly identical图 3 As-SesTPS1编码蛋白与已知倍半萜合成酶蛋白的相似性分析Fig. 3 Similarity analysis of As-SesTPS1-coding protein and other known sesquiterpene synthase proteins |
萜类合成酶基因根据其氨基酸序列的相关性以及功能的评价,分为7大亚家族TPSa~TPSg,使用MEGA5.0软件中的NJ法构建聚类树(图 4),从聚类结果可以看出,初级代谢相关萜类合成酶分别聚类为TPSc和TPSe亚家族,而次级代谢相关的被子植物单萜合成酶、倍半萜合成酶和裸子植物萜类合成酶分别聚类为TPSb、TPSa、TPSd。TPSf和TPSg亚家族成员酶产物为非环化单萜,其中TPSf是进化关系比较遥远的一个分支。本研究中的目的基因As-SesTPS1被分到由被子植物倍半萜合成酶基因组成的TPSa亚族中,可确定其编码倍半萜合成酶类蛋白。
 | 图 4 植物萜类合成酶基因家族系统进化树Fig. 4 Phylogenetic tree of genes in plant TPS family |
利用ExPASy Proteomics Server的在线软件Protparam(http://www.cn.expasy.org/tools/protparm.html)对As-SesTPS1蛋白的理化性质进行预测分析,推测其分子式为C2890H4481N747O859S22,相对分子质量为641 400,等电点为5.14,为酸性蛋白,正电残基(Arg+Lys)为89,负电残基(Asp+Glu)为64。其半衰期在体外哺乳动物网织红细胞为30 h,在酵母体内大于20 h,在大肠杆菌体内大于10 h。不稳定系数为44.57,为不稳定蛋白,脂肪系数为90.18。对该氨基酸序列进行亲疏水性分析,结果显示其总平均疏水指数(grand average of hydropathicity,GRAVY)为-0.343,预测其为亲水性蛋白。通过TMHMM软件分析得知,该编码蛋白质无跨膜区域,全部位于膜外。通过PSORT服务器的分析,初步判断As-SesTPS1蛋白定位于细胞质中。运用predictprotein(http://www.predictprotein)预测As-SesTPS1基因编码蛋白质的二级结构,α螺旋结构(Helix)占总氨基酸的64.93%,其他结构占总氨基酸的35.07%。运用Swiss-model(http://swissmodel.expasy.org/)进行三维结构预测,用于建立该模型的氨基酸残基为3~556位,该模型以 [3g4dA]蛋白为模板,序列同源性为44.53%。运用Discovery Studio软件对As-SesTPS1基因编码的蛋白质进行三维结构建模,见图 5。
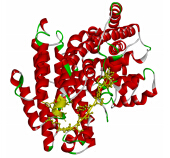 | 黄色球棍结构和棒状结构分别表示倍半萜保守结构域RRx8W和DDxxD Yellow ball & stick model and stick model represent conserved motif RRx8W and DDxxD respectively 图 5 白木香As-SesTPS1基因的预测蛋白三维结构Fig. 5 Predicted 3D structure of As-SesTPS1 protein in A. sinensis |
本课题组前期转录组测序中,分别制备了白木香不同组织部位RNA-Seq测序样品,通过Illumina HiSeqTM 2000测序平台构建了各组织的转录组测序文库[7]。本研究以获得的白木香转录组序列为参考数据库,进行基因注释,基因的表达量使用RPKM(Reads Per Kb per Million reads)法[12],根据RPKM值筛选出满足FDR≤0.001,且差异表达倍数不低于2倍(log2Ratio≥1)[13, 14]的基因作为差异表达基因。RPKM法能消除基因长度和测序量差异对计算基因表达的影响,计算得到的基因表达量可直接用于比较不同样品间的基因表达差异。本研究选择的CL5155.Contig3_B1基因即为差异表达基因,沉香样品、过渡样品、白木样品的RPKM值分别为124.44、41.73、7.14,其在不同样品中的表达量有明显差异,在沉香样品中最高。根据其扩增得到的As-SesTPS1基因利用qRT-PCR检测,结果表明,As-SesTPS1基因在沉香样品中高表达,在过渡样品中有少量表达,在白木样品中几乎没有表达。根据统计学分析,沉香样品中As-SesTPS1基因表达水平显著高于未结香部位(P<0.01),验证了As-SesTPS1基因主要在沉香部位高表达。将qRT-PCR扩增产物进行1%琼脂糖电泳检测,内参基因Histone在白木香3个不同部位样品中扩增产物亮度一致,目的基因As-SesTPS1在3个样品中扩增产物结果符合预期,只有沉香样品能扩增到目的条带,实验结果进一步验证了qRT-PCR结果的可靠性。
4 讨论沉香作为传统名贵中药,其倍半萜等特征成分大多具有一定的生物活性,克隆并研究沉香活性成分的相关合成基因,对于沉香药用价值的开发及利用具有积极的促进作用。本研究通过对白木香倍半萜合成酶As-SesTPS1基因的研究,为从基因水平探索沉香代谢的生理生化研究奠定基础。
研究表明,萜类合成酶在三维结构上有很高的同源性,由α-螺旋、短的连接环和拐角等立体结构组成,并含有2个明显的结构区域,即C末端结构区域和N末端结构区域[15]。酶的活性中心是C末端的一个疏水区域,由6个α-螺旋组成,这个疏水区域有利于底物疏水烃基部分的进入和结合[16]。N末端没有特殊的功能元件,突变分析显示,这个区域起到方便C末端催化活性区域正确折叠的脚手架作用。几乎所有的萜类合成酶都含有一个天冬氨酸富集基序(DDxxD),这个基序被认为起到结合金属离子的作用,其定位在活性位点的入口处,在引导底物催化时发挥重要的作用,若基序发生突变则会导致酶催化活性的下降或产生异常的产物[17]。本研究获得的倍半萜合成酶基因As-SesTPS1具有倍半萜合成酶的保守结构域RRx8W和DDxxD,聚类到萜类TPSa家族,经BLASTp分析发现其与多条愈创木烯型倍半萜合成酶基因具有较高相似性;经qRT-PCR验证,确定其在结香部位具有较高的表达水平,显著高于未结香部位的表达水平(P<0.01),这与测序结果相符,说明该基因编码的蛋白具有催化倍半萜合成的功能。
将本研究获得的As-SesTPS1基因与之前获得的倍半萜合成酶基因进行BLASTp相似性分析,发现它们之间存在较大差异,As-SesTPS1基因与Kumeta等[5]和Xu等[6]得到的愈创木烯合成酶基因编码的氨基酸序列相似性分别为54%、53%,与本课题组获得的As-SesTPS基因编码的氨基酸序列相似性为41%,推测As-SesTPS1基因为一种新的倍半萜合成酶基因,可能编码不同结构类型倍半萜的合成酶蛋白。本研究结果将为深入研究沉香倍半萜合成酶基因之间的共性与特征差异提供基础数据,并为今后提高白木香中倍半萜类物质量提供了理论依据。另外,本研究通过对As-SesTPS1基因在沉香基因组与cDNA的扩增结果的比对,为后期研究内含子与外显子间的关系,并从转录水平解析倍半萜合成酶基因的功能奠定基础。
| [1] | 中国科学院中国植物志编辑委员会. 中国植物志 [M]. 北京: 科学出版社, 1999. |
| [2] | 中国药典 [S]. 一部. 2010. |
| [3] | 刘军民, 徐鸿华. 国产沉香研究进展 [J]. 中药材, 2005, 28(7): 628-629. |
| [4] | 岳跃冲, 范燕萍. 植物萜类合成酶及其代谢调控的研究进展 [J]. 园艺学报, 2011, 3(2): 379-388. |
| [5] | Kumeta Y, Ito M. Characterization of δ-guaiene synthases from cultured cells of Aquilaria, responsible for the formation of the sesquiterpenes in agarwood [J]. Plant Physiol, 2010, 154(4): 1998-2007. |
| [6] | Xu Y, Zhang Z, Wang M, et al. Identification of genes related to agarwood formation: transcriptome analysis of healthy and wounded tissues of Aquilaria sinensis [J]. BMC Genom, 2013, 14(1): 227-243. |
| [7] | 吴宏清, 王 磊, 陶美华, 等. 化学诱导后白木香转录组文库的构建与测序 [J]. 生物技术通报, 2013(8): 63-67. |
| [8] | 吴宏清, 王 磊, 何 欣, 等. 白木香倍半萜合成酶基因As-SesTPS的克隆及生物信息学与表达分析 [J]. 中草药, 2014, 45(1): 94-101. |
| [9] | 吴宏清, 王 磊, 章卫民, 等. 药用植物白木香高质量总RNA提取方法的研究 [J]. 中药材, 2013, 25(7): 1055-1059. |
| [10] | Okudera Y, Ito M. Production of agarwood fragrant constituents in Aquilaria calli and cell suspension cultures [J]. Plant Biotechnol, 2009, 26(3): 307-315. |
| [11] | Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method [J]. Method, 25(4): 402-408. |
| [12] | Mortazavi A, Williams B A, McCue K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq [J]. Nature, 2008, 5(7): 621-628. |
| [13] | Audic S, Claverie J M. The significance of digital gene expression profiles [J]. Genome Res, 1997, 7(10): 986-995. |
| [14] | Benjamini Y, Yekutieli D. The control of the false discovery rate in multiple testing under dependency [J]. Ann Stat, 2001, 25(9): 1165-1188. |
| [15] | Yu F N A, Utsumi R. Diversity, regulation, and genetic manipulation of plant mono-and sesquiterpenoid biosynthesis [J]. Cell Mol Life Sci, 2009, 66(18): 3043-3052. |
| [16] | Schilmiller A L, Schauvinhold I, Larson M, et al. Monoterpenes in the glandular trichomes of tomato are synthesized from a neryl diphosphate precursor rather than geranyl diphosphate [J]. Proc Natl Acad Sci USA, 2009, 106(26): 10865-10870. |
| [17] | Degenhardt J, Köllner T G, Gershenzon J. Monoterpene and sesquiterpene synthases and the origin of terpene skeletal diversity in plants [J]. Phytochemistry, 2009, 70(15): 1621-1637. |
 2015, Vol. 46
2015, Vol. 46


