2. 湖北省果品办公室, 湖北 武汉 430070
2. The office for development and management of fruit industrialization in Hubei Province, Wuhan 430070, China
简单重复序列(simple sequence repeat,SSR)又称微卫星(microsatellite)、串联短重复序列(short tandem repeats,STR)、简单序列长度多态性(simple sequence length polymorphism,SSLP),是一种由1~6个核苷酸组成的串联重复单元,广泛分布在真核和原核生物基因组中[1]。基于PCR的SSR标记有重复性好、可靠性高、操作容易、共显性等特点,成为构建连锁遗传图谱、研究群体遗传学、进行分子标记辅助育种、系谱分析、品种指纹图谱绘制等的理想工具[1, 2]。传统的SSR(基因组SSR,genomic-SSR)标记开发周期长、成本高。而以EST-SSR为代表的genic-SSR能直接反映基因编码蛋白信息,可与基因功能直接关联,从而直接鉴定某些重要性状;此外,由于引物来自保守度较高的基因编码区,使其在不同种属间也具有通用性,在亲缘物种之间矫正连锁图谱和比较作图方面具有较强的优势[1, 2]。随着第2代测序技术的发展应用,转录组测序为genic-SSR的开发提供了较EST数据大很多的数据基础,从而加速SSR标记,特别是与重要性状关联或比较作图方面的SSR标记的开发。
茯苓Poria cocos (Schw.) Wolf是一种隶属于担子菌门Basidiomycetes,多孔菌科Polyporaceae,的高等真菌,其菌核是一种传统的中药,《中国药典》2015年版收录有茯苓与茯苓皮两味药材[3]。茯苓是一种药食两用、大宗的常用中药材,约10%的中成药(约300种)原料配方中含有茯苓[4, 5]。茯苓的主要化学成分为多糖及三萜类化合物,具有抗肿瘤、抗氧化、抗惊厥等多种生物活性[6]。茯苓主要分布于我国中部及西南各省,具有悠久的人工栽培历史,但由于很难获得其有性态担孢子,生产上育种处于长期无性繁殖,导致菌种退化;且由于菌种管理不规范,菌种名称较为混乱[7, 8]。
茯苓的SSR分子标记研究尚未见文献报道,本研究通过对已获得的茯苓转录组序列进行SSR搜索,并对找出的SSR序列进行分析,同时注释其所在序列的基因功能来了解茯苓转录组序列所含SSR的序列特征及组成情况,推测其可能相关联的性状。此外,本研究还利用公开的茯苓基因组数据进行比较研究,得到转录组SSR与基因组SSR的分布与序列特征的异同。本研究可为茯苓的遗传多样性、良种选育、SSR分子标记,特别是与性状连锁基因的分子标记的开发等提供研究基础。
1 数据来源样品于2012年采自湖北省英山县石头咀镇的栽培地,由湖北省中医药研究院王克勤研究员鉴定为茯苓Poria cocos (Schw.) Wolf菌核。根据Shu等[9]报道,将菌核接种到PDA培养基上,进一步分离纯化得到纯茯苓菌种。分别提取茯苓菌核、菌丝RNA,利用Illumina HiSeq 2000进行转录组测序,测序结果使用Trinity软件进行从头组装,得到共含35 299 182核苷酸的Unigene 41 327条[9]。从JGI网站(http://genome.jgi-psf.org/Wolco1/ Wolco1.home.html)[10]下载茯苓的基因组数据作为对比参考数据,数据含348个scaffold,共50 483 556核苷酸。该基因组测序使用的是1956年分离自美国佛罗里达州阿拉楚瓦郡的茯苓单孢培养菌株MD-104 SS10。
2 方法 2.1 简单重复序列SSR搜索分析在Ubuntu 14.04 LTS(32位)操作系统中使用MISA软件(MIcro-SAtellite identification tool,MISA,http://pgrc.ipk-gatersleben.de/misa/misa.html)对转录组Unigene及基因组scaffold序列进行SSR位点搜索。设置单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸最少重复次数分别为15、6、5、4、3、3。复合SSR 2个位点间最大间隔碱基数为100。将生成的文本文件导入到Excel(Microsoft Office Excel 2013)中,对SSR的基本信息进行统计分析。
2.2 茯苓转录组数据中含SSR的Unigene的GO分类对茯苓转录组数据中1 887条含有SSR的Unigene序列使用BlastX比对到蛋白质数据库nr(GenBank non-redundant),比对参数e值<10−5。根据nr蛋白注释信息,使用Blast2GO软件[11]分析得到转录组数据中含SSR的Unigene的基因本体(gene ontology,GO)注释信息。得到每个Unigene的GO注释后,利用WEGO在线软件(http://wego.genomics.org.cn/cgi-bin/wego/index.pl)[12]对注释的Unigene做GO功能分类统计,分析含SSR的Unigene的功能分布特征。
2.3 茯苓转录组数据中含SSR的Unigene的KEGG代谢通路分析通过BlastX,将含SSR的Unigene序列比对到京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)数据库中,比对参数e值<10−5。比对得到Unigene在KEGG数据库的注释信息,及其在KEGG orthology(KO)系统中的相应K编号。根据K编号将Unigene注释到相应的代谢通路上,并进行代谢通路分析。
2.4 数据统计分析使用SPSS 19.0.0软件对数据进行χ2检验。
3 结果与分析 3.1 茯苓转录组及基因组SSR的分布本研究利用Perl脚本MISA(MIcro-SAtellite identification tool,MISA,http://pgrc.ipk-gatersleben.de/misa/misa.html)搜索茯苓转录组及其基因组序列的SSR,设置的SSR最小长度均不小于12 bp。从总长度约为35.30 Mb的41 327条转录组Unigene序列中搜索到2 197个SSR,分布于1 887条Unigene上;其中含多个SSR的Unigene占14.47%,共273条。从对比的总长度50.38 Mb的348条基因组scaffold序列中搜索到SSR 2 877个,多于从转录组数据搜索到的。然而,转录组与基因组中搜索到的SSR平均密度分别为58.78个/Mb、54.00个/Mb,转录组SSR密度略高于基因组。经统计计算,转录组SSR与基因组SSR的平均长度无显著差异,平均长度分别为19.59、20.74 bp。见表 1。
|
|
表 1 茯苓转录组和基因组序列中SSR统计 Table 1 Description of SSR in transcriptome and genome sequences of P. cocos |
在转录组与基因组序列中搜索到的SSR中,分别含有5.40%和4.95%的复合SSR,SPSS χ2检验显示无差异(表 1),说明搜索到的转录组与基因组SSR具有一定的一致性。此外,SSR重复基元(考虑序列互补,且包含复合SSR的重复基元)按核苷酸数分类后,各类型SSR重复基元的基元种类数在茯苓转录组和基因组中亦无显著性差异;但各类型含有的SSR数量及频率却有显著差异(表 2)。在转录组SSR中,核苷酸重复的基元共有241种,少于从基因组数据得到的292种,而且,在转录组SSR中六核苷酸重复基元最多,占35.64%,其次是三核苷酸,占31.04%;而在基因组SSR中则分别是五核苷酸重复基元(32.15%)、六核苷酸基元(25.72%)。转录组SSR及基因组SSR中单核苷酸及四核苷酸重复基元均相对较少,且四核苷酸重复基元的频率无差异(P>0.05);单核苷酸重复在基因组SSR只有C/G型,重复长度(21 bp)小于转录组SSR(24 bp)(表 3)。但是,按单一重复基元的数量上看,转录组及基因组SSR均为二核苷酸重复最多,分别为(CG/CG)n和 (AG/CT)n。转录组SSR 2~6核苷酸类型重复基元的最高重复数均小于基因组SSR。转录组SSR以单核苷酸C/G重复次数最多,为24次;但以重复14次的ACCACG/CGTGGT六核苷酸的SSR最长,为84 bp。而基因组SSR以AC/GT二核苷酸重复44次为最多,以六核苷酸重复ACCTGC/AGGTGC重复26次,达156 bp最长。转录组SSR与基因组SSR比较,各类型重复基元中数量最多的重复基元只有单核苷酸、三核苷酸及四核苷酸相同,分别为C/G、CCG/CGG、ACGC/CGTG;二核苷酸、五核苷酸及六核苷酸的最多重复基元均不同。此外,在转录组SSR与基因组SSR中,除单核苷酸外,2~6核苷酸的最多重复基元均不是最长的重复基元(表 3)。
|
|
表 2 茯苓SSR重复基元出现频率 Table 2 Occurrence frequency of different SSR motifs of P. cocos |
|
|
表 3 重复基元序列特征 Table 3 Sequence features of different SSR motifs of P. cocos |
通过MISA搜索,在茯苓转录组序列中确定出1 887条Unigene含有SSR,为了解茯苓中含SSR基因的功能,本研究对含SSR的Unigene进行了GO分类注释。GO将基因功能共分为3个本体,即分子功能(molecular function)、细胞组分(cellular component)、生物进程(biological process),其下又分为了很多亚类,各类间互相关联,从不同角度对基因的功能进行分类注释。通过对含SSR的茯苓Unigene进行GO注释,可以全面描述茯苓中含SSR基因和基因产物的属性。
将搜索到含有SSR的Unigene序列使用BlastX比对到nr蛋白数据库,取比对最高的为序列的注释信息。其中1 561条Unigene注释得到了注释信息,326条无注释结果。再使用Blast2GO软件,进行含SSR的Unigene的GO注释,取得其GO分类号,1 887条Unigene中只有115个Unigene得到了相应的GO分类号,为6.09%,其他1 772个不能成功注释。将含有SSR序列的1 887条Unigene编号及其对应的GO号使用在线工具WEGO(http://wego. genomics.org.cn/cgi-bin/wego/index.pl)进行分类,得到其基因功能分布(图 1)。
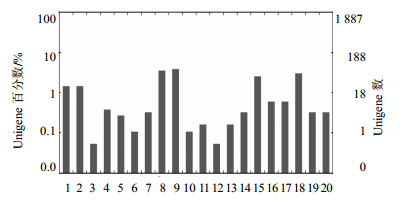
|
1-细胞 2-细胞成分 3-细胞被膜 4-大分子复合物 5-细胞器6-细胞
器组分 7-抗氧化 8-结合活性 9-催化活性 10-分子信息传递
11-结构分子 12-翻译调节 13-转运 14-生物调节 15-细胞进程 16-建立定位 17-定位 18-代谢进程 19-色素沉积 20-对刺激的响应
1~6-细胞组分 7~13-分子功能 14~20-生物进程 1-cell 2-cell part 3-envelope 4-macromolecular complex 5-organelle 6-binding 7-anti-oxidant 8-catalytic 9-hydrolase 10-molecular transducer 11-nucleotide binding 12-structural molecule 13-transferase 14-translation regulator 15-transporter 16-biological regulation 17-cellular metabolic process 18-cellular process 19-localization 20-macromolecule metabolic process 1—6-cell part 7—13-molecular function 14—20-biological process 图 1 茯苓转录组中含SSR的Unigene的GO分类 Fig.1 GO classification of Unigenes containing SSR in transcriptome of P. cocos |
结果表明,在有GO注释的115个Unigene中,有107条(93.04%)被注释到分子功能类中,而生物进程及细胞组分类分别有74条(64.35%)和30条(26.09%)。在107条被注释到分子功能类的Unigene中,分别有74条、68条被注释为催化活性(catalytic activity)和结合活性(binding),为有GO注释Unigene的64.35%、59.13%,其中多被注释为转移酶活性(transferase activity)、水解酶活性(hydrolase activity)、核苷酸结合(nucleotidebinding)。而在生物进程类中,以代谢进程(metabolicprocess)及细胞进程(cellular process)下的细胞代谢进程(cellular metabolic process)为主,有43条Unigene,占注释为该类的58.11%。同时,细胞组分类中几乎全注释(28/30,93.33%)为细胞组分(cell part)。综合可知,已被注释出的含有SSR的Unigene主要的功能是与细胞的基础代谢相关的。
3.4 茯苓转录组数据中含SSR的Unigene的KEGG代谢通路分析由于Unigene的GO注释只有较少比例被注释出来,为了更加全面地了解茯苓转录组序列含有SSR的Unigene的功能,将其注释到KEGG数据库中,分析得到其在茯苓代谢中的通路富集情况。注释KEGG代谢通路时,会给每一个功能的基因给定一个K编号(K numbers),并注释到具有相应归类的代谢通路(pathway)中。
在对茯苓转录组1 887条含SSR的Unigene进行KEGG代谢通路分析时,有1 223条(64.81%)Unigene有注释结果,另外的664条(35.19%)未得到注释。统计被注释的Unigene发现,只得到了412个K编号,即说明Unigene中有许多相互间功能相同,平均达每2.97个Unigene具有相同的功能。当将有K编号注释的Unigene注释到KEGG代谢通路图中时,只有752个Unigene,对应245个K编号被注释到219个通路图(map)中;有471个Unigene,167个K编号未被注释到通路图中。最终,相当于只有39.85%含SSR的Unigene被注释到KEGG代谢通路中。
在对注释到的219个通路图进行分析时,利用KEGG数据库的分类,将其归类到全部7大类代谢通路中,其中被注释到新陈代谢(metabolism)及遗传信息处理(genetic information processing)类的Unigene最多,分别为314、297个,占全部含SSR的Unigene的16.64%及15.94%,占能注释到通路的Unigene的41.76%、39.49%(图 2)。
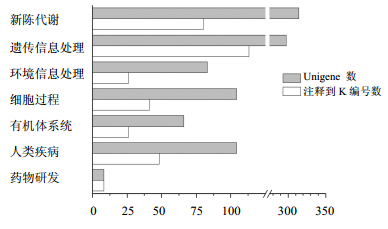
|
图 2茯苓转录组中含SSR的Unigene的KEGG代谢通路分类 Fig.2 KEGG pathway classification of Unigenes containing SSR in transcriptome ofP.cocos |
对注释到新陈代谢通路类的Unigene作进一步的分类分析,其能注释到13个亚类中的除化学结构转化图(chemical structure transformation maps)类外的其他12个亚类(图 3)。且除总览图(global and overview maps)类外,注释到碳水化合物代谢(carbohydrate metabolism)类通路的Unigene最多,有176个Unigene,占注释到新陈代谢类的56.05%;其次是注释到氨基酸代谢(amino acid metabolism)类、脂类代谢(lipid metabolism)类,这与GO注释得出的基础代谢的结果相一致。
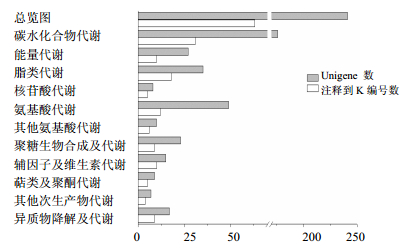
|
图 3 茯苓转录组中注释到新陈代谢通路的含SSR的Unigene的通路分析 Fig.3 Analysis on Unigenes containing SSR in transcriptome of P.cocos annotated into metabolism pathway |
本研究对茯苓菌核、菌丝两个样本高通量测序组装后的Unigene序列进行SSR分布及序列特征分析,从41 327条Unigene中找到2 197个SSR,分布在1 887条Unigene中,包含SSR的序列出现频率为4.57%;比报道的多孔菌目EST-SSR的9.80%出现频率低,但比同时报道的多孔菌科EST-SSR 1.14%频率高。另外,茯苓转录组SSR密度为58.78个/Mb,即平均相隔17.01 kb出现1个SSR序列,比从基因组中找出SSR的密度略高;比报道多孔菌目的149.81个/Mb少,比多孔菌科平均34.81 个/kb多[13]。这种差异,一方面与物种本身SSR数量差异有关,还与原始序列数量、长度,以及搜索SSR时长度设定最低标准不同有关。本研究与陈媛媛[16]报道的EST-SSR同时在这3个方面都不同。
同时,由于上述因素的影响,也形成了SSR的序列特征的差异。本研究发现茯苓转录组SSR数量最丰富的是六核苷酸重复基元,其次是三核苷酸基元;这与一般认为的二核苷酸为最、三核苷酸次之有所不同。在茯苓基因组SSR中也同样出现五核苷酸、六核苷酸重复基元较丰富的情况,由于参数设定不同,这与基因组报道的不同[10]。此外,茯苓单核苷酸、三核苷酸、四核苷酸重复基元数量最多的C/G、CCG/CGG、ACGC/CGTG重复基元与从基因组搜索到的最多的基元相同。从单一重复基元计,转录组、基因组中重复基元数量最多的均为二核苷酸,分别为 (CG/CG)n、(AG/CT)n,占总SSR数的10.97%及12.03%。
4.3 茯苓转录组SSR的功能关联通过对茯苓转录组中含SSR的Unigene进行GO分类发现其主要归类于生物进程类下的细胞代谢进程、分子功能类下的核苷酸结合及细胞组分类下的细胞组分类。另外,通过KEGG代谢通路注释,Unigene多为新陈代谢及遗传信息处理功能,且在新陈代谢类通路中主要分布于碳水化合物代谢、氨基酸代谢等通路中。综合GO分类及KEGG代谢通路注释结果可推知,茯苓转录组中含SSR的Unigene主要为生物体的基础代谢相关的功能。但在注释GO分类及KEGG代谢通路时出现了多个Unigene共同注释到相同功能上,这种情况的出现除了正常的基因家族等多个基因行使相同功能外,也可能与转录本在加工时的可变剪接、软件拼接错误等有关。因此,对转录组SSR的应用还需要进行相应的引物筛选等工作,同时,可有针对性地选择与一定功能相关的基因作为SSR标记位点,从而利于目标性状的筛选。
| [1] | Kalia R K, Rai M K, Kalia S, et al. Microsatellite markers: an overview of the recent progress in plants [J]. Euphytica, 2011, 177(3): 309-334. |
| [2] | 常 玮, 赵 雪, 李 侠, 等. 大豆EST-SSR标记开发及与Genomic-SSR的比较研究 [J]. 中国油料作物学报, 2009, 31(2): 149-156. |
| [3] | 中国药典 [S]. 一部. 2015. |
| [4] | Wang W, Dong H, Yan R, et al. Comparative study of lanostane-type triterpene acids in different parts of Poria cocos (Schw.) Wolf by UHPLC-Fourier transform MS and UHPLC-triple quadruple MS [J]. J Pharm Biomed Anal, 2015, 102: 203-214. |
| [5] | Xu Z Y, Tang W R, Xiong B, et al. Effect of revulsive cultivation on the yield and quality of newly formed sclerotia in medicinal Wolfiporia cocos [J]. J Nat Med, 2014, 68(3): 576-585. |
| [6] | Wang Y Z, Zhang J, Zhao Y L, et al. Mycology, cultivation, traditional uses, phytochemistry and pharmacology of Wolfiporia cocos (Schwein.) Ryvarden et Gilb.: A review [J]. J Ethnopharmacol, 2013, 147(2): 265-276. |
| [7] | 熊 杰, 林芳灿, 王克勤, 等. 茯苓基本生物学特性研究 [J]. 菌物学报, 2006, 25(3): 446-453. |
| [8] | 蔡志欣, 蔡丹凤, 陈美元, 等. 32个茯苓菌株的SRAP分析 [J]. 食药用菌, 2013, 21(2): 96-98. |
| [9] | Shu S H, Chen B, Zhou M C, et al. De novo sequencing and transcriptome analysis of Wolfiporia cocos to reveal genes related to biosynthesis of triterpenoids [J]. PLoS One, 2013, 8(8): e71350. |
| [10] | Floudas D, Binder M, Riley R, et al. The Paleozoic origin of enzymatic lignin decomposition reconstructed from 31 fungal genomes [J]. Science, 2012, 336(6089): 1715-1719. |
| [11] | Conesa A, Gotz S, Garcia-Gomez J M, et al. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research [J]. Bioinformatics, 2005, 21(18): 3674-3676. |
| [12] | Ye J, Fang L, Zheng H, et al. WEGO: a web tool for plotting GO annotations [J]. Nucleic Acids Res, 2006, 34 (Web Server issue): W293-297. |
| [13] | 李小白, 向 林, 罗 洁, 等. 转录组测序 (RNA-seq) 策略及其数据在分子标记开发上的应用 [J]. 中国细胞生物学学报, 2013, 35(5): 720-726. |
| [14] | 蔡丹凤, 陈美元, 郭仲杰, 等. 茯苓栽培菌株的RAPD分析 [J]. 中国农学通报, 2010, 26(20): 57-60. |
| [15] | 谢贤安, 汪思迪, 曾晓丽, 等. 茯苓菌属遗传多样性的ISSR分析 [J]. 湖北农业科学, 2008, 40(10): 1111-1113. |
| [16] | 陈媛媛. 猪苓EST-SSR标记的开发及遗传多样性研究 [D]. 杨凌: 西北农林科技大学, 2014. |
 2015, Vol. 46
2015, Vol. 46


