2. 生物能源持续开发利用教育部工程研究中心, 云南 昆明 650500;
3. 云南师范大学-台湾新竹清华遗传-环境互作联合实验室, 云南 昆明 650500
2. Engineering Research Center of Biological Energy Development and Utilization, Ministry of Education, Kunming 650500, China;
3. Join-Laboratory of YNNU-NTHU for Genetic & Environment Interaction, Kunming 650500, China
灯盏花又名灯盏细辛,为菊科(Compositae)飞蓬属Erigeron L. 植物短葶飞蓬Erigeron breviscapus (Vant.) Hand. -Mazz.,广泛分布于我国西南的高山、亚高山地区,尤以云南较多,资源量占全国95%以上[1]。云南的苗、壮、白、彝、傈僳等民族均有利用该植物的历史,以全草入药,用于感冒头痛、牙痛、胃痛、风湿疼痛、脑血管性头痛、瘫痪、骨髓炎、慢性气管炎等[2]。灯盏花临床上主要用于治疗高血压、脑栓塞、冠心病、心绞痛、心肌缺血等[3],被列为我国治疗心血管疾病临床用药的基本药物。近年来,由于生境恶化,外加野生资源的过度采挖,导致野生资源面临枯竭,虽人工栽培已解决灯盏花的资源供应瓶颈。但鉴于对灯盏花遗传学背景了解的匮乏,导致栽培灯盏花在育种方面滞后,使得栽培灯盏花在有效组分量、产量、抗性上极不稳定。对其遗传背景的了解,不仅有助于阐述灯盏花药材道地性的形成机制,也有助于资源的评价及分子辅助育种。基于二代测序技术的转录组测序,由于具高通量、精确性高、低成本、高敏感度等优点,可不需了解其全基因组背景下,通过测序结果与模式植物的数据库比对,掌握非模式生物基因组的基本信息及其表达状况,目前已成为转录本结构、转录本结构变异、基因表达水平、非编码区域功能、低丰度全新转录本,甚至是基因调控网络构建的重要手段[4, 5, 6, 7, 8, 9]。在模式生物酿酒酵母Saccharomyces cerevisiae[10]、裂殖酵母Fission yeast[11]、拟南芥Arabidopsis thaliana L.[12]、小鼠[13]、人[14]中广泛运用,在非模式生物的药用植物中,目前在银杏Ginkgo biloba L.[15]、虎杖Polygonum cuspidatum Sieb. et Zucc.[16]、罗汉果Siraitia grosvenorii (Swingle) C. Jeffrey ex A. M. Lu & Zhi Y. Zhang[17]、铁皮石斛Dmdrobium officinale Kimura et Migo和人参Panax ginseng C. A. Meyer[18]等也用于有效成分生物合成途径的研究。灯盏花的黄酮类及咖啡酸酯量与土壤中N、P、K量具有明显的相关性,与全N量呈显著的负相关[19, 20]。本研究在对低氮胁迫的灯盏花全植转录组测序文库构建的基础上,利用Illumina HiSeqTM 2000测序平台株进行转录组文库测序,以期获得灯盏花的低氮胁迫响应机制,发掘与黄酮类化合物合成途径相关的基因,为下一步灯盏花分子遗传图的构建及分子辅助育种奠定基础。
1 材料与方法 1.1 材料及处理材料为云南师范大学-台湾新竹-清华遗传环境互作联合实验室培养的无菌苗,经笔者鉴定为短葶飞蓬Erigeron breviscapus (Vant.) Hand. -Mazz. 的幼苗(原始材料采集于云南丘北,实验室编号为E6)。以MS培养基为基本培养基,培养条件:25 ℃、2 000 lx、光周期16 h光照/8 h黑暗。实验中,首先将无菌丛生苗转入生根培养基进行培养,18~20 d后,当丛生苗长出约1 cm的根时,选取长势相近的灯盏花生根苗,分别转入正常对照组(CK)和低氮处理组(LN)培养基中进行培养,培养条件与无菌苗培养相同。CK培养基N素量为60 mmol/L,LN培养基的N量降为CK的75%(45 mmol/L),其他条件相同。培养14 d后,分别随机从CK和LN培养瓶中选取植株各15株,鲜质量约500 mg,液氮速冻后于−80 ℃冰箱保存备用。
1.2 灯盏花总RNA的提取采用Piotr Chomczynski和Nicoletta Sacchi改良的异硫氰酸胍CTAB法提取灯盏花样品总RNA[21]。Agilent 2100生物分析仪对总RNA完整性计数(RIN)值及28 S/18 S值进行检测,验证RNA的完整性。
1.3 cDNA文库的制备检验合格的RNA用于构建cDNA文库,使用Illumina TruSeq RNA sample pre Kit试剂盒及其推荐的配套试剂。具体流程如下:(1)mRNA的富集纯化:用带有寡核苷酸Oligo-(dT)的磁珠富集mRNA。磁珠与mRNA 3’端poly(A)在结合缓冲液中相互结合,用加热的10mmol/L Tris-HCl Buffer洗脱,得到纯化的mRNA。(2)mRNA的片段化:纯化的mRNA加入片段化缓冲液,94 ℃处理5 min可将mRNA打断成短片段,然后用乙醇沉淀法回收产物。(3)cDNA第1链的合成:以短片段mRNA为模板,加入第1链合成反应体系,即缓冲液2 µL、RNase抑制剂0.25 µL、反转录酶1 µL、1µl dNTPs (10mmol/L)和1 µL六碱基随机引物 (12 µmol/L)。混合均匀后室温静置10 min,42 ℃水浴反应1 h,反应结束后置于冰上2 min,即合成cDNA的第1链。(4)cDNA第2链的合成:cDNA第1链的合成反应结束后,在其体系中再加入第2链合成体系,即缓冲液5 µL、dNTPs (10mmol/L) 2 µL、DNA聚合酶I 100U、RNase H 50U和DNA连接酶60U。混合均匀后16 ℃反应3 h,纯化合成产物。(5)末端修复:用T4 DNA Polymerase、Klenow DNA Polymerase和T4 PNK对合成的cDNA 3’末端补平、5’端进行磷酸化修饰。之后对产物进行纯化。(6)cDNA 3’加dA:用Klenow Exo-给经过末端修饰的cDNA 3’末端加入1个腺苷酸,以便与接头连接。(7)接头连接:用T4 DNA Ligase将经过磷酸化和腺苷酸化的cDNA片段与接头进行连接。纯化产物,并用琼脂糖凝胶电泳和胶回收,收集(200±25) bp的cDNA片段。所用接头序列信息如下:TruSeq Universal Adapter(5’端接头):5’-3’AATGATACGG-CGACCACCGAGATCTACACTCTTTCCC TACAC-GACGCTCTTCCGA TCT;TruSeq Adapter(3’端接头,带下划线的6 bp碱基为随机序列Index,共有24种Index):5’-3’GATCGGAAGAGCACACGTCT-GAACTCCAGTCACNNNNNNATCTCGTATGCCGTCTTCTGCTTG。PCR扩增:用与接头相对应的引物对回收的(200±25)bp cDNA片段进行PCR扩增,回收产物并纯化,cDNA文库制备完成。构建好的文库用Illumina HiSeqTM 2000测序平台进行测序。
1.4 原始数据过滤测序仪测序得到的原始图像数据经碱基读出转化为序列数据,并以Fasta格式记录序列及其碱基质量信息,每个读出碱基的测序质量值(Qphred)范围为2~35。Qphred与测序错误率(e)有如下关系:Qphred=−10 lg e,即测序质量值10、20、30分别对应着测序错误率10%、1%和0.1%。原始数据中的测序读长(raw reads)中包含有很多带接头、低质量的 [Qphred≤5的碱基数占整个测序读长(reads)超过50%] 和含有N(不确定碱基)的比例超过10%的reads。为了保证信息分析质量,必须对测序读长过滤,过滤之后的序列称为有效读长(clean reads),后续分析都基于clean reads。
1.5 转录组de novo组装灯盏花及其近属植物目前都还没有基因组测序信息,所以本研究采用无参考基因组的转录组分析方法。使用短序列拼接软件Trinity(v2012-10-05)对测序数据集(reads set)进行de novo组装[22]。Trinity的拼接是基于reads之间具有一定长度的重叠区域,reads的初步拼接产物称为Contig。相同的原理,将Contig通过重叠区域进一步组装得到更长的序列,直至所得序列两端无法再延长。Trinity组装的结果称为转录本(transcript),一个转录本即可看作是一个广泛意义上的基因。组装过程中,可能会产生具有主要路径相同、局部分支不同的结果,转录本序列号中用comp_*为具有相同主干的一组序列编号,用seq1、seq 2等表示不同的分支序列。挑选每组中最长的转录本作为该组基因的拼接基因(Unigene)(图 1)。
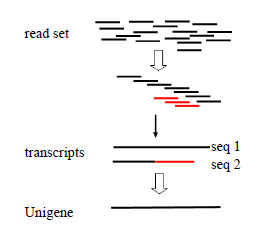 | 图 1 de novo组装流程图Fig.1 Flow chart of de novo assembling |
以e值<1×10−5为阈值,将Unigene序列与Nr(NCBI non-redundant protein sequences)、Nt(NCBI nucleotide sequences)、Swiss-Prot、KOG(euKaryotic Ortholog Groups,真核生物直系同源蛋白聚簇)、KO(KEGG Ortholog database,Kyoto Encyclopedia of Genes and Genomes,京都基因与基因组百科全书)等数据库做Blast比对,用Hmmscan在Pfam(Protein family)数据库和用Blast2GO在GO(Gene Ontology)数据库中也做同样的比对,得到该Unigene在相应数据库的蛋白功能及结构注释信息。如果不同库之间的比对结果有矛盾,按Nr、Swiss-Prot、KO及COG的优先级顺序确定Unigene的比对结果。跟以上库皆比对不上的Unigene用软件ESTScan预测其编码区并确定序列的方向。
2 结果 2.1 灯盏花植株总RNA的获得使用改良异硫氰酸胍-CTAB法提取灯盏花植株总RNA,经Agilent 2100生物分析仪检测,CK及LN样品总RNA分别为505.67、672.42 ng/μL,而其相应RIN值分别为7.3、6.8,28 S/18 S值分别为1.3、1.2,28 S/18 S值均大于1.0,RNA总量远大于20 μg,满足转录组测序需求,可用于下一步测序文库的构建。
2.2 测序数据输出通过Illumina HiSeqTM 2000进行高通量测序,结果见表 1。从灯盏花LN和CK样本中分别获得35、869、305个和25、823、354个raw reads,平均读长100 bp。碱基质量分值达到20分的碱基比例均超过98%,分值达到30分的碱基均超过94%。灯盏花LN和CK样本GC量分别为43.42%和43.59%。
| 表 1 灯盏花转录组Illumina测序数据统计 Table 1 Sequencing data of transcriptome via Illumina sequencing in E. breviscapus |
用组装软件Trinity对全部clean reads做de novo组装,共得到226 401条转录本,平均读长1 058 bp,N50(将转录本按长度从大到小排列,累加转录本长度,当总长达到总碱基数的50%时的转录本长度)为1 644 bp,有142 919条(63.13%)转录本读长超过500 bp,有88 914条(39.27%)读长超过1 000 bp。挑选每组(具有相同comp序列号)转录本中最长的转录本作为该组基因的Unigene,总共得到101 156条Unigene,平均读长768 bp,N50为1 290 bp。其中有44 908条(44.39%)Unigene读长超过500 bp,有24 185条(23.91%)Unigene读长超过1 000 bp。转录本和Unigene的长度频数分布见图 2。
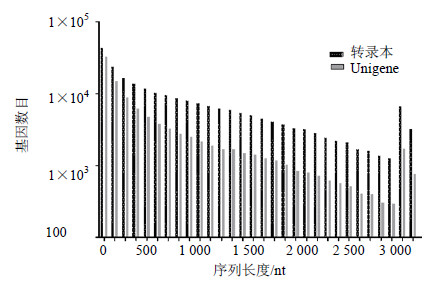 | 图 2 组装所得转录本和Unigene长度频数分布 Fig.2 Length frequency distribution of transcript and Unigene after assembling in E. breviscapus |
将用于组装的clean reads定位到组装结果转录本上(图 3),发现reads在转录本上的覆盖度分布特点为距离转录本的5’端和3’端越近,平均测序深度越低,总体分布较为均匀,总测序数据的组装效率较高。
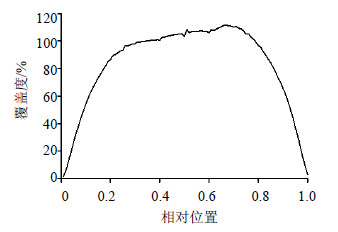 | 图 3 reads在组装转录本上覆盖度的分布 Fig.3 Overlap distribution of reads in assembled transcript in E.breviscapus |
通过将组装获得的Unigene与各公共数据库进行比对,结果见表 2。将组装和筛选得到的Unigene序列与Nr、Nt、Swiss-Prot、COG和KO等数据库进行同源性比对,以e值<1×10−5为阈值,得到与Nr、Nt、Swiss-Prot的比对结果,以及GO、COG、KEGG Pathway的功能注释。另外,通过Hmmscan搜索Pfam得到Unigene的功能域及蛋白家族注释信息。经过序列比对,101 156条Unigene中总共有59 538条(58.86%)在公共数据库中寻得相似序列。其中,Nr、Nt与Swiss-Prot中大多数Unigene有多于1个的比对结果(最多10个)。Nr中总共得到512010条相似序列,与所比对的Unigene同源性≥80%的有81 567条(15.94%),同源性在80%~60%的有221 326条(43.23%)。Swiss-Prot中总共得到306015条相似序列,其中,与所比对的Unigene同源性≥80%的有21 738条(7.11%),80%~60%的有55 588条(18.17%)。对Nt因为是直接进行核酸序列比对,同源性要求较高,得到的比对结果同源性全部大于69%,在90 776条相似序列中,同源性≥90%的有9 065条(9.99%),80%~90%的有34 665条(38.19%)(图 4)。Nr数据库的注释结果中,比对到葡萄Vitisv vinifera L. 和大豆Glycine max Merrill. 最多,同为65 536条(12.80%)。其次为三角叶扬Populus trichocarpa Torr. 和拟南芥Arabidopsis thaliana (L.) Heynbh,分别有61 983条(12.11%)和47 561条(9.29%)。种属的注释见表 3。
| 表 2 灯盏花转录组中Unigene在公关数据库的功能注释 Table 2 Functional annotation of Unigene in E.breviscapus transcriptome in public protein databases |
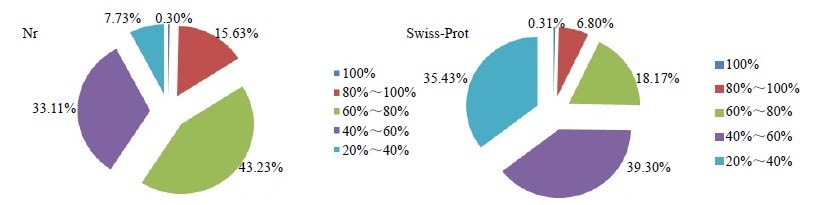 | 图 4 在Nr和Swiss-Prot中的同源性比对结果Fig.4 Homologous comparison between Nr and Swiss-Prot dadabase |
| 表 3 Nr注释中的物种分布 Table 3 Species distribution of Unigene annotated from Nr dadabase |
通过Nr、Swiss-Prot、GO、COG、KO等数据库的注释信息,在本研究构建的灯盏花转录组数据库中获得了参与黄酮类生物合成途径的多个酶,包括PAL、C4H、CHS、CHI、F3H、F3′H、ANS等(表 4)。通过与公共数据库比对获得的注释信息,从中获得了类黄酮生物合成途径中的主要基因,包括PAL、C4H、CHS、CHI、F3H、F3′H、ANS等(表 4)。
| 表 4 灯盏花类黄酮合成相关酶基因 Table 4 Related enzyme gene to flavonoid biosynthesis in E. breviscapus |
GC量是衡量测序质量可信度的一个指标,本实验GC量在(50±10)%内说明样本测序质量较好。比较转录本和筛选之后的Unigene的序列读长分布,发现被筛选掉的序列数量与序列读长呈正比,这可能是因为组装过程中较长的序列是经过了较为复杂的组装路径,这其中产生了较多的分支,而最终筛选Unigene只挑选最长的序列,故而其他分支的结果就都被略去了。本研究中对灯盏花转录组测序序列组装的结果质量较高,获得了读长较长的转录本和Unigene。序列长度通常与比对结果呈正相关,研究获得了较长的序列组装结果,为之后的数据库比对奠定了良好的基础。
3.2 影响灯盏花黄酮类生物合成相关基因植物体内合成类黄酮的种类和数量除了与黄酮类合成途径、黄酮和黄酮醇合成途径、异黄酮合成途径直接相关外,还与其上游的苯丙烷合成途径及下游的类黄酮化合物在植株体内的合成种类和数量有关,受PAL、F3H、F3′H等酶相对活性的调控[23, 24]。通过转录组文库测序可以发掘到大量涉及类黄酮合成的相关基因,如PAL、C4H、CHS、CHI、F3H、F3′H、ANS等,可为下一步研究灯盏花的低氮胁迫、功能基因发掘及分子辅助育种奠定基础。
本研究以无菌培养的灯盏花组培苗为材料,经75%低氮胁迫处理和正常对照处理,提取总RNA,构建文库,并通过Illumina HiSeqTM 2000测序平台进行转录组高通量测序,对数据进行质量评估,进而挖掘到影响灯盏花类黄酮生物合成的主要相关基因。为下一步进行灯盏花转录组及其与氮元素的互作规律研究奠定了良好的基础,同时为下一步灯盏花遗传环境互作研究及分子辅助育种奠定基础。以期能够通过这些基础,在转基因作物行不通的情况下,培育出高产、含药用成分高的种质资源。
| [1] | 杨生超, 文国松, 刘雪玲, 等. 灯盏花种质资源遗传关系的ISSR分析 [J]. 中草药, 2010, 41(9): 1523-1527. |
| [2] | 刘 宏, 杨祥良, 徐辉碧. 灯盏花的研究进展 [J]. 中草药, 2002, 33(6): 89-91. |
| [3] | 林 雄, 褚克丹. 灯盏花素的药理学研究进展 [J]. 海峡药学, 2006, 17(6): 5-8. |
| [4] | Mortazavi A, Williams B A, McCue K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq [J]. Nat Methods, 2008, 5(7): 621-628. |
| [5] | Zhang G J, Guo G W, Hu X D, et al. Deep RNA sequencing at single base-pair resolution reveals high complexity of the rice transcriptome [J]. Genome Res, 2010, 20(5): 646-654. |
| [6] | Lu T T, Lu G J, Fan D L, et al. Function annotation of the rice transcriptome at single-nucleotide resolution by RNA-seq [J]. Genome Res, 2010, 20(9): 1238-1249. |
| [7] | Filichkin S A, Priest H D, Givan S A, et al. Genome-wide mapping of alternative splicing in Arabidopsis thaliana [J]. Genome Res, 2010, 20(1): 45-58. |
| [8] | Shah S P, Morin R D, Khattra J, et al. Mutational evolution in a lobular breast tumour profiled at single nucleotide resolution [J]. Nature, 2009, 461(7265): 809-813. |
| [9] | Ponting C P, Oliver P L, Reik W. Evolution and functions of long noncoding [J]. RNA Cell, 2009, 136(4): 629-641. |
| [10] | Yao Y Y, Guo G G, Ni Z F, et al. Cloning and characterization of microRNAs from wheat (Triticum aestivum L.) [J]. Genome Biol, 2007, 8(6): R96. |
| [11] | Nagalakshmi U, Wang Z, Waern K, et al. The transcriptional landscape of the yeast genome defined by RNA sequencing [J]. Science, 2008, 320(5881): 1344-1349. |
| [12] | Wilhelm B T, Marguerat S, Watt S, et al. Dynamic repertoire of a eukaryotic transcriptome surveyed at single-nucleotide resolution [J]. Nature, 2008, 453(7199): 1239-1243. |
| [13] | Lister R, O'Malley R C, Tonti-Filippini J, et al. Highly integrated single-base resolution maps of the epigenome in Arabidopsis [J]. Cell, 2008, 133(3): 523-536. |
| [14] | Mortazavi A, Williams BA, McCue K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq [J]. Nat Methods, 2008, 5(7): 621-628. |
| [15] | Wang E T, Sandberg R, Luo S, et al. Alternative isoform regulation in human tissue transcriptomes [J]. Nature, 2008, 456(7221): 470-476. |
| [16] | 林小涵. 银杏和木兰属植物的转录组; 叶绿体基因组及其相关研究 [D]. 北京: 中国医学科学院北京协和医学院, 2011. |
| [17] | 郝大程, 马 培, 穆 军, 等. 中药植物虎杖根的高通量转录组测序及转录组特性分析 [J]. 中国科学: 生命科学, 2012, 42(5): 398-412. |
| [18] | 唐 其. 罗汉果转录组, 表达谱的高通量测序及甜苷 V 生物合成关键酶的克隆 [D]. 北京: 北京协和医学院, 2010. |
| [19] | 郭 溆. 基于转录组测序的石斛生物碱和人参皂苷生物合成相关基因的发掘, 克隆及鉴定 [D]. 北京: 北京协和医学院, 2013. |
| [20] | 苏文华, 张光飞, 周 鸿, 等. 氮素对短葶飞蓬生长和次生代谢产物积累的影响 [J]. 云南植物研究, 2010, 32(1): 41-46. |
| [21] | 苏文华, 张光飞, 周 鸿, 等. 短葶飞蓬黄酮及咖啡酸酯的含量与土壤氮供应量的关系 [J]. 植物生态学报, 2009, 33(5): 885-892. |
| [22] | Piotr C, Nicoletta S. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol chloroform extraction: twenty-something years on nature protocols [J]. Nat Protoc, 2006, 1(2): 581-584. |
| [23] | Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome [J]. Nat Biotechnol, 2011, 29(7): 644-652. |
| [24] | 刘春霞, 王 玥, 张云峰, 等. 氮, 磷营养限制及光照, 蔗糖浓度对黄酮代谢相关基因表达的影响 [J]. 江苏农业科学, 2010(1): 4-8. |
 2015, Vol. 46
2015, Vol. 46


