2. 内蒙古大学 自治区离子束生物工程重点实验室, 内蒙古 呼和浩特 010021
2. Key Laboratory of Ion Beam Bio-engineering, Inner Mongolia University, Hohhot 010021, China
黄芪Astragali Radix是一种传统中药材,具有“补药之长”的美称,黄芪品种繁多,目前以《中国药典》2010年版收载的黄芪为正品,黄芪为豆科植物蒙古黄芪Astragalus membranaceus (Fisch.) Bge. var. mongholicus (Bge.) Hsiao或膜荚黄芪A. membranaceus (Fisch.) Bge. 的干燥根。春、秋二季采挖,除去须根和根头,晒干。具有补气固表、利尿托毒、排脓、敛疮生肌的功效[1]。黄芪隶属于豆科,黄芪属是一个多态性类群,种类繁多,变异较大,全世界有11个亚属2 500多种,主要分布于北半球的温带地区和南美洲,中亚和西亚也有广泛分布[2]。
内蒙古地区黄芪的栽培历史悠久,清代吴其浚《植物名实图考》记载:黄芪“有数种,山西、蒙古产者佳。”由于黄芪主产于北方,现代中医处方,常常写为“北芪”。中药材的临床应用十分讲究道地。从本草考证可知,黄芪的道地产区有由西南向东北迁移的趋势,逐渐过渡至目前道地以山西、内蒙的蒙古黄芪为主的趋向,开展道地药材的研究不仅能体现中医药特色,而且是保证中医药持续发展的需要[3],目前由于黄芪栽培过程中存在种质混杂、退化等现象,使黄芪产量和质量均有所下降,严重影响了中药现代化、国际化的进程。因此需要积极开展黄芪种质资源的收集、保护和评价等基础研究工作。
ISSR分子标记技术以其遗传多态性高、稳定可靠、不受环境因素影响等优点,广泛应用于物种鉴定、遗传作图、基因定位、系统发育与进化等多个领域[4]。本课题应用ISSR分子标记技术,从稳定遗传的DNA分子水平上对内蒙古地区9个盟市不同来源的黄芪样本进行遗传多样性分析,为促进黄芪生物多样性的保护及可持续性利用、发挥蒙古黄芪的道地性优势、推进规范化和产业化生产奠定基础。
1 材料与仪器所用黄芪药材采自内蒙古9个盟市30个地区,包括共计80个样本的野生种和栽培种(表 1),经内蒙古医科大学张传领讲师鉴定为蒙古黄芪Astragalus membranaceus (Fisch.) Bge. var. mongholicus (Bge.) Hsiao和膜荚黄芪A. membranaceus(Fisch.) Bge. 的干燥根。dNTP(2 mmol/L)(购自invitrogen),10×PCR缓冲液、MgCl2(25 mmol/L)、Taq酶(5 U/μL)均购自Thermo Scientific,引物根据加拿大哥伦比亚大学(University of British Columbia,UBC)公布的ISSR引物设计,由上海生工生物工程技术有限公司合成。
| 表 1 样品来源信息 Table 1 Information of sample sources |
实验主要仪器有Sigma高速冷冻离心机、ND 2000C超微量分光光度计(Thermo)、梯度基因扩增仪(美国AB公司)、紫外凝胶成像分析系统(基因有限公司)。
2 方法 2.1 基因组DNA提取参考改进CTAB法[5]提取黄芪总DNA,采用2% CTAB提取液,其100 mL配方为:NaCl 8.19 g、1 mol/L Tris-HCl(pH 8.0)10 mL、CTAB 2 g,EDTA 0.584 5 g。用ND 2000C超微量分光光度计进行DNA浓度和纯度的检测,以A260/A280值在1.8~2.0为纯度满意,同时用1%琼脂糖凝胶电泳检验DNA质量,最后将其稀释至40 ng/μL,-20 ℃储存备用。
2.2 ISSR-PCR反应体系优化及引物筛选不同植物的ISSR反应体系中的各成分浓度、反应程序均不相同[6, 7],因此建立精确的ISSR-PCR反应体系对保证ISSR实验结果的准确可靠非常重要。本研究利用单因素实验和正交试验相结合的方法,首先对DNA模板、MgCl2、引物、dNTPs、TaqDNA聚合酶5个因素分别进行单因素浓度梯度实验,由实验结果发现,DNA浓度梯度对反应的影响并不大,在10~50 ng均条带清楚,而到60 ng时,背景略深,条带清晰度减弱,本实验选择40 ng的DNA模板为黄芪ISSR-PCR反应体系的最佳用量。然后从其余4个因素中初步选出条带相对清晰的3个浓度(水平)进行后续正交试验,反应体系见表 2。
| 表 2 正交试验反应体系 Table 2 Reaction system of orthogonal test |
从UBC公布的96条ISSR引物中选出22条引物对随机选取2个地区的黄芪DNA样本进行扩增预试验,同时对每一个引物进行梯度退火实验,确定最佳退火温度,最终筛选出了15条条带数目相对较多、条带清晰且重复性好的引物进行后续的ISSR分析。
2.3 ISSR扩增及产物检测PCR扩增程序:94 ℃预变性5 min,94 ℃变性45 s,52 ℃退火45 s,72 ℃延伸2 min,40个循环后,最后72 ℃延伸7 min,4 ℃保存。
取ISSR-PCR扩增产物5 μL,在0.5×TBE缓冲液中2%琼脂糖凝胶100 V电压电泳过中线,以DL2000为Marker,EB(0.5 μg/mL)染色15 min,凝胶成像分析。
2.4 数据分析ISSR扩增图谱在相同迁移率位置上,有带记为“1”,无带记为“0”形成“0,1”矩阵图输入Excel表格,用SLT_NTsys2.10e遗传多样性分析软件计算任意样品间的遗传距离,构建聚类图。
3 结果 3.1 黄芪ISSR-PCR最佳反应体系建立由正交试验PCR产物电泳结果(图 1)发现,3号组合反应体系条带数最多且最清晰,最终确定最优ISSR-PCR反应体系:10×PCR缓冲液 2.0 μL,MgCl2 1.5 mmol/L,引物0.5 μmol/L,dNTP 0.4 mmol/L,Taq酶1.5 U,DNA模板40 ng,总反应体积为20 μL,该反应体系产生的ISSR图谱清晰,反应系统稳定,适合黄芪样本的ISSR分析。
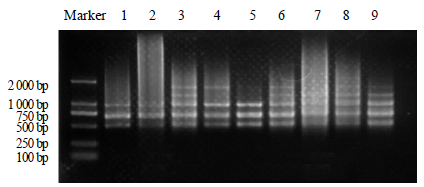 | 图 1 ISSR-PCR电泳图 Fig.1 Electrophoretogramof ISSR-PCR |
15条ISSR引物共扩增出169条带,其中157条表现出多态性,占总带数的92.9%,平均每条引物扩增的DNA谱带数为11条,变幅在8~14,扩增条带的相对分子质量从100~2 000 bp不等,见图 2。多态位点百分率(PPB)在75%~100%(表 3)。
 |
1~24-随机选取的样本 1—24-radom samples 图 2 引物819对部分黄芪样本扩增结果 Fig.2 Amplification of primer 819 on some A. membranaceus samples |
| 表 3 ISSR引物最佳退火温度及多态性分析 Table 3 Optimum annealing temperature of ISSR primers and polymorphism analysis |
基于ISSR扩增所产生的169条DNA带,以30个采集地的黄芪样本为代表经遗传统计软件分析获得了遗传距离矩阵,结果发现,各地区栽培蒙古黄芪之间的遗传距离分布在0.242 7~0.730 8,变幅较大;栽培膜荚黄芪之间的遗传距离分布在0.353 8~0.603 8,相对变幅要小一些。而来自同一地区乌兰察布市的野生蒙古黄芪(F2)与当地栽培种(F1、F3、F4)之间的遗传距离变幅为0.338 3~0.423 7,差异不大;来自同一地区呼和浩特的野生(H1)和当地栽培种(H2~H5)之间的遗传距离变幅为0.426 6~0.606 1,差异相对明显。
对内蒙古9个盟市30个黄芪样本采集地的ISSR进行聚类分析,得树状图(图 3),由图 3可以看出,所有材料在遗传系数为0.60处形成2大分支,其中来源于乌兰察布市的黄芪(F1~F4)明显有别于其他各盟市,并且与地域很近的呼和浩特野生种H1聚在了一支,这一分支中共包含2个野生种(F2和H1),说明乌兰察布市的栽培品种与野生种亲缘关系很近。
 | 图 3 内蒙古地区不同居群黄芪ISSR标记聚类图 Fig.3 Clustering dendrogram of ISSR of A. membranaceus from different populations in Inner Mongolia |
在第2大分支中,在遗传系数0.62处又明显地分为3类,其中来自呼伦贝尔的野生种(A1)单独为一类;兴安盟(B)、锡盟(C)、通辽(D)的黄芪聚在一起,赤峰(E)、包头(G)、呼和浩特(H)的黄芪聚在一类,除个别的样品外,整体上呈现出从北到南按地域聚类的特点。
通过聚类分析还发现,样本中的栽培膜荚黄芪(B2、H5、E3、H3、E1、E7、E6)聚在了一大类中,但比较分散,此类中也包含蒙古黄芪,这与张茹等[8]研究的“不同产地、不同种的黄芪是先以产地聚类,后以种来聚类,表明蒙古黄芪虽为膜荚黄芪变种,但是其遗传变异程度还未达到形成一个新的物种的程度”结论相一致。
4 讨论DNA分子标记是指能反映生物个体或种群间基因组中某种差异特征的DNA片段,它能够直接反映基因组DNA间的差异[9],本实验应用的ISSR分子标记因其操作方便,经济实惠,不需要知道任何靶标序列的背景信息,且具有更高的遗传多态性和重现性[10],该技术已被成功地应用于植物遗传多样性、DNA指纹图谱绘制、分子生态学研究等众多领域。然而,ISSR标记技术也有不足[11],其不足之处在于PCR扩增反应的最适条件需要一定时间摸索,因此,本研究采用单因素实验和正交试验相结合的实验设计方法,对影响ISSR-PCR反应的多个因素进行了系列调整与优化,实验结果表明,ISSR反应对DNA模板的用量不是特别的敏感,在20 μL的反应体系中DNA量在10~50 ng均可获得比较理想的实验结果;实验中发现Taq 酶、dNTPs、Mg2+、引物浓度对PCR产率及多态性水平扩增都有着很大的影响;因此,要获得较稳定、清晰、可重复的条带,对ISSR-PCR反应体系的各种影响因子进行实验是完全必要的。同时,PCR扩增程序中,退火温度能明显地影响ISSR-PCR扩增条带特异性,退火温度过低或过高都会导致特异性条带减少,因此本实验还对筛选出的15条引物逐个进行退火温度梯度实验,确定了每个引物的最佳退火温度。本实验15条ISSR引物扩增产物表现出的PPB在75%~100%,显示黄芪药材基因多样性很丰富,其引物880、827、816、829、828的扩增PPB为100%,可用于后期黄芪种质资源鉴定研究的一个参考。
内蒙古地区南北地域狭长,地理生态环境差异大,黄芪的遗传多样性丰富。本实验通过对内蒙古地区9个不同盟市黄芪样本间的遗传多样性分析发现,内蒙古黄芪的遗传距离变异幅度很大,地域相距不远栽培的膜荚黄芪、蒙古黄芪和野生黄芪之间遗传距离最大,亲缘关系最远,这可能与种质资源本身遗传差异及生存环境迥异有关,而来自同一盟市不同居群的黄芪样本遗传距离最小,亲缘关系最近,体现出栽培过程中生态环境对种质资源的影响,在栽培过程中,人工选择的结果使黄芪种质趋于更加接近。总体上内蒙古黄芪遗传距离的远近与地理位置生态环境具有一定的联系但不绝对,可能与药用植物本身对环境的适应能力和药用植物通过遗传变异适应不同生境有关。
通过对内蒙古地区黄芪样本聚类分析发现,野生来源的黄芪A1明显区别于其他地区的栽培黄芪而单独聚为一类,但来自乌兰察布市不同采样地点的栽培种F1、F3、F4与野生种F2及呼和浩特野生种H1聚在了一大类中,体现出乌兰察布市该地区的黄芪栽培品种与野生黄芪资源遗传相似度高,亲缘关系更为接近,这可能与乌兰察布市黄芪栽培的地理环境与野生环境更为接近有关,如果后续实验能对该地区的栽培黄芪与野生黄芪药材主成分量上做进一步分析比较,将会对发掘内蒙古药性强的优势黄芪种质资源起到极大的促进作用。
黄芪是常用的中医临床用药,商品需求量大,长期受到掠夺性的开采,野生资源已近枯竭[12],目前市场上的黄芪主要来源于人工栽培,内蒙的黄芪以其含粉质、糖分高、条杆粗壮、药性最强、品质最好畅销国内外,因此,对内蒙古黄芪种质资源进行遗传多样性分析和客观真实评价,为发挥内蒙古黄芪的道地性优势和深度挖掘内蒙古黄芪优良品种遗传潜力、保护当地黄芪种质资源具有重要意义。
| [1] | 中国药典 [S]. 一部. 2010. |
| [2] | 中国科学院中国植物志编辑委员会. 中国植物志 [M]. 北京: 科学出版社, 1993. |
| [3] | 杨庆珍, 刘德旺, 王冬梅, 等. 黄芪生态型与品质的相关性研究 [J]. 中草药, 2014, 45(16): 2395-2399. |
| [4] | 张安世, 刘莹, 赵利新. 均匀设计优化太行菊ISSR-PCR反应体系 [J]. 生物技术, 2013, 23(6): 45-49. |
| [5] | 刘敏, 严萍, 詹若挺, 等. 黄芪干燥根DNA提取方法的研究 [J]. 药物生物技术, 2010, 17(5): 393-396. |
| [6] | 孙稚颖, 姚辉. 不同产地菘蓝ISSR分析与鉴定 [J]. 中草药, 2014, 45(22): 3323-3326. |
| [7] | 欧立军, 颜旺, 廖亚西, 等. 天门冬ISSR分子标记技术的建立与体系优化 [J]. 中草药, 2011, 42(2): 353-357. |
| [8] | 张茹. 药用黄芪ISSR遗传多样性分析 [D]. 太原: 山西农业大学, 2014. |
| [9] | 唐晓晶. DNA分子标记在中药材鉴定中的应用研究 [D]. 北京: 中国中医科学院中药研究所, 2006. |
| [10] | Ye C J, Yu Z W, Kong F N, et al. R-ISSR as a new tool for genomic fingerprinting, mapping, and gene tagging [J]. Plant Mol Biol Rep, 2005, 23(2): 167-177. |
| [11] | 周延清. 简单重复序列间区标记技术 [M]. 北京: 化学工业出版社-现代生物技术与医药科技出版中心, 2005. |
| [12] | 张兰涛. 黄芪种质资源评价研究 [D]. 北京: 中国协和医科大学, 2004. |
 2015, Vol. 46
2015, Vol. 46


