2. 广州中医药大学中药学院, 广东 广州 510006
2. School of Chinese Materia Medica, Guangzhou University of Chinese Medicine, Guangzhou 510006, China
单核苷酸多态性(single nucleotide polymorphism,SNP)是指基因组中由于单个碱基的替换、插入或缺失所引起的多态性[1],广泛存于基因序列中,参与了基因的表达水平高低以及表达产物活性强弱的调控[2]。在药用植物体内调控活性成分生物合成过程的关键酶基因表达水平或表达产物活性强弱如果受SNP的影响而发生变化,就会引起这些成分在中药材的量出现变化,进而影响药效,从而对中药材的质量产生显著影响。已有研究证实SNP可以影响甘草的活性成分甘草酸的量高低[3, 4]。因此,选择中药材活性成分生物合成的关键酶基因,研究其SNP与活性成分量及药效的关系,可为研究中药材的种质资源以及建立评价中药材真伪优劣的新方法提供有益的参考。由于绝大多数中药材尚缺乏基因组序列数据,对中药材单个基因的研究也不多,这就很难找到中药材关键酶基因SNP的信息,也就无从研究SNP与活性成分量及药效的关系。
人参来源于五加科(Araliaceae)植物Panax ginseng C. A. Mey. 的干燥根[5],是最重要的中药材之一,具有滋补强壮、补气养血等多种功效,被誉为“百药之王”,在我国已有2 000多年的栽培和应用历史。我国人参栽培地域广阔,生态条件千差万别,加上长期的人工选择,形成了种类繁多的农家类型[6]。人参的临床疗效、药理活性、人参皂苷量与其产地、栽培品种、生长年限等关联紧密[7, 8, 9]。人参皂苷是人参中公认的主要活性成分,其生物合成过程较为清楚,其中一个关键酶为达玛烯二醇合成酶(dammarenediol synthase,DS)[10],研究该基因的SNP对于评价人参种质资源的遗传多样性,建立更加有效的人参质量评价新方法,合理开发利用人参种质资源具有重要意义,目前还未见其相关SNP研究报道。因此,本研究选择DS基因为研究对象,采用巢式PCR-直接测序法,通过对不同样品的序列比对,以发现人参的DS基因序列是否存在SNP及其类型。
1 仪器与材料 1.1 主要试剂及仪器EasyPure Plant RNA试剂盒、5K DNA Ladder购自北京全氏金生物技术有限公司,2×PCR PrimeSTAR HS(Premix)购自Taraka生物公司,Thermo Maxima 1st strand cDNA synthesis Kit逆转录试剂盒购自Thermo Scientific生物公司。Veriti 96-Well Fast Thermal Cycler(美国Applied Biosystems公司),TissuelyserII组织研磨机(德国Qiagen公司),Eppendorf 5424R台式冷冻高速离心机(德国Eppendorf公司),Gel Doc XR+凝胶成像仪(美国Biorad公司)。
1.2 材料人参Panax ginseng C. A. Mey. 样品采自我国吉林、辽宁等人参主产区的种植基地,并记录GPS位置信息,清洗干净后,置-80 ℃保存于澳门科技大学中药质量研究国家重点实验室,由广州中医药大学吴文如副教授鉴定,样品信息见表 1。
| 表 1 序列分析所用的样品 Table 1 Samples used in sequence analysis |
根据Easy Pure Plant RNA试剂盒使用指南进行总RNA的提取。步骤:将存储于-80 ℃的人参取出,切取0.2 g组织,用无菌水冲洗干净,置于2 mL EP管中,加入1 mL BB6(含20 μL β-巯基乙醇)以及1粒5 mm瓷珠,于Tissuelyser II组织研磨机中研磨至无组织块,室温孵育3 min。12 000×g离心2~5 min,小心吸取上清至RNase-free的离心管中。向上清中加入0.5倍体积无水乙醇,涡旋彻底混匀,分散沉淀。将得到的溶液和沉淀一起加入离心柱中,12 000×g离心30 s,弃掉流出液。加500 μL CB6,室温,12 000×g离心30 s,弃掉流出液。向离心柱中央加入80 μL的DNase I工作液(70 μL 缓冲液+10 μL DNase I),室温放置15 min,加500 μL CB6,室温12 000×g离心30 s,弃掉流出液。加500 μL WB6,12 000×g离心30 s,弃掉流出液,并重复此步骤1次。室温12 000×g离心2 min,彻底去除残留的乙醇,在室温静置数分钟彻底晾干离心柱。加50 μL RNase-free H2O在离心柱的中央,室温静置1 min。室温12 000×g离心2 min,洗脱RNA。将RNA 置于-80 ℃保存。
逆转录反应按Thermo Maxima 1st strand cDNA synthesis Kit逆转录试剂盒操作指南进行,20 μL的反应体系包括人参总RNA约100 ng,5×Reaction Mix 4 μL,Maxima Enzyme mix 2 μL,离心混匀。反应程序:25 ℃、10 min,50 ℃、15 min,85 ℃、5 min。产物于-20 ℃保存。
2.2 人参DS基因巢式扩增引物的设计与合成在GenBank(http://www.ncbi.nlm.nih. gov/GenBank/)检索到人参DS基因记录5条,其中包含有mRNA的编码区(长度2 310 bp)序列3条(GenBank登录号GU183405.1、AB265170.1、JN596111.1),利用在线工具BLAST(http://blast.ncbi.nlm.nih. gov/Blast.cgi)比对分析,相似度为99%。根据DS基因保守序列,采用引物设计软件Primer 5.0,选择巢式PCR的方式扩增,设计了一对外侧引物F1和R1,以获得包括编码区序列的基因模板;同时设计了2对上下游分段内侧引物F2、R2以及F3、R3,其中包括大于100 bp的序列重叠,以便覆盖包含DS基因编码区的完整序列。引物交由北京六合华大基因科技股份有限公司合成,采用MOP方式纯化。引物序列见表 2,引物设计示意图见图 1。
| 表 2 人参DS基因巢式PCR引物序列 Table 2 Nested PCR primers in DC gene of P. ginsengused |
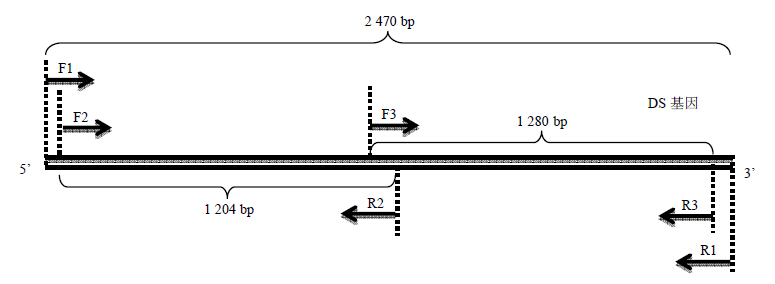 | 图 1 DS基因巢式PCR引物设计示意图Fig.1 Amplified region by nested PCR primers in DS gene |
采用巢式PCR的方式,选择引物F1、R1进行第1轮扩增,取其产物2 μL作为第2轮扩增的模板,至2个PCR管中,分别选择引物F2、R2和F3、R3进行第2轮扩增。反应体系:Premix PrimeSTAR HS 25 μL,模板2 μL,引物(20 μmol/L)各0.7 μL,加水至50 μL。反应程序:预变性94 ℃、2 min,98 ℃变性10 s,61~63 ℃(根据引物不同)退火10 s,72 ℃延伸,共35个循环,最后72 ℃延伸7 min,产物于4 ℃保存。
2.4 人参DS基因PCR产物电泳检测和测序PCR产物的电泳检测选择1.5%琼脂糖凝胶,电压100 V,时间30 min。PCR产物和DNA Marker各上样3 μL。电泳结束后,取出凝胶,于Gel Doc XR+凝胶成像仪中拍照记录。
选择含有预期片段大小的PCR产物原液,按Sanger法进行测序,双向测序,利用CodonCode Aligner 4.24软件进行拼接。测序工作由深圳华大基因公司广州测序部进行。
2.5 人参DS基因序列的比对分析各样品的测序结果通过在线工具BLAST进行序列的查询和同源性比较,搜索参数:数据库选择Necleotide collection(nr/nt),方法使用Highly similar sequences(megablast)。利用DNAMAN7.0生物统计软件分析各样品序列间的相似度,同时生成典型序列,并将得到的典型序列与GenBank中收录的人参(登录号:GU183405.1、AB265170.1、JN596111.1、BD420539.1)、西洋参(登录号:GU997679.1、KC316048.1、KC524472.1)、三七(登录号:GU997680.1、KC422652.1、KC953035.1)、越南人参(登录号:KF306328.1)等物种的DS基因进行同源性比较,并以越南人参为外类群,构建进化树。由于DNA序列是具有离散特征状态的数据,因此采用最大似然法对多序列比对后的数据进行聚类分析[11]。利用DNAMAN软件计算样品间的遗传距离并发现SNP。
3 结果与分析 3.1 PCR扩增产物电泳结果巢式PCR第1轮扩增产物电泳凝胶图显示有约为2 470 bp的扩增条带,与设计的DS基因目的条带相符,但也出现较多杂带,仅极少量样品未见2 470 bp目的条带(图 2)。
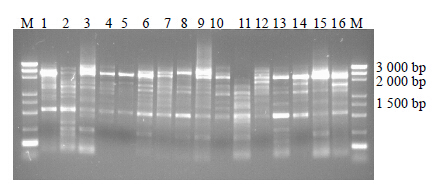 | M-Marker 1~6-DMYHJ5 7~12-SZ6 13~15-SZ10 16-SZ15,下同 M-Marker 1—6-DMYHJ5 7—12-SZ6 13—15-SZ10 16-SZ15,same as below 图 2 巢式PCR第1轮引物F1、R1扩增部分产物电泳结果Fig.2 Electrophoresis of amplification partial products F1 and R1 of 1st round of nested PCR |
巢式PCR第2轮扩增产物电泳凝胶图显示,F2、R2和F3、R3产物分别出现了约为1 204 bp和1 280 bp的扩增条带,与设计目的条带相符,且亮度极高,背景清晰,符合测序要求(图 3、4)。57份人参样品中,仅3份样品的DS基因片段未成功扩增,共获得113个符合测序要求的DS基因上下游分段PCR产物,其中成功测序并拼接序列103条,DS上段测序成功率为82.5%,下段为98.2%(表 1),表明扩增成功率和测序成功率较高。
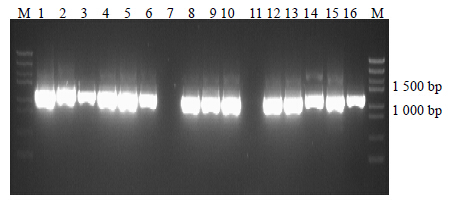 | 图 3 巢式PCR第2轮引物F2、R2扩增部分产物电泳结果Fig.3 Electrophoresis of amplification partial products F2 and R2 of 2nd round of nested PCR |
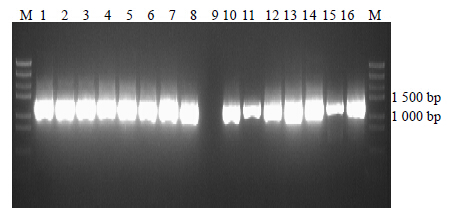 | 图 4 巢式PCR第2轮引物F3、R3扩增部分产物电泳结果Fig.4 Electrophoresis of amplification partial products F3 and R3 of 2nd round of nested PCR |
成功测序并拼接的DS基因上下游分段序列共103条,其典型序列如图 5和6,序列间的相似度达99%以上。得到的典型序列(DS consensus sequence)与GenBank中收录的人参等物种的DS基因同源性比较,结果与人参DS基因序列(登录号:GU183405.1、AB265170.1、JN596111.1、BD420539.1)相似度高达100%,判断扩增所得到的序列为DS基因。遗传聚类分析结果表明,越南人参为一类;三七单独为一类,人参和西洋参聚类在一起,但与三七遗传距离差异不大,结果见图 7。
 | 图 5 DS基因上游分段典型序列 (DMYHJ5-3)Fig.5 Typical sequence of DMYHJ5-3 amplified from upstream of DS gene |
 | 图 6 DS基因下游分段典型序列 (DMYLX6-6)Fig.6 Typical sequence of DMYLX6-6 amplified from downstream of DS gene |
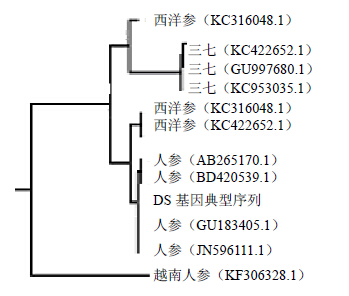 | 图 7 人参样品DS基因典型序列同源性比对结果Fig.7 Homologous alignment comparison of typical sequence in DS gene of P. ginseng samples |
利用DNAMAN软件,以人参DS基因(AB265170.1)序列为参照,对所得的103条序列进行了SNP位点分析和比对,发现了6个样品共有7个SNP,结果见图 8。本研究中获得的DNA序列已在美国国立生物信息中心基因库(GenBank,National Center for Biotechnology Information)注册,Accession No.:KM232663、KM232664、KM232665、KM232666、KM232667、KM232668。
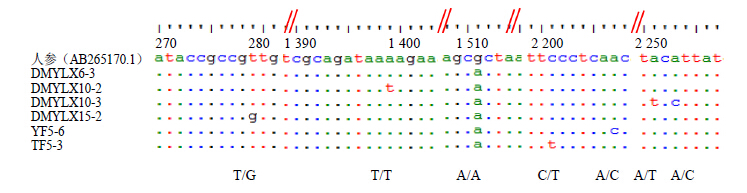 | 图 8 人参样品DS基因片段SNP位点Fig.8 SNP locus in DS gene of P. ginseng samples |
检测未知SNP有许多种方法,如温度梯度凝胶电泳(TGGE)、变性梯度凝胶电泳(DGGE)、单链构象多态性(SSCP)、变性的高效液相色谱检测(DHPLC)、限制性片段长度多态性(RFLP)、随机扩增多态性DNA(RAPD)等,但这些方法只能发现样品是否含有SNP,不能确知SNP的位置和碱基类别。要想发现新的SNP,必须对那些含有SNP的DNA进行测序以直接获取目的片段的碱基序列,再通过序列间的比较,就可以直观而有效地找出SNP,对于一些比较小的基因,其检出率高达100%[12]。因此,本研究采用了对扩增产物进行直接测序方法以发现人参DS基因中的SNP。
DNA片段的直接测序中,引物的合理设计是保证成功扩增和测序的前提。引物最好在模板cDNA的保守区域内设计,这可通过物种间相似序列的比较而确定。在NCBI上搜索不同物种的同一基因,通过序列分析软件,各基因相同的序列就是该基因的保守区。为保证引物的特异性,通常引物设计完成后,应对其进行BLAST检测,如果与其他基因不具有互补性,就可以进行下一步的实验。但由于目前缺乏药用植物的基因组序列,因此难以判断本研究中所设计的引物的特异性。为解决这个问题,研究中采用了特异性强和扩增效率高的巢式PCR技术进行DS序列的扩增,结果产物质量好,测序成功率高。巢式PCR利用2套PCR引物对进行2轮PCR扩增反应。在第1轮扩增中,以植物基因为模板,利用外侧引物扩增产生目的基因片段,再以此基因片段产物为模板,利用内侧引物进行第2轮扩增。因为与2套引物都互补的模板很少,通过2次扩增,降低了扩增多个靶位点的可能性,从而增加了检测的特异性和可靠性。
采用Sanger法1次单向测序,一般只能保证800 bp左右的碱基结果是比较可靠的。然而人参DS基因mRNA的编码区序列长度为2 310 bp,因此难以通过1次PCR产物的测序获得全长测序。研究中将DS基因分为上下游2段,采用分段引物,并设计了其产物包括大于100 bp的序列重叠,以便于进行序列的拼接。为充分保证测序结果的真实性,研究中还结合采用双向测序,再通过CodonCode Aligner软件进行序列对比修正和拼接,以消除测序误差。由于对序列两端约50 bp误差部分进行的剪切,使得DS上、下2段序列之间仍存在约24 bp的序列未覆盖,未能拼接包含DS基因编码区的完整序列。有待针对中间部分序列再设计第3对引物,以实现DS基因编码区的完整测序。
在SNP检测诸多方法中,假阳性是造成多种人工精心设计的方法无法进入临床应用的最大障碍[13]。实验中采用了兼具高保真性和高扩增效率的PCR用DNA聚合酶。因其具有极强的3’→5’外切酶活性而显示出超群的校正功能,可以实现对靶序列的高保真性、高灵敏度、高特异性、高成功率的扩增,从而保证测序结果真实可信。
本研究发现的DS基因SNP标记来源于DNA的转录区域。由于选择压力,SNP在单个基因、整个基因组中以及种群间的分布是不均匀的。SNP在非编码区中要多于编码区,而且在编码区也是非同义突变(有氨基酸序列的改变)的频率比其他方式突变的频率低得多[14]。与现在常用的其他DNA分子标记,如ITS、SSR等相比较,虽然DS基因其多态性较低,但该基因来源于表达功能基因,其SNP可成为一种反映功能变化的分子标记。DS基因是调控中药人参的主要活性成分人参皂苷生物合成过程的一个关键酶,其SNP可以影响人参皂苷的生物合成,例如引起人参皂苷种类和量上的差异,从而导致药效的变化。本研究初步建立了发现人参DS基因SNP的方法,也可用于建立检测人参样品的DS基因是否含有SNP及其类型的方法,还可用于分析SNP与药效、活性成分量的关系,从而建立反映人参药效的质量评价新方法。本研究也为大多数中药缺乏基因组序列数据的情况下,进行中药材基因SNP的研究提供了一个可行方法,有望为人参种质资源研究,人参属药材的品种分类、鉴定、遗传研究,以及建立反映人参药效的质量评价新方法研究提供有价值遗传研究工具和分子标记资源,也可供同类药材研究参考。
| [1] | Brooker A J. The essence of SNPs [J]. Gene, 1999, 2344(2): 177-186. |
| [2] | 张必弦, 朱延明, 来永才, 等. 单核苷酸多态性 (SNP) 的研究进展 [J]. 安徽农业科学, 2012, 40(21): 10755-10756. |
| [3] | 沈湛云. 甘草β-AS基因多态性和β-香树酯醇合成的相关性研究 [D]. 北京: 北京中医药大学, 2010. |
| [4] | 张 宁. 甘草鲨烯合酶编码基因多态性及其对鲨烯积累的影响研究 [D]. 北京: 北京中医药大学, 2010. |
| [5] | 中国药典 [S]. 一部. 2010. |
| [6] | 宿武林, 吕学明, 宿艳霞. 论长白山人参主要品种类型栽种方式及其商品分类 [J]. 人参研究, 2008, 20(4): 34-39. |
| [7] | 侯双利, 韩 梅, 刘翠晶, 等. 人参β-actin基因实时荧光定量PCR方法的建立 [J]. 中草药, 2014, 45(17): 2530-2533. |
| [8] | 陈文学. 不同农家类型人参质量评价 [D]. 长春: 吉林农业大学, 2011. |
| [9] | 姬 庆, 郜玉钢, 赵 岩, 等. 酶法转化制备人参皂苷单体的研究进展 [J]. 中草药, 2014, 45(22): 3356-3360. |
| [10] | Liang Y, Zhao S. Progress in understanding of ginsenoside biosynthesis [J]. Plant Biol, 2008, 10(4): 415-421. |
| [11] | 常 青, 周开亚. 分子进化研究中系统发生树的重建 [J]. 生物多样性, 1998, 6(1): 55-62. |
| [12] | 高秀丽, 景奉香, 杨剑波, 等. 单核苷酸多态性检测分析技术 [J]. 遗传, 2005, 27(1): 110-122. |
| [13] | 陈琳玲, 张 佳, 彭翠英, 等. DNA聚合酶高保真机理的新发现及其在SNP分析中的应用 [J]. 遗传, 2005, 27(2): 279-283. |
| [14] | Zhao Z M, Fu Y X, Hewett-Emmett D, et al. Investigating single nucleotide polymorphism (SNP) density in the human genome and its implications for molecular evolution [J]. Gene, 2003, 312: 207-213. |
 2015, Vol. 46
2015, Vol. 46



