苦荞Fagopyrum tatarium (L.) Gaertn. 又名鞑靼荞麦,是一种具有药食两用价值的小杂粮[1]。由于含有丰富的营养物质,尤其是黄酮类化合物,苦荞逐渐以保健食品的身份被大众所接受。因此,诸如苦荞芽菜这样的苦荞衍生产品逐渐被开发出来用于商业目的。芦丁是芽期苦荞中的主要黄酮类生物活性物质[2],但随着近年来多种花青素在芽期苦荞中被鉴定,花青素作为苦荞中一种重要的生物活性物质也得以证明[3]。花青素不仅为苦荞提供了大部分的红色色素,而且具有较高的抗氧化能力[4],另外消费者也更青睐红色的苦荞芽菜。因此,促进花青素的积累能进一步提高苦荞芽菜的品质和经济价值[5]。
花青素的代谢途径属于苯丙氨代谢途径的一个分支,花青素的生物合成则是由多个结构基因编码的蛋白构成多酶复合体而完成。类黄酮3′-羟化酶(flavonoid 3′-hydroxylase,F3′H)属于细胞色素P450单加氧酶类,是由二氢山柰酚合成花青素途径中的第一个关键酶,它能在辅因子NADPH和O2的作用下,催化二氢山柰酚的单加氧反应形成二氢槲皮素,并在花青素合成相关酶的进一步催化下生成紫红色花青素(矢车菊色素,cyanidin),使植物果实和花瓣呈现红色[6]。研究表明,F3′H基因在转录水平上的丰度能有效影响花青素的合成[7,8,9,10],且其表达丰度受到多种环境因子的影响[11,12]。虽然该基因在其他植物中的研究已比较成熟,但在苦荞中,F3′H基因的研究未见相关报道,其表达水平与苦荞花青素积累之间的关系也有待探讨。
本研究以苦荞栽培品种“西荞2号”为材料,利用同源克隆和cDNA末端快速克隆(RACE)技术克隆苦荞F3′H(FtF3′H)基因,利用原核表达技术实现了FtF3′H 基因的异源表达,并且探讨了冷胁迫对芽期苦荞的子叶和胚轴中FtF3′H 基因的表达水平与花青素量的影响,为深入研究该酶在苦荞花青素代谢中调控机制提供基础资料。
1 材料与试剂 1.1 材料和菌种苦荞种子:“西荞2号”,由西昌学院王安虎教授鉴定并惠赠。大肠杆菌 DH5α、大肠杆菌BL21(DE3)和原核表达载体pET-30 b (+)质粒由本实验室保存。
1.2 试剂植物RNAout试剂盒(天恩泽基因工程有限公司);逆转录试剂盒RevertAidTM First Strand cDNA Synthesis Kit(Fermentas公司);cDNA末端快速克隆试剂盒SMART RACE cDNA Amplification Kit(Clontech公司);胶回收试剂盒、质粒DNA提取试剂盒、pMD 19-T Simple Vector克隆载体、Taq DNA聚合酶、Pyrobest® DNA Polymerase(高保真DNA聚合酶)、T4 DNA连接酶和限制性内切酶KpnI和BamHI等(大连宝生物公司);其他化学药品为进口或国产分析纯试剂。
2 方法 2.1 FtF3′H基因的克隆采用植物RNAout试剂盒提取苦荞花蕾总RNA,通过逆转录试剂盒RevertAidTM First Strand cDNA Synthesis Kit中的Oligo-dT引物逆转录以获得cDNA第一链,保存于-20 ℃冰箱备用。
根据甜荞(Fagopyrum esculentum Moench,ADT63065)、山葡萄(Vitis amurensis Rupr.,ACN38268)和拟南芥(Arabidopsis thaliana,NP_196416)的F3′H保守片段和特征氨基酸序列标签(GGEK),设计兼并引物F3′Hdf和F3′Hdr扩增FtF3′H基因保守片段。参照获得的保守片段,分别设计3’-RACE引物(F3′H3w、F3′H3z和F3′H3n)和5’-RACE引物(F3′H5w、F3′H5z和F3′H5n),并根据SMART RACE cDNA Amplification Kit说明扩增其3′-末端和5′-末端序列。根据DNAman7拼接获得的全长序列,设计特异引物F3′Hf和F3′Hr扩增FtF3′H基因的ORF序列。FtF3′H基因保守片段、3’-末端、5’-末端序列以及ORF序列扩增的PCR产物经回收克隆至pMD19-T Simple Vector,利用蓝白斑筛选、菌落PCR筛选阳性克隆子并送上海英骏公司测序。克隆所用引物见表 1。
|
|
表 1 基因克隆和半定量RT-PCR分析所用引物 Table 1 Primers used for gene cloning and semi-quantitativeRT-PCRanalysis |
利用DNAman 7进行序列拼接、基因编码蛋白的理化性质的推导。利用NCBI(http://www.ncbi.-nlm.nih.gov/)Blast-p和SignalP(http://www.cbs. dtu.-dk)对FtF3′H基因的氨基酸序列进行同源比对和信号肽分析。利用SPOMA(http://pbil.ibcp.fr/htm/index.php)对推导的氨基酸序列进行二级结构分析。
2.3 FtF3′H基因的原核表达以克隆至pMD19-T Simple Vector上的FtF3′H基因ORF序列为模板设计添加了酶切位点Kpn I和BamH I的引物F3′Hnf和F3′Hnr(表 1)。将扩增获得的含有酶切位点的PCR产物克隆至pMD19-T Simple Vector,构建重组质粒FtF3′H-T。将重组质粒FtF3′H-T 与表达载体pET-30b (+) 分别用Kpn I和BamH I双酶切,构建重组质粒pET-30b (+) - FtF3′H,并转化大肠杆菌DH5α感受态细胞,挑取多个单菌落,进行菌落PCR,单、双酶切(Kpn I和BamH I)鉴定和测序鉴定,正确的克隆用于下一步的诱导表达。
将重组质粒pET-30b (+) -FtF3′H转入大肠杆菌BL21(DE3)感受态细胞。挑取单菌落,接种于10 mL(含卡那霉素50 μg/mL)LB液体培养基中,于37 ℃摇床上培养16 h。按2%的接种量接种于50 mL LB培养基中(含卡那霉素50 μg/mL),37 ℃培养至吸光度(A600)值达到0.6时,加IPTG至终浓度为1 mmol/L,30 ℃低温诱导。诱导过程中0、1、2、4、6、8和10 h取3 mL菌液。以转入表达载体pET-30b (+) 空质粒的细胞作为对照,在诱导0和10 h时取3 mL菌液。所有样品经超声波破碎后,离心并收集上清,上清进行不连续SDS-PAGE(4%的浓缩胶、12.5%分离胶)电泳分析。
2.4 苦荞种子的萌发及冷胁迫处理苦荞种子的萌发参照Kim等[13]的方法,略有改动:将苦荞种子置于40 ℃水浴浸泡30 min后,置于25 ℃植物培养箱萌发。选取萌发后第8 d长势相近的2份芽期苦荞为材料,一份于25 ℃下继续培养4 d(14 h光照/10 h黑暗,相对湿度50%),作为对照组;另一份于4 ℃下继续培养4 d(14 h光照/10 h黑暗,相对湿度50%)进行冷胁迫处理,作为实验组。分别收集3份对照组和实验组的苦荞子叶和胚轴用于提取总RNA和花青素。
2.5 芽期苦荞不同组织FtF3′H基因表达量分析参照李成磊等[14]的方法采用半定量RT-PCR(semi-quantitative RT-PCR),以苦荞持家基因histone H3(HM628903,FtH3)作为内参,利用引物(表 1)FtF3′Hsmf 和 FtF3′Hsmr检测FtF3′H的基因表达量,以分析该基因在苦荞芽子叶和胚轴的表达水平。每条特定电泳条带的光密度通过Quantity One软件扫描获得,以FtF3′H光密度值与对应样本内参基因FtH3的光密度值的比值来表示FtF3′H基因的表达量。
2.6 芽期苦荞不同组织花青素的测定依照Chu等[15]的方法提取冷胁迫前后的苦荞芽子叶和胚轴的花青素。利用分光光度计分别测定样品在530 nm和657 nm波长下的A值和鲜质量(M),并计算样品中花青素的量(Q花青素)。
Q花青素=(A530-0.25×A657) / M
2.7 数据的分析和处理FtF3′H基因表达量和花青素的测定均设3组平行试验。采用SPSS 13.0统计软件通过Turkey’s检验和t-检验对芽期苦荞子叶和胚轴中花青素积累和FtF3′H基因的表达量在冷胁迫过程中的变化进行差异显著分析。
3 结果与分析 3.1 FtF3′H基因cDNA的克隆以获得cDNA模板为材料,使用兼并引物F3′Hdf和F3′Hdr从苦荞扩增出约300和500 bp的2条长片段(图 1-A)。测序结果经Blast分析表明,较小片段长度为344 bp,含有F3′H编码酶蛋白的特征序列GGEK。使用3条特异引物F3′H3w、F3′H3z 和F3′H3n经3’-RACE 3轮扩增后得到长约600 bp的特异条带(图 1-B)。测序结果表明,该片段长度为580 bp,包含终止密码TGA,3′-TUR和Poly A结构,表明获得该基因cDNA的3’末端。使用3条特异引物F3′H5w、F3′H5z和F3′H5n经5’-RACE 3轮扩增后得到1条约1 000 bp的特异条带(图 1-C)。测序结果表明,该片段长度为1 104 bp,包含起始密码ATG和5′-UTR。使用特异引物F3′Hf和F3′Hr扩增F3′H基因的cDNA,得到约1 600 bp的特异条带(图 1-D)。测序分析表明该序列长度为1 691 bp,其中包含1个1 470 bp的ORF,47 bp 的5′-UTR,155 bp的3′-TUR以及22 bp的Poly A尾,其编码氨基酸与其他植物来源的F3′H的氨基酸同源性为70%~80%,其中与山葡萄(V. amurensis,ACN38268)同源性最高为80%,与紫罗兰(Matthiola incana,AAG49301)同源性最低为70%。结果表明,已成功克隆苦荞F3′H基因ORF,命名为FtF3′H。
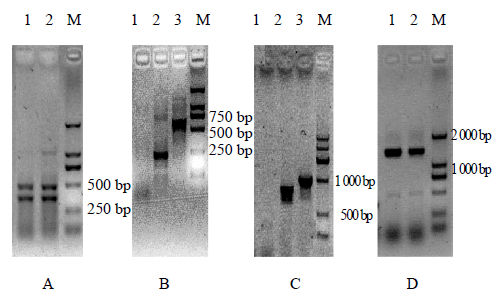 | A-1,2-FtF3′H保守片段的扩增产物 B-1-3-FtF3′H的3′-RACE的第1-3轮扩增产物 C-1-3-FtF3′H的5′-RACE第1-3轮扩增产物 D-1,2-FtF3′H ORF的扩增产物 M-Marker A-1,2 amplification of conserved fragment B-1-3 the first,second,and third round PCR products of 3′-RACE; C-1-3 the first,second,and third round PCR products of 5′-RACE D-1,2 amplification of FtF3′H ORF M-Marker图 1 FtF3′H基因的克隆 Fig. 1 Cloning of FtF3′H gene from F. tataricum |
利用DNAman7软件分析FtF3′H基因序列,结果显示FtF3′H基因编码1个包含489个氨基酸残基的蛋白,推导的相对分子质量为54 000,等电点(PI)为7.48。利用SPOMA分析表明FtF3′H二级结构包含47.03%的α-螺旋,9.82%的β-片层,4.7%的β-折叠以及38.45%的不规则卷曲。多重序列比对结果(图 2)显示,FtF3′H与其他植物来源的F3′Hs一样,拥有典型的细胞色素P450家族蛋白功能保守序列。其中位于N端的脯氨酸富集铰链区(Proline-rich “hinge” region)代表序列为“PPGPKPVP”,该铰链区不仅在蛋白的折叠过程中扮演者非常重要的角色[16],同时对F3′H蛋白锚定于内质网外膜也具有重要意义。其C端序列主要包含氧分子结合域(oxygen binding pocket motif)和血红素结合域(heme binding domain)2个结构域。其中氧分子结合域由高度保守序列“AGTDTS”组成,该序列与底物的选择和结合有关,被认为促使形成氧分子的结合域。另外,值得注意的是FtF3′H氨基酸序列包含“GGEK”序列,该序列是区分F3′H与F3′5′H重要标志,为所有F3′H所具备的一个重要特征[17]。
 | SRSI-底物识别为点1 Motif I-脯氨酸富集铰链区 Motif II-氧分子结合域 Motif III-EXXR triad Motif IV-血红素结合域 SRSI-substrate recognition site 1 Motif I-proline-rich “hinge” region Motif II-oxygen binding pocket motif Motif III-EXXR triad Motif IV-heme binding domain图 2 FtF3′H的多重序列比对 Fig. 2 Sequence multi-alignment of FtF3′Hs from F. tataricum |
含有重组质粒pET-30b (+) -FtF3′H的基因工程菌经IPTG诱导后,采用SDS-PAGE分析诱导表达产物(图 3)。结果显示,诱导后的实验组在相对分子质量66 400~44 300,出现一条明显的约为54 000的新生蛋白质条带,与预测的苦荞FtF3′H理论相对分子质量一致。产物表达量随IPTG诱导时间逐渐升高,表明FtF3′H基因在大肠杆菌中实现了异源表达。
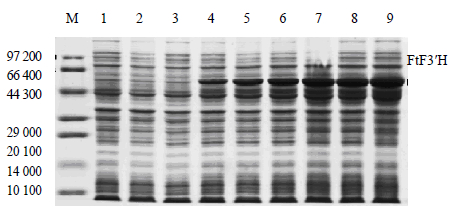 | M-蛋白质Marker1~2-分别为pET-30b (+) 空载体诱导前和诱导后;3~9-分别为重组质粒pET-30b (+)-FtF3′H诱导前和诱导后1、2、4、6、8和10 h M-standard protein Marker; Lanes 1—2-samples of negative control group collected before (1) and after (2) induction by IPTG; Lane 3-sample collected before induction by IPTG; Lanes 4-9-samples collected after induction by IPTG for 1,2,4,6,8,and 10 h图 3 重组FtF3′H蛋白表达的SDS-PAGE检测 Fig. 3 SDS-PAGE detection of recombinant FtF3′H expression |
采用半定量RT-PCR检测冷胁迫后FtF3′H基因在芽期苦荞子叶和胚轴中的相对表达量,结果见图 4。由图 4可以看出,对照组苦荞子叶和胚轴中FtF3′H基因的表达水平都较低,并且在不同组织中的表达量差异不显著(P>0.05)。当苦荞受到冷胁迫后,子叶和胚轴中FtF3′H基因的表达量显著升高(P<0.01),且在胚轴中的表达量是子叶中的1.27倍,差异极显著(P<0.01)。可见,FtF3′H基因参与了芽期苦荞冷胁迫的应答。
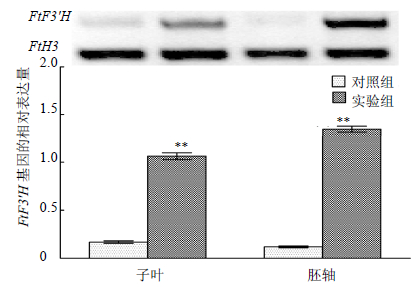 | 与对照组比较,**P < 0.01 **P < 0.01,vs control group图 4 冷胁迫下芽期苦荞FtF3′H基因表达量分析 (  ±s,n=3)
Fig. 4 Expression of FtF3′H from F. tataricum sprouts under cold stress ( ±s,n=3)
Fig. 4 Expression of FtF3′H from F. tataricum sprouts under cold stress ( ±s,n=3) ±s,n=3)
|
冷胁迫对芽期苦荞子叶和胚轴花青素量的影响,结果见图 5。由图可知,冷胁迫明显增加了芽期苦荞子叶和胚轴中的花青素量(P<0.01),且胚轴中的花青素量显著高于子叶(P<0.01),为子叶中花青素量的2倍。另外,虽然对照组苦荞子叶和胚轴中的花青素都较低,但胚轴中的花青素量也显著高于子叶(P<0.01)。可见,冷胁迫能显著提高芽期苦荞中花青素量,并且胚轴为芽期苦荞花青素积累的主要组织。
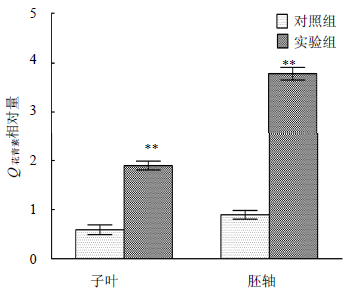 | 与对照组比较:**P < 0.01 **P < 0.01,vs control图 5 冷胁迫下芽期苦荞花青素的测定 (  ±s,n=3)
Fig. 5 Determination of anthocyanin content from F.tataricum sprouts under cold stress ( ±s,n=3)
Fig. 5 Determination of anthocyanin content from F.tataricum sprouts under cold stress ( ±s,n=3) ±s,n=3)
|
植物细胞色素P450含有多个保守结构域。其中值得注意的是“GGEK”序列不仅作为F3′H的重要标志,其在帮助形成有活性的F3′H过程中也扮演着重要角色。研究发现从大豆中获得的F3′H cDNA序列,由于编码区内单个碱基C的缺失,产生了一个缺少“GGEK”一致性序列和血红素结合域缩短的多肽链,由此导致蛋白质功能丧失[18]。本研究从苦荞中获得的FtF3′H基因所编码的蛋白具有该“GGEK”一致性序列并包含细胞色素P450的应有的其他保守结构域,因此可推断FtF3′H应该为一个具有活性的蛋白。F3′H基因的异源表达及其编码蛋白的活性测定相关研究资料比较丰富,例如高粱[19]、欧洲油菜[20]以及拟南芥[12]等植物来源的F3′H基因实现了大肠杆菌或者酵母中的异源表达并证明了其催化活性。本研究通过构建FtF3′H基因的原核表达载体成功实现了其在大肠杆菌中的异源表达,然而由于FtF3′H由427~444位的18个氨基酸残基组成了一个跨膜螺旋、缺少二硫键且为疏水性蛋白使其原核表达产物未能正确折叠并形成大量包涵体,因此本研究未能进行其活性的测定。
冷胁迫对花青素的生物合成的影响研究已经在多种植物中被报道。拟南芥中,冷胁迫明显提高了CHS、CHI和F3H基因的表达量,并导致花青素的大量积累[21];血橙中,冷胁迫显著增强了花青素合成相关基因(CM1、PAL、CHS、DFR、ANS、UFGT和 GST)的表达量和花青素的量[22]。同样在紫甘蓝的研究中,冷胁迫也被发现能上调花青素合成相关基因的表达量与提高花青素的量[23]。本研究发现,FtF3′H基因对冷胁迫的响应非常强烈,且花青素的量也于冷胁迫后显著升高。已有研究表明,矢车菊3-O-芸香糖苷是芽期苦荞中的主要花青素[24],另外FtF3′H是由二氢山柰酚合成矢车菊素类花青素途径中的第一个关键酶[6,25]。由此可推断,FtF3′H基因表达量的显著增加可能是为了满足在冷胁迫下苦荞对矢车菊3-O-芸香糖苷的需求,体现了FtF3′H在苦荞花青素合成中的重要地位。
目前关于F3′H的研究热点主要集中于其过量表达对花青素合成的影响。Ueyama等[26]通过抑制蝴蝶草中F3′5′H的表达,并同时过量表达F3′H发现,转基因蝴蝶草植株中矢车菊素类的花青素量显著升高。Han等[27]将MdF3′H于烟草中过量表达分析表明,转基因烟草中矢车菊素量明显高于野生型烟草。Shih等[10]将高粱SbF3′H基因转入拟南芥tt7突变体,使其重新获得在氮缺乏条件下积累矢车菊素类花青素的能力。本研究对芽期苦荞中FtF3′H基因在不同组织中的表达量进行了分析,并初步确立了FtF3′H基因在苦荞花青素合成中的重要地位。进一步通过转基因技术在烟草或者苦荞毛状根中稳定表达FtF3′H基因,深入研究该基因对花青素合成的影响具有重要意义。
| [1] | Kim S J, Zaidul I, Suzuki T, et al. Comparison of phenolic compositions between common and tartary buckwheat (Fagopyrum) sprouts [J]. Food Chem, 2008, 110(4): 814-820. |
| [2] | Kim S J, Kawaharada C, Suzuki T, et al. Effect of natural light periods on rutin, free amino acid and vitamin C contents in the sprouts of common (Fagopyrum esculentum Moench) and tartary (F. tataricum Gaertn.) buckwheats [J]. Food Sci Technol Res, 2006, 12(3): 199-205. |
| [3] | Li X, Thwe A A, Park N I, et al. Accumulation of phenylpropanoids and correlated gene expression during the development of tartary buckwheat sprouts [J]. J Agric Food Chem, 2012, 60(22): 5629-5635. |
| [4] | Watanabe M. An anthocyanin compound in buckwheat sprouts and its contribution to antioxidant capacity [J]. Biosci Biotechnol Biochem, 2007, 71(2): 579-582. |
| [5] | Tsurunaga Y, Takahashi T, Katsube T, et al. Effects of UV-B irradiation on the levels of anthocyanin, rutin and radical scavenging activity of buckwheat sprouts [J]. Food Chem, 2013, 141(1): 552-551. |
| [6] | 许志茹, 崔国新, 李春雷, 等. 芜菁的类黄酮3′羟化酶基因克隆和UV-A诱导表达特性 [J]. 植物生理学通讯, 2008, 44(5): 931-935. |
| [7] | Huang W, Sun W, Wang Y. Isolation and molecular characterisation of flavonoid 3′-hydroxylase and flavonoid 3′, 5′-hydroxylase genes from a traditional Chinese medicinal plant, Epimedium sagittatum [J]. Gene, 2012, 497(1): 125-130. |
| [8] | Zhou W, Gong Y, Lu X, et al. Molecular cloning and characterization of a flavonoid 3′-hydroxylase gene from purple-fleshed sweet potato (Ipomoea batatas) [J]. Mol Biol Rep, 2012, 39(1): 295-302. |
| [9] | Jeong S, Goto-Yamamoto N, Hashizume K, et al. Expression of the flavonoid 3′-hydroxylase and flavonoid 3′, 5′-hydroxylase genes and flavonoid composition in grape (Vitis vinifera) [J]. Plant Sci, 2006, 170(1): 61-69. |
| [10] | Shih C H, Chu I K, Yip W K, et al. Differential expression of two flavonoid 3′-hydroxylase cDNAs involved in biosynthesis of anthocyanin pigments and 3-deoxyanthocyanidin phytoalexins in sorghum [J]. Plant Cell Physiol, 2006, 47(10): 1412-1419. |
| [11] | Toda K, Akasaka M, Dubouzet E G, et al. Structure of flavonoid 3′-hydroxylase gene for pubescence color in soybean [J]. Crop Sci, 2005, 45(6): 2212-2217. |
| [12] | Schoenbohm C, Martens S, Eder C, et al. Identification of the Arabidopsis thaliana flavonoid 3′-hydroxylase gene and functional expression of the encoded P450 enzyme [J]. Biol Chem, 2000, 381(8): 749-753. |
| [13] | Kim H J, Park K J, Lim J H. Metabolomic analysis of phenolic compounds in buckwheat (Fagopyrum esculentum M.) sprouts treated with methyl jasmonate. [J]. J Agric Food Chem, 2011, 59(10): 5707-5713. |
| [14] | Li C, Bai Y, Li S, et al. Cloning, characterization, and activity analysis of a flavonol synthase gene FtFLS1 and its association with flavonoid content in tartary buckwheat [J]. J Agric Food Chem, 2012, 60(20): 5161-5168. |
| [15] | Chu H, Jeong J C, Kim W J, et al. Expression of the sweetpotato R2R3-type IbMYB1a gene induces anthocyanin accumulation in Arabidopsis [J]. Physiol Plant, 2012, 148(2): 189-199. |
| [16] | Kemper B. Structural basis for the role in protein folding of conserved proline-rich regions in cytochromes P450 [J]. Toxicol Appl Pharmacol, 2004, 199(3): 305-315. |
| [17] | Zabala G, Vodkin L. Cloning of the pleiotropic T locus in soybean and two recessive alleles that differentially affect structure and expression of the encoded flavonoid 3′ hydroxylase [J]. Genetics, 2003, 163(1): 295-309. |
| [18] | Toda K, Yang D, Yamanaka N, et al. A single-base deletion in soybean flavonoid 3′-hydroxylase gene is associated with gray pubescence color [J]. Plant Mol Biol, 2002, 50(2): 187-196. |
| [19] | Boddu J, Svabek C, Sekhon R, et al. Expression of a putative flavonoid 3′-hydroxylase in sorghum mesocotyls synthesizing 3-deoxyanthocyanidin phytoalexins [J]. Physiol Mol Plant Pathol, 2004, 65(2): 101-113. |
| [20] | Xu B B, Li J N, Zhang X K, et al. Cloning and molecular characterization of a functional flavonoid 3′-hydroxylase gene from Brassica napus [J]. J Plant Physiol, 2007, 164(3): 350-363. |
| [21] | Zhang F, Gonzalez A, Zhao M, et al. A network of redundant bHLH proteins functions in all TTG1-dependent pathways of Arabidopsis [J]. Development, 2003, 130(20): 4859-4869. |
| [22] | Crifò T, Petrone G, Lo Cicero L, et al. Short cold storage enhances the anthocyanin contents and level of transcripts related to their biosynthesis in blood oranges [J]. J Agric Food Chem, 2011, 60(1): 476-481. |
| [23] | Zhang B, Hu Z, Zhang Y, et al. A putative functional MYB transcription factor induced by low temperature regulates anthocyanin biosynthesis in purple kale (Brassica Oleracea var. acephala f. tricolor) [J]. Plant Cell Rep, 2012, 31(2): 281-289. |
| [24] | Kim S J, Zaidul I S M, Maeda T, et al. A time-course study of flavonoids in the sprouts of tartary (Fagopyrum tataricum Gaertn.) buckwheats [J]. Sci Hortic, 2007, 115(1): 13-18. |
| [25] | Brugliera F, Barri R G, Holton T A, et al. Isolation and characterization of a flavonoid 3′-hydroxylase cDNA clone corresponding to the Ht1 locus of Petunia hybrida [J]. Plant J, 1999, 19(4): 441-451. |
| [26] | Ueyama Y, Suzuki K I, Fukuchi-Mizutani M, et al. Molecular and biochemical characterization of torenia flavonoid 3′-hydroxylase and flavone synthase II and modification of flower color by modulating the expression of these genes [J]. Plant Sci, 2002, 163(2): 253-263. |
 2014, Vol.45
2014, Vol.45 


