2. 福建农林大学园林学院, 福建 福州 350002
2. College of Landscape Architecture, Fujian Agriculture and Forestry University, Fouzhou 350002, China
草珊瑚Sarcandra glabra (Thunb.) Nakai是一种广谱中药材,全株可入药,具有很高的药用价值。《中国药典》2010年版列为法定药材使用,还被广泛应用于保健品、饮料、食品、日用化工等方面,草珊瑚原料需求的不断增加,市场前景十分广阔[1]。其近缘植物有金粟兰属的金粟兰Chloranthus spicatus(Thunb.) Makino、及己C. serratus (Thunb.) Roem et Schult、宽叶金粟兰C. henryi Hemsl、四川金粟兰C. sessilifolius K. F. Wu、台湾金粟兰C. oldhamii等,形态极其相似,容易误采、误用,是与草珊瑚功效不尽相同的非药典收载的混淆品。而且金粟兰属植物多具有一定的肝毒性[2]。因此对草珊瑚种源的分类、鉴定和种群遗传关系研究具有重要的应用价值。
核糖体DNA中的18 S、5.8 S、28 S基因组序列在大多数生物中趋于保守,在生物种间变化小,而内转录间隔区(internal transcribed spacer,ITS)中的ITS1和ITS2作为非编码区,承受的选择压力较小,相对变化较大,并且能够提供详尽的系统学分析所需要的可遗传性状。通过ITS序列分析可以反映生物体在遗传上的变异,从分子水平理清其遗传背景[3],克服了传统研究方法的弱点,提高了药用植物种质资源研究的水平,为确定科学有效的保护策略和恢复措施提供依据。近年ITS已被广泛用于药用植物品种鉴定[4, 5]、亲缘关系确定[6, 7]、遗传多样性分析[8]等方面。本实验测定并分析了草珊瑚18个居群植物和金粟兰6个居群的ITS1、5.8 S、ITS2全长序列,并搜索GenBank数据库中收录的其他金粟兰属植物的ITS序列,探讨了草珊瑚及其他金粟兰属植物ITS序列的变异情况,为草珊瑚的资源鉴定、品种识别提供新思路。
1 仪器与材料PCR仪(Eppendorf,型号5332),电泳系统(北京市六一仪器厂,型号DYY—12),低温冷冻离心机(Eppendorf,型号5810R),凝胶成像分析仪(BIO-RAD ChemiDoc XRS),微量移液器(Eppendorf)。
CTAB提取液,TAE缓冲液,琼脂糖(Promega公司),溴化乙锭(Fluka公司),DNA Taq聚合酶(Takara公司),三氯甲烷、无水乙醇、异丙醇均为国产分析纯。
本研究实验样品是由福建中医药大学梁一池教授鉴定并收集的采自不同产地的18批样品,为金粟兰科植物草珊瑚Sarcandra glabra (Thunb.) Nakai,集中种植于福建中医药大学时珍园,金粟兰属其他植物则分别被鉴定并收集于福建中医药大学时珍园、漳州卫生职业学院与福建省热带作物科学研究所。并进行了随机的编号,来源见表 1。
| 表 1 草珊瑚及金粟兰属植物样品信息及rDNA序列特征 Table 1 Samples information and characteristics of rDNA sequences of S. glabra and C. spicatus |
采取样品的嫩叶,采用本研究室改良的CTAB法提取草珊瑚幼嫩叶片的基因组DNA[9],用1%琼脂糖凝胶电泳检测。然后对所提取的草珊瑚DNA进行纯化。
2.2 ITS序列PCR扩增与测序设计ITS通用引物(ITSF:5’-AGGTGAACCTGCGGAAGATCATTGT-3’;ITSR:5’-GGTGGCCCCG-CCTGACCT-3’)对草珊瑚、金粟兰样品进行PCR扩增,扩增序列的通用反应体系:总体积20 μL,其中包括10×缓冲液2.0 μL,dNTP 20 nmol,2 μmol/L引物各2 μL,Taq酶1 U,DNA 20 ng,灭菌蒸馏水12.8 μL。PCR反应程序:94 ℃预变性3 min后,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,35个循环后72 ℃延伸8 min。获得PCR产物通过1.5%的琼脂糖凝胶电泳,并用EB染色观察。扩增产物回收后,经华大基因公司测序,获得24条ITS序列;并于GenBank数据库中搜索其他金粟兰属植物的ITS序列信息,获得及己(AF280412.1)、宽叶金粟兰(AF280415.1)、四川金粟兰(AF280413.1)、台湾金粟兰(AF280414.1)ITS序列。
2.3 数据统计及分析所有ITS序列用ClustalX 2.1软件对所有序列进行多重比较,并进行UPGMA分析其遗传距离,建立聚类图谱,并用Genedoc软件分析ITS序列变异情况,用Treeview软件分析聚类结果。
3 结果 3.1 序列测定结果所获得ITS序列,根据Genebank中已公布的草珊瑚与金粟兰ITS的序列(AF280408.1、AF280411.1)确定rDNA内转录区间ITS1和ITS2与3个编码区18 S、5.8 S、26 S的界限。测得草珊瑚18个样品和金粟兰属6个样品的ITS1、5.8 S rDNA和ITS2全序列分别为663、658 bp。其中草珊瑚ITS1长度为289 bp,5.8 S rDNA序列长度为163 bp,ITS2序列长度为211 bp;ITS1的(G+C)量在58.13%~59.17%,ITS2的(G+C)量在56.87%~57.15%。金粟兰ITS1长度为290 bp,5.8 S rDNA序列长度为163 bp,ITS2序列长度为205 bp。ITS1的(G+C)量在55.52%~56.21%,ITS2的(G+C)量均为54.63%,见表 1。
3.2 草珊瑚ITS序列变异情况及与其他金粟兰属植物ITS序列鉴别位点分析测序结果显示,ITS1区前100 bp段第34、53、54位点突变的样品有S1、S5、S7 3个样品。利用ClustalX 2.1及Genedoc软件分析得到草珊瑚18个样品ITS序列间的变异情况,见表 2。
| 表 2 草珊瑚样品ITS序列差异 Table 2 Differences in ITS sequence of samples |
Genedoc软件分析显示18个草珊瑚受试样品相互间ITS序列相似度达99%,18个草珊瑚受试样品共有11个变异位点,占663 bp ITS序列1.6%,其中8个位点在ITS1区289 bp占2.7%,3个位点在ITS2区211 bp占1.4%。其中222 bp位点的变异性较大,18个样品中有4个样品有变异,34 bp和634 bp在18个样品中均有2个样品有变异,而其余位点都仅有1个位点的变异。11位点变异中,3个位点为碱基颠换,2个碱基缺失,另外6个位点为碱基转换,受试样品中草珊瑚ITS2序列比ITS1序列稳定,变异位点较少。
同时对草珊瑚与金粟兰属植物的ITS序列变异情况进行分析显示,在ITS1区,草珊瑚与金粟兰、及己、宽叶金粟兰、四川金粟兰、台湾金粟兰的变异位点分别达46、49、47、48、53个,而在ITS2区,分别达47、43、43、48、45个。因此,草珊瑚与其他5种金粟兰属植物在ITS1和ITS2均有较多位点的变异,其中在ITS1区变异位点达46~53个位点,占289 bp ITS1序列的15.9%~18.3%,在ITS2区变异位点达43~48个位点,占211 bp ITS2序列的20.3%~22.7%。除去其中个别草珊瑚不用种群间的变异位点,这些位点都可以用于草
珊瑚与金粟兰属植物的分子鉴别,以上数据表明ITS2提供更多位点将草珊瑚区别于其他5种金粟兰植物。综合以上2方面的数据表明,草珊瑚ITS1序列相对于ITS2更适合于草珊瑚属内不同种群的分子鉴别分析,而ITS2更适合于草珊瑚属与金粟兰属不同植物的分子鉴别分析。
3.3 基于ITS序列的模式识别用ClustalX 2.1软件UPGMA分析法对表 1中的所有序列进行聚类分析,建立聚类图谱,并用Treeview软件分析表 1中所有样品间ITS序列相似性聚类,结果见图 1。所有样品聚为2类,18个来源于不同产地的草珊瑚聚为一类,其他5种金粟兰属植物聚为一类,且其中6个来源不同种群的金粟兰聚为一类,而其他4种金粟兰属植物又聚为一类。由聚类图可见基于ITS序列,来源于不同产地的草珊瑚都可与其他5种金粟兰属植物进行基于ITS序列变异的分子鉴定。
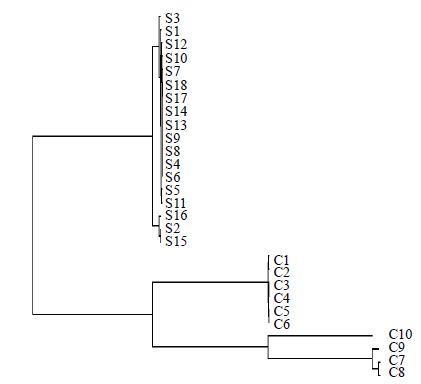 | 图 1 基于ITS的草珊瑚样品与其他5种金粟兰的UPGMA聚类Fig. 1 UPGMA clustering of S. glabra and five plants in Chloranthus Swartz based on ITS |
通过18个来源于不同产地的草珊瑚ITS序列的测序分析表明,ITS序列碱基变异形式有碱基缺失、碱基颠换与碱基转换3种,其中一半以上以碱基转换方式突变。表明草珊瑚的ITS1与ITS2序列的稳定性在受试的18个不同种群中稳定性并不一样,ITS1的变异性比ITS2大,一定程度表明ITS1序列相对于ITS2更适合于草珊瑚属内不同种群的分子鉴别分析。而通过比较草珊瑚与金粟兰属5种植物的ITS序列比较分析表明ITS1序列变异15.9%~18.3%,ITS2序列变异达20.3%~22.7%,为草珊瑚鉴别于金粟兰属其他5种植物提供丰富的位点。而草珊瑚ITS2在种群内更稳定,因此,ITS2更适合于草珊瑚属与金粟兰属不同植物的分子鉴别分析。进一步通过聚类分析,表明18个不同种群的草珊瑚种内变异极小,且与金粟兰属5种植物的种间变异没有交叉,分别聚为有明显差异的2类,一定程度表明基于ITS序列,来源于不同产地的草珊瑚都可与其他5种金粟兰植物进行基于ITS序列变异的分子鉴定。
rDNA-ITS序列是近年来探讨植物种内变异和种间、近缘属间分子系统关系的重要分子标记之一。本研究将该序列用于中药草珊瑚不同种群及与金粟兰属其他5种近缘种的品种鉴定中,通过对草珊瑚、金粟兰属植物序列的变异位点分析,为草珊瑚种内变异及其与金粟兰属其他5种近缘种的分子鉴定提供思路与分子标记。本研究为如何采用ITS序列进行科学有效的对草珊瑚容易混淆的品种进行分子鉴定具有重要的意义,也为其他中药材分子鉴定有借鉴意义。
| [1] | 倪开诚, 闵 芳, 郭卫东, 等. 采用ISSR分子标记进行草珊瑚8个种源的遗传多样性分析 [J]. 中草药, 2008, 39(9): 1392-1396. |
| [2] | 曹聪梅. 中国金粟兰科植物药用亲缘学研究 [D]. 北京: 中国医学科学院北京协和医学院, 2009. |
| [3] | Yang Z Y, Chao Z, Huo K K, et al. ITS Sequence analysis used for molecular identification of the bupleurum species from northwestern China [J]. Phytomedicine, 2007, 14(6): 416-423. |
| [4] | Wang H, Kim M K, Kim Y J, et al. Molecular authentication of the oriental medicines Pericarpium citri reticulatae and Citri unshius pericarpium using SNP markers [J]. Gene, 2012, 494(1): 92-95. |
| [5] | Jigden B, Wang H, Kyum K M, et al. Authentication of the oriental medicinal plant Ligusticum tenuissimum (Nakai) Kitagawa (Korean Go-Bon) by multiplex PCR [J]. Planta Med, 2010, 76(6): 648-651. |
| [6] | Wang H, Kim M K, Kwon W S, et al. Molecular authentication of Panax ginseng and ginseng products using robust SNP markers in ribosomal external transcribed spacer region [J]. J Pharm Biomed Anal, 2011, 55(5): 972-976. |
| [7] | Ding G, Zhang D Z, Feng Z Y, et al. SNP, ARMS and SSH authentication of medicinal Dendrobium officinale Kimura et Migo and application for dentification of Fengdou drugs [J]. Biol Pharm Bull, 2008, 31(4): 553-557. |
| [8] | Zhang F M, Ge S, Chen W L. Phylogeny of the Aconitum delavayi complex (Ranunculaceae) based on evidence from nuclear ribosomal ITS sequences [J]. Acta Phytotaxonomica Sin, 2003, 41(3): 220-228. |
| [9] | 王关林, 方宏筠. 植物基因工程原理与技术 [M]. 北京: 科学出版社, 1998. |
 2014, Vol. 45
2014, Vol. 45



