2. 中国医学科学院 北京协和医学院药用植物研究所海南分所, 海南省南药资源保护与开发重点实验室, 海南 万宁 571533;
3. 山东中医药大学药学院, 山东 济南 250012
2. Hainan Provincial Key Laboratory of Resources Conservation and Development of Southern Medicine Hainan Branch Institute of Medicinal Plant, Chinese Academy of Medical Sciences & Peking Union Medical College, Wanning 571533, China;
3. College of Pharmacy, Shandong Traditional Chinese Medicine University, Jinan 250012, China
白木香Aquilaria sinensis (Lour.) Gilg又称土沉香,属瑞香科沉香属植物,是我国生产名贵芳香类药材沉香的唯一正品植物来源,现已被列为国家二级濒危保护植物。沉香味辛、苦,性微温,具有行气止痛、温中止呕、纳气平喘等功效[1]。白木香是一种典型的伤害诱导型药用植物,只有在受到伤害后才能在其伤口周围木质部产生倍半萜等沉香类物质。研究表明,伤害诱导沉香形成是白木香防御反应的产物[2]。
为研究伤害诱导沉香形成的分子机制,课题组前期对健康和伤害处理的白木香进行了转录组分析,并重点分析了参与倍半萜合成与调控的关键基因;发现外源茉莉酸(JA)处理显著诱导倍半萜合酶基因的表达,经茉莉酸酯(MeJA)处理的愈伤组织中倍半萜成分的含量相对于健康组织升高了70多倍[3]。白木香悬浮细胞经MeJA处理后,可增加5个δ-愈创木烯合酶基因的表达量,同时增加倍半萜δ-愈创木烯的量[4]。此外,白木香树经机械伤害和外施MeJA处理,内源茉莉酸素(JAs)量显著增加,同时产生3种倍半萜物质[5, 6]。这些结果表明,JA信号在白木香诱导结香中扮演重要角色。
大量的研究表明,JA是伤信号转导途径中长距离运输的重要信号分子,依赖于JA作为调节反应信号的防御应答也是植物次生代谢物质积累的前提[7, 8, 9]。NINJA(Novel Interactor of JAZ)是JA信号途径中的负调控因子;在拟南芥中,Pauwels等[10]发现了互作蛋白(jasmonate Zim-domain,JAZ)抑制蛋白与协同抑制因子TOPLESS(TPL)和TPL相关蛋白(TPLs)的结合需要NINJA蛋白的参与,共同调控着JA信号途径相关次生代谢物的合成并达到植物防御的目的。本实验首次报道了白木香NINJA(AsNINJA1)基因cDNA全长的克隆及表达分析,为进一步研究AsNINJA1蛋白的功能及揭示JA信号在白木香倍半萜形成中的作用机制奠定基础。
1 材料与方法 1.1 材料中国医学科学院药用植物研究所温室栽培的3年生白木香茎诱导产生,生长良好且基本一致的白木香愈伤组织,转接到含有100 μmol/L浓度MeJA的MS固体培养基中,25 ℃暗培养,8个不同时间段(0.5、1、2、4、6、8、16、24 h)取样,分别设置3个生物学重复。以同一批生长一致的白木香愈伤组织同时转接到不含MeJA的MS培养基中25 ℃暗培养,分别在相同时间段(0.5、1、2、4、6、8、16、24 h)取样,均设3个重复,作为对照。所有的材料取样后均置于液氮中速冻后,-80 ℃冰箱保存备用。
1.2 试剂植物总RNA提取试剂盒、Fast HiFidelity Polymerase高保真酶、大肠杆菌DH5α感受态、多聚酶链反应(PCR)产物胶回收试剂盒、pZeroBack/blunt零背景连接试剂盒购于天根生化科技(北京)有限公司。pUC-T载体UltraSYBR Mixture购于北京康为世纪生物科技有限公司。SMARTerTM RACE cDNA Amplification Kit试剂盒购于日本宝生物公司产品。RevertAid First Strand cDNA Synthesis Kit购于ThermoFisher SCIENTIFIC公司。其他试剂均为国产分析纯。本研究所用引物由上海生工生物工程股份有限公司合成,产物测序由英潍捷基贸易有限公司完成。
1.3 总RNA的提取和cDNA合成将经MeJA化学处理的白木香愈伤组织材料及未处理的健康白木香愈伤组织材料置于液氮中研磨,依据天根生化科技(北京)有限公司的植物总RNA提取试剂盒说明书提取总RNA,利用NanoDrop 2000进行测定和1%琼脂糖电泳检测。健康的不经MeJA处理不同时间段的白木香愈伤组织混样提取总RNA,利用SMARTerTM RACE cDNA Amplification Kit(Clontech公司)试剂盒合成5′-和3′-RACE cDNA,反转录的条件及程序均按照试剂盒说明书进行,用于AsNINJA1基因全长的克隆。经MeJA化学处理的和健康不经MeJA处理白木香愈伤组织提取总RNA,利用RevertAid First Strand cDNA Synthesis Kit(ThermoFisher SCIENTIFIC公司)反转录成cDNA第一链,作为qRT-PCR的模板。
1.4 AsNINJA1基因全长克隆基于白木香转录组数据库得到AsNINJA1基因的UniGene片段,通过NCBI Blast比对发现该序列并不是全长,运用引物设计软件Primer 5.0分别设计AsNINJA1基因5’-和3’-末端的扩增的特异性引物GSP1、GSP2。以健康的不经MeJA处理不同时间段的白木香愈伤组织混样提取的总RNA反转录得到的RACE cDNA为模板,反应体系和PCR程序按照SMARTerTM RACE cDNA Amplification Kit(Clontech公司)试剂盒说明书进行操作。1%琼脂糖凝胶电泳检测PCR产物,切胶,用天根公司琼脂糖凝胶回收试剂盒回收目的片段。将回收的PCR目的产物与pUC-T载体连接,转化DH5α菌株,在含有氨苄的LB平板上进行培养,挑取单克隆摇菌,经菌液PCR扩增和电泳检测后选择阳性克隆送英潍捷基贸易有限公司测序。将测序结果在NCBI基因预测工具(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)上寻找一个完整的开放阅读框(open reading frame,ORF),通过设计全长引物扩增全长(表 1)。反应体系:快速高产聚合酶0.5 μL,5×快速高产PCR 缓冲液5 μL,dNTP Mixture(2.5 mmol/L)2 μL,NINJA-LF(10 μmol/L)1 μL,NINJA-LR(10 μmol/L)1 μL,cDNA 1.0 μL,ddH2O 14.5 μL,终体系为25 μL。PCR反应程序:94 ℃、5 min;94 ℃、30 s,62 ℃、30 s,72 ℃、80 s,35个循环;72 ℃ 延伸7 min;4 ℃保存。PCR产物回收后用pZeroBack/blunt零背景连接试剂盒进行连接、转化、涂平板,菌液PCR如前。
| 表 1 基因克隆及实时荧光定量PCR检测引物 Table 1 Primers for gene cloning and real-time PCR detection |
AsNINJA1基因编码蛋白的理化性质预测采用ExPASy Proteomics Server提供的在线工具ProtParam(http://web.expasy.org/protparam)和ProtScale(http://web.expasy.org/protscale)进行分析。利用在线工具InterProScan(http://www.ebi.ac.uk/Tools/pfa/iprscan/)对AsNINJA1蛋白结构域进行分析。使用SignalP3.0Server(http://www.cbs.dtu.dk/ services/SignalP/)和TargetP 1.1 Server(http://www.cbs.dtu.dk/services/TargetP/)进行分泌蛋白预测。利用WOLF PSORT(http://wolfpsort.org/)分析亚细胞定位,TRMHMM server v2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0/)进行跨膜域分析。氨基酸序列比对利用NCBI的蛋白质序列数据库进行,通过MEGA 5构建NJ系统进化树,bootstrap 重复次数为1 000次。
1.6 实时荧光定量PCR(qRT-PCR)利用qRT-PCR(美国伯乐公司的iQTM5实时PCR检测系统)的方法检测经MeJA处理的白木香愈伤组织中AsNINJA1基因不同时间段的表达量。反应体系如下:2×UltraSYBR 预混液 20 μL,正反向引物(10 μmol/L)各0.4 μL,cDNA模板0.8 μL,加水至终体积20 μL,每个反应体系设置3个重复。实时PCR反应程序:95 ℃、10 min;95 ℃、15 s,60 ℃、1 min,40个循环;RT-PCR反应以α微管蛋白基因TUA为内参[11],不同时间段基因的表达以健康的不进行MeJA处理的白木香愈伤组织中的表达量为对照。将Real-time PCR仪所得的样品Ct值,利用2-ΔΔCt方法转化为基因相对表达量值。应用Excel软件进行数据录入及表的绘制,采用SPSS16.0软件进行统计与One-way ANOVA方差分析,并用Duncan检验法进行多重比较。
2 结果与分析 2.1 白木香AsNINJA1基因全长的克隆根据前期转录组测序得到的AsNINJA1基因的UniGene序列设计特异引物,通过RACE-PCR技术获得5’端序列和3’端序列克隆测序的结果与已知的序列片段拼接得到全长cDNA序列为1 982 bp。为验证全长的结果,根据拼接后得到的全长序列设计引物,以cDNA为模板,扩增白木香AsNINJA1基因全长序列并测序,与预计的片段大小一致,克隆后测序,结果与拼接结果一致。
从得到的白木香AsNINJA1基因cDNA序列全长,通过NCBI基因预测工具(ORF Finding)获得一个长为1 221 bp完整的开放阅读框(ORF)。5’端存在为525 bp的非翻译区(UTR),3’端的UTR区长为255 bp,其中含为18 bp的poly(A)尾,命名为AsNINJA1基因。图 1为经过PCR扩增得到的AsNINJA1基因编码区(CDS)序列检测电泳图。
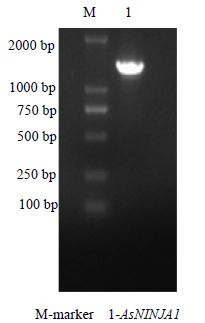 | 图 1 AsNINJA1基因CDS的扩增Fig. 1 CDS amplification of AsNINJA1 gene |
AsNINJA1蛋白的分子式为C1886H2971N553O624S10,编码406个氨基酸,相对分子质量为43 697 。等电点pI为6.02,带负电残基(Asp+Glu)为46,带正电残基为37(图 2)。该蛋白的不稳定系数为47.64,属于不稳定蛋白;脂肪系数为68.45,亲水性系数为-0.617,表明其为疏水性蛋白;摩尔消光系数为24 200,在哺乳动物体内的半衰期为30 h,酵母体内大于20 h,菌内大于10 h。整个AsNINJA1中疏水性最大值是1.678,最小值是-2.589;其中145~155、175~194、306~322和358~367出现强烈的疏水性,其他为亲水性区域,平均亲水系数小于-0.6,此结果表明AsNINJA1可能是一种水合能力较强的蛋白。
2.2.2 信号肽、跨膜区及亚细胞定位预测分析TargetP 1.1 Server预测结果显示,线粒体信号肽(mitochondrial targeting peptide,mTP)为0.051,分泌通路信号肽(secretory pathway signalpeptide,SP)为0.049,其他为0.956。SignalP 4.0 Server预测结果显示AsNINJA1蛋白为非分泌蛋白,不具有信号肽。TRMHMM server v2.0跨膜区预测AsNINJA1编码的蛋白没有跨膜区域,为非膜蛋白。亚细胞定位结果表明该蛋白定位于细胞核中。
2.2.3 结构域预测InterProScan在线工具预测AsNINJA1蛋白保守结构域,共有3个结构域(图 2)。AsNINJA1蛋白的保守结构域主要含Ninja结构域,以及2个未明确分类的保守结构域。Ninja结构域位于331~380(PF07897),是植物响应胁迫过程中JA信号通路的负调控因子NINJA特有的结构域;还有一类保守结构域在IPR中没有明确分类,位于(PTHR31413和PTHR31413:SFO)331~379两个结构域仍未命名。
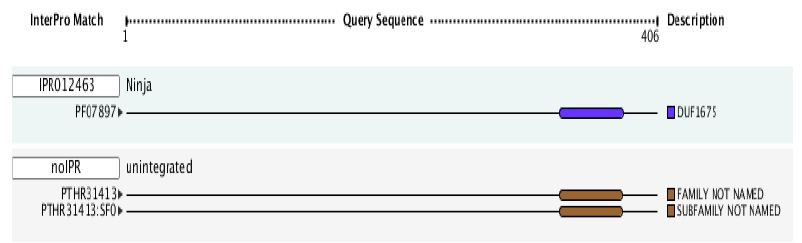 | 图 2 AsNINJA1蛋白保守结构域预测分析Fig. 2 Predicted characteristic conserved domains of AsNINJA1 protein |
通过NCBI在线工具Blast进行数据比对,结合相关文献的查阅,筛选出包括白木香AsNINJA1在内的17个物种,属NINJA家族蛋白的17种蛋白,并下载它们的蛋白质序列(图 3)。使用MEGA5.05进行多序列的比对,采用相邻连接法构建HDR进化树,进行聚类分析。结果如图 4所示,白木香AsNINJA1与可可JAZ互作因子蛋白聚为一类,两者亲缘关系最近,其中与可可的NINJA蛋白关系最近,两蛋白保守结构域序列相似性为88.7%。
 | 图 3 白木香AsNINJA1蛋白与可可NINJA蛋白保守结构域序列相似性比较分析Fig. 3 Comparison on sequence similarity of conservative domains of AsNINJA1 protein in A. sinensis and NINJA protein in cocoa |
 | 图 4 白木香AsNINJA1系统发生树Fig. 4 Phylogenetic tree of AsNINJA1 in A. sinensis |
不同时间段MeJA诱导白木香愈伤组织中AsNINJA1基因相对表达结果如图 5所示。MeJA能明显诱导白木香愈伤组织中AsNINJA1基因mRNA的表达,MeJA处理后0.5 h AsNINJA1基因相对表达量相对于对照提高了40多倍,随后增长量下降,2 h后快速增高直至4 h时达到最大值,高达对照表达量近100倍;24 h后基因的表达量降低至正常水平。AsNINJA1基因相对表达在不同处理时间差异显著,处理4 h与其他各时间段均差异显著。而对照组在不同时间段AsNINJA1基因的相对表达量比较低,并且无明显变化,无显著性差异。
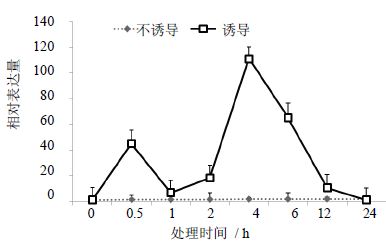 | 图 5 MeJA诱导白木香愈伤组织中AsNINJA1基因的表达Fig. 5 Effect of MeJA on AsNINJA1 gene expression in A. sinensis callus |
植物由于受制于不能移动的特性,长期以来受到来自于环境中生物和非生物的胁迫形成了一套有效的防御系统。近几十年来,植物响应外界胁迫的防御机制的研究一直都是备受关注的热点。沉香资源短缺、价格昂贵,具有伤害后经过较长时间才能产生的特点,因此揭示外界伤害胁迫诱导形成的分子机制对于结香至关重要。
本研究首次从白木香中克隆得到了JA信号途径中的第一个关键蛋白基因AsNINJA1,这也是沉香属植物第一次获得该基因。生物信息学分析表明,该基因编码的氨基酸序列具有典型的NINJA蛋白家族的结构域。因此,推测AsNINJA1蛋白可能作为JA信号通路中的一个负调控因子与JAZ抑制蛋白发生相互作用,共同调控着相关基因的表达。qRT-PCR结果表明,该基因的表达对MeJA诱导的化学伤害敏感,且主要在早期响应伤害胁迫,后期则基本恢复原表达水平。对于MeJA处理后,AsNINJA1基因的表达并未出现一个逐渐增高的趋势,而是在初期0.5 h时出现表达量高于对照40倍的高峰期,随后降低再升高的表达特征,这与前人的研究结果一致。即植物细胞在受到伤害胁迫后,植物细胞膜快速释放出内源JA作为第二信号分子,参与JA相关防御反应并产生相应的次生代谢物[12, 13, 14, 15, 16]。总之,qRT-PCR结果在一定程度上说明AsNINJA1基因属于防御反应早期应答的基因。研究结果为从分子水平上调控AsNINJA1基因表达量及其揭示白木香倍半萜合成分子机制的研究提供了基础数据。但是,AsNINJA1基因究竟在沉香形成的过程发挥着怎样的作用,与信号通路中其他的蛋白如何相互作用共同调控响应JA信号途径相关基因的表达有待更深入的探讨。
| [1] | 中国药典 [S]. 一部. 2010. |
| [2] | 张兴丽. 伤害诱导的白木香防御反应与沉香形成的关系研究 [D]. 北京: 北京林业大学, 2013. |
| [3] | Xu Y H, Zhang Z, Wang M X, et al. Identification of genes related to agarwood formation: transcriptome analysis of healthy and wounded tissues of Aquilaria sinensis [J]. BMC Genomics, 2013, 14: 227. |
| [4] | Kumeta Y, Ito M. Characterization of δ-guaiene synthases from cultured cells of Aquilaria, responsible for the formation of the sesquiterpenes in agarwood [J]. Plant Physiol, 2010, 154: 198-200. |
| [5] | 张 争, 杨 云, 魏建和, 等. 白木香茎中内源茉莉酸类和倍半萜类物质对机械伤害的响应 [J]. 园艺学报, 2013, 40(1): 163-168. |
| [6] | 徐艳红, 杨 欣, 梁 良, 等. 茉莉酸甲酯诱导的白木香cDNA文库的构建及初步鉴定 [J]. 中草药, 2014, 45(1): 102-106. |
| [7] | Glauser G, Dubugnon L, Mousavi S A R, et al. Farmer E E. Velocity estimates for signal propagation leading to systemic jasmonic acid accumulation in wounded Arabidopsis [J]. J Biol Chem, 2009, 284: 34506-34513. |
| [8] | Koo A J, Gao X, Jones A D, et al. A rapid wound signal activates the systemic synthesis of bioactive jasmonates in Arabidopsis [J]. Plant J, 2009, 59: 974-986. |
| [9] | Endt D V, Silva M S, Kijne J W, et al. Identification of a bipartite jasmonate-responsive promoter element in the Catharanthus roseus ORCA3 transcription factor gene that interacts specifically with AT-hook DNA-binding proteins [J]. Plant Physiol, 2007, 144: 1680-1689. |
| [10] | Pauwels L, Barbero G F, Geerinck J, Tilleman S, et al. NINJA connects the co-repressor TOPLESS to jasmonate signaling [J]. Nature, 2010, 464: 788-791. |
| [11] | Gao Z H, Wei J H, Yang Y, et al. Selection and validation of reference genes for studying stress-related agarwood formation of Aquilaria sinensis [J]. Plant Cell Rep, 2012, 31: 1759-1768. |
| [12] | Creelman R A, Teerney M L, Mullet J E. Jasmonoic acid/methyl jasmonate accumulate in wounded soybean hypocotyls and modulate wound gene expression [J]. Proc Natl Acad Sci USA, 1992, 89: 4968-4941. |
| [13] | Farmer E E, Johnson R R, Ryan C A. Regulating of expression of proteinase inhibitor genes by methyl jasmonate and jasmonic acid [J]. Plant Physiol, 1992, 98: 995-1002. |
| [14] | Farmer E E, Ryan C A. Octadecanoid-derived signals in plants [J].Trends Cell Biol, 1992a, 2: 236-241. |
| [15] | Farmer E E, Ryan C A. Octadecanoid precursors of jasmonic acid activate the synthesis of wound-inducible proteinase inhibitors [J]. Plant Cell, 1992b, 4: 129-134. |
| [16] | Albrecht T, Kehlen A, Stahl K, et al. Quantification of rapid transient increases in jasmonic acid in wounded plants using a monoclonal antibody [J]. Planta, 1993, 191: 86-94. |
 2014, Vol. 45
2014, Vol. 45


