2. 吉林农业大学中药材学院, 吉林 长春 130118
2. College of Chinese Medicinal Materials, Jilin Agricultural University, Changchun 130118, China
中药是中国劳动人民在长期的生活实践中积累的宝贵财富。自《神农本草经》至今,经过几千年的发展,在中药知识日益完善的同时,也暴露出一些不可避免的问题。中药药用植物的准确分类和鉴别是研究中药品质以及制定中药质量标准的重要前提。中药传统的鉴定方法主要包括基原鉴定、性状鉴定、显微鉴定和理化鉴定。传统的基原鉴定和性状鉴定需要工作者丰富的理论知识和多年的实践经验,同时受到药用植物表型及遗传变异等多种变量的影响[1],相对于目前已知的药用植物物种而言,明显需要大量的分类学人才才能完全建立中药的识别分类体系,这显然是很大的挑战。中药饮片、粉末以及含有原型生药的传统中成药采用性状鉴定显然不可取,而显微鉴定与理化鉴定对仪器和操作技术要求较高、可重复性较低、操作相对复杂,且近缘物种差异不显著、单一鉴定方法效率较低。因此,迫切需要发展方便、快捷、高效以及易于标准化的物种分析鉴定方法。
DNA条形码(DNA barcoding)在中药药用植物分类上具有明显的优势:1)不受植物样本个体发育的影响。一些植物在生长发育过程中,形态上差异变化会比较大,但DNA序列却不会发生变化。2)相比于传统分类学,DNA条形码分类鉴定技术能够更好地区分形态相似的物种,避免了传统形态分类学易犯的错误。3)依托于逐渐成熟的PCR和更加先进的分子标记技术,DNA条形码能够快速、准确、方便地进行物种的鉴别鉴定。4)DNA条形码技术上手快、操作简单,能够快速建立物种鉴定人才的学科网络,有效缓解现今分类学家紧缺、新的分类学人才较少的现状。
DNA条形码技术的出现为药用植物的快速准确鉴定开辟了一条新的道路。由于不同药用植物的DNA特异性,DNA条形码可以弥补传统分析鉴定方法鉴别过程中存在的多种难题,是林奈发明双名法之后分类学方面最大的进步,被认为是生命研究领域的“大科学计划”[2]。本文综述了DNA条形码在药用植物鉴定中的应用研究,并对常用单位点序列和复合序列的鉴定应用展开了讨论。同时针对近期提出的以叶绿体全基因组作为“超级条形码”的可行性进行了分析讨论和展望,以期为中药药用植物的快速准确鉴定提供新的研究思路。
1 DNA条形码的概念DNA条形码(DNA barcoding)是指通过测定DNA序列信息以鉴别物种或其变异类型的一种新型分子生物学技术。近些年来,随着生物技术的发展,以DNA指纹(DNA fingerprinting)技术为代表的分子水平的标记技术纷纷出现,并被广泛地应用于物种鉴别或其遗传变异方向,成为DNA条形码技术的前身。然而,常用的植物DNA分子标记技术(如RFLP、RAPD、SSR、ISSR等)虽然可以弥补传统形态鉴定学上的一些难点和缺陷,但是却各自存在自身难以克服的缺点,如通用性较低、可重复性差、难以标准化等。
2003年,加拿大学者Hebert在文章中首先提出将DNA条形码技术应用到物种鉴别中[1]。线粒体细胞色素氧化酶亚基1(cytochrome C oxidase subunit 1,CO1)在动物任何分类水平上都具有良好的鉴别能力,进而提出以一段650 bp长的CO1序列作为鉴别动物物种的条形码候选序列。近些年来,国内外学者所得出大量的CO1实验结果表明,CO1具有很高的通用性,能够有效并且高效地鉴别动物物种。
然而对于植物界的分类研究进展相对比较缓慢,主要由于在线粒体基因组方面。植物线粒体基因组因杂交和基因渗入而导致变异较少,进化速率较动物慢,遗传分化较小,所以适用于动物物种鉴别的CO1片段就不适用于植物,同时由于核基因通常具有多拷贝的特性,且种内变异较大,所以很难设计通用引物,并且在扩增时对模板DNA的纯度要求很高。随着对叶绿体基因组的研究不断深入,科学家发现叶绿体基因组序列相对保守且同时具有一定的变异,容易设计通用引物,满足植物条形码的初步要求。此后,植物DNA条形码技术得到进一步的发展[3, 4, 5, 6, 7]。
生物条形码联盟(CBOL)最初推荐作为植物叶绿体DNA条形码候选序列有matK、rpoC1、rpoB、accD、nhdJ和YCF5等,但是由于后3种在一些主要植物中缺失,所以不适合作为通用的植物DNA条形码候选序列[8]。2009年11月,在墨西哥召开的第三届DNA条形码国际学术大会上,与会学者达成共识,一致建议在matK和rbcL这2种目前常用的DNA条形码序列之外,应对ITS和trnH-psbA这2种新的候选序列进行进一步的分析评估。
2 单位点序列 2.1 MatKMatK与其他基因序列相比,进化速率相对较快、长度适中,具有种间差异度较高和碱基转换/颠换数较低等特点[9, 10],缺点在于不同分支类群间很难使用通用引物进行扩增和测序,不同的类群往往需要不同的引物来扩增[11, 12]。Lahaye等[13]应用特定的matK基因引物对1 667个植物材料进行扩增[14],成功率达到100%,单独使用matK对这几种植物物种的鉴别成功率均在90%以上。但是96%的样本是兰科植物[15],而且并没有对种间的姊妹种进行鉴定识别,如果加上姊妹种,可能会降低matK的识别率。
与之相反的是,Fazekas等[3]在其研究中对32个种的92个物种应用matK进行鉴定,成功率仅有56%。Newmaster等[16]在研究中发现matK对肉豆蔻科鉴别成功率只有48.6%。虽然CBOL规定matK为植物DNA条形码序列的几个标准候选序列之一,近些年在寻找该片段的通用引物方面,大量的学者也进行了相关的工作,但是至今仍没能取得理想的结果。因此,matK难以成为通用条形码。
2.2 TrnH-psbATrnH-psbA是叶绿体中的一段基因间隔区,在植物中进化速率较快。其平均长度大多数在450 bp左右,长度适宜,由于两端存在保守序列,所以很容易设计通用引物,且该片段的引物具有高通用性、高扩增成功率的特点[17]。但是该片段不同物种间间隔区的长度或拷贝的变异性较大,序列长度变化区间为296~1 120 bp,另外该序列存在较高比率的插入/缺失现象,较难在大范围取样时进行序列比对[11],导致鉴别非同属的植物物种非常困难[15, 18]。Kress等[19]认为trnH-psbA所分析鉴定的植物物种中有92%具有独特的间隔区序列,符合理想的条形码序列标准,能够进行准确鉴定。同时,该基因片段在内毛楠属植物的鉴别上有70%的识别率[16],对兰科植物的识别率更高达90%[13],但在伞形科独活属和禾本科甜茅属的鉴别中,因变异较小而无法成功鉴别[8, 20]。
尽管如此,作为CBOL推荐的植物DNA条形码候选序列之一,trnH-psbA还是具有很大潜力。如果能够找到合适的数据分析方法,该片段的主要缺点(拥有过多的插入/缺失片段)将可能起到良好的作用,可以增加物种鉴别的信息量,同时提升准确率。
2.3 RbcLRbcL在Genbank中有多达50 000条数据,其具有种间通用、容易扩增、容易比对等特点,被CBOL推荐为植物DNA条形码候选序列之一。Newmaster等[21]通过比对发现其能够鉴别同属85%左右的物种,因此认为rbcL不但可以作为苔藓类植物的DNA条形码候选序列,在其他植物类群中也具有较好的鉴定效率。
但是Kress等[19]认为rbcL序列在有花植物中进化较慢,不适合作为种间水平上的鉴定标记;Lahaye等[13]和Fazekas等[3]发现该片段的物种区分度要小于matK和trnH-psbA,虽然具有较高的扩增率,但是物种鉴别的正确率仅为48%。其主要原因是rbcL的全长较长,为1 400 bp左右,实验中需要4对引物才能获得其全长,但是理想的DNA条形码序列要求的片段长度都较短,所以导致在物种鉴定过程中易扩增但是鉴别率较低。许多研究结果也同样得出rbcL在种间水平的差异并不大,因此不适于用作单独的DNA条形码候选序列,但是可以与其他的序列片段相组合进行鉴定。
2.4 ITS序列ITS序列是核糖体中的一段DNA片段,包括ITS1、ITS2和5.8 S 3个常用的DNA序列片段,广泛存在于真菌和可进行光合作用的真核生物当中,具有很高的种间差异性[22]。
在早期研究中,ITS1的识别效果好于ITS2[15, 23],具有81.5%的正确识别率,但在陆生植物中的扩增效率较差,仅为60.4%[15]。因此,CBOL只推荐将ITS序列作为辅助DNA条形码用于植物物种鉴定[8]。5.8 S片段相关研究很少。
ITS2基因序列位置介于ITS1和5.8 S之间,具有片段短、易扩增、易测序等特点[24, 25],更有学者指出,ITS2对于DNA已经降解的药材也有一定效用[26]。Chen等[27]基于6 600余个药用植物样本对常用的7个条形码候选序列(matK、rbcL、trnH-psbA、rpoC1、ycf5、ITS1、ITS2)进行对比研究,结果表明ITS2序列的种间准确鉴定率最高,达到92.7%。因此正式提出将ITS2作为药用植物的DNA条形码候选序列。随后的研究表明ITS2能够很好地鉴别芸香科、蔷薇科、豆科、菊科、大戟科、黄芩属、重楼属、鼠尾草属等多个科属物种[28, 29, 30, 31, 32, 33, 34, 35]。
尽管ITS2有着相对较高的测序准确率,但是由于其存在多拷贝的特性,对同属的物种进行鉴别时,序列容易发生基因重叠现象,进而造成鉴定效率下降。因此,作为ITS序列的代表,ITS2基因序列仍然未能很好地解决药用植物DNA条形码通用性的问题。
2.5 其他除了以上提到的条形码候选序列,目前也有将rpoB、rpoC1、atpF-atpH、psbK-psbI、ycf5以及trnL用于植物物种鉴别的报道[36, 37],但是由于没有足够的种间变异性而无法适用于多个不同的物种。
单位点序列除了变异性、扩增效率受到限制外,基因的缺失也是单位点序列不能作为通用条形码的一个重要原因,如藻类因不含matK序列而无法应用单一的DNA条形码进行鉴定。DNA条形码提倡应用一条简单的序列对所有物种进行鉴定,但是基于单位点序列自身特点的限制,采用多序列或多位点进行物种鉴定被越来越多的研究人员所接受。
3 复合序列植物中单基因位点片段识别率较低[3, 11, 13, 15, 19, 38, 39],很难像CO1在动物界那样得到广泛应用,尤其难以区分杂交品种或者有基因渗透的物种。因此,在筛选基因作为植物DNA条形码候选序列时,不应该只关注单片段序列,还可以考虑多片段组合。2007年9月,在中国台湾举行的第二次国际生命条形码大会上,与会者经过讨论,提出了多种片段组合,以期能够弥补单位点序列的不足。
2009年,CBOL植物工作组通过对7种最常用的单位点序列从通用性、序列质量和鉴定效率等方面进行综合评估,推荐以matK+rbcL组合作为植物通用条形码复合序列,对397个不同科属的样本鉴定效率为72%[8]。
不同的植物类群间进化速率差别较大,而多基因片段组合方案给植物DNA条形码鉴定研究指出了一条新的方向,理想的片段组合应该能够检测出多重水平的种间差异[16]。近几年各国学者对组合片段进行了一部分研究,但现有的结果仍不能满足条形码所规定的标准,仍需要大量的实验结果提供更加充足的证据证实其物种鉴别能力。
3.1 RpoC1+rpoB+matK或RpoC1+matk+trnH-psbA该组合由Chase在2007年提出[11],目的是结合保守的rpoC1和rpoB易设计通用引物、扩增效率高的特点和高度变异的matK或trnH-psbA的高识别性、高鉴定成功率。
3.2 RbcL+trnH-psbAKress等[15]在2007年提出此组合片段的设想,准确地说采用的是rbcL中的一小段基因rbcL-a。通过实验对比发现,单独采用rbcL-a在种水平上的识别率为76.3%,trnH-psbA的识别率为83%,两者结合之后可达到95%,因此推荐其组合片段作为植物DNA条形码的候选序列。
3.3 MatK+atpF-atpH+psbK-psbI或MatK+atpF-atpH+trnH-psbAPennisi等[21]同样在2007年提出了matK+atpF-atpH+psbK-psbI或matK+atpF-atpH+trnH-psbA组合片段,采用UPGMA聚类法,物种单系性分辨率分别为93.1%和89.3%。
Fazekas等[3]分析了上述5种不同的组合片段,发现rpoC1+rpoB+matK或rpoC1+matk+trnH-psbA组合的成功识别率仅61%;rbcL+trnH-psbA组合的成功识别率为64%;matK+atpF-atpH+psbK-psbI或matK+atpF-atpH+trnH-psbA组合单独使用时成功鉴别率仅为45%和44%,联用了几个片段之后成功率达到69%,是上述5种片段组合成功率最高的一种。
复合序列作为替代单基因序列的鉴定序列,有着更广泛的适用性和更高的鉴别效率。组合片段序列一般由一段进化速率较快的序列搭配一段进化速率较慢的序列,但目前多数研究都是直接将几个序列拼接成一个新的组合,这样可能会严重影响对于鉴定物种的准确性。基因间区序列变异率较高,有利于提高鉴定效率,如trnH-psbA等,但不同物种间的基因间区大小差异较大,有些物种甚至会出现“负间区”,即2个待测序列片段重叠而导致间区消失。另外,因不同科属间基因排序可能不同,甚至存在基因缺失,很难设计通用引物并扩增到共有序列。所以在大范围的物种鉴别中,基因间区序列的获得及比对是影响条形码鉴定的一个重要因素。不同的植物类群间进化速率差别较大,理想的片段组合应该能够检测出多重水平的种间差异[16]。由于还缺少相当的样本进行分析,组合片段的可行性仍然值得商榷。CBOL在进行常用7个不同单序列组合评价时也发现,简单的序列组合并不能显著提高物种鉴定效率[8]。
4 药用植物DNA条形码发展趋势探讨自DNA条形码这一概念提出以来,其技术发展日益成熟,能够更加客观、精确、简便地对物种进行鉴定,可以一次性进行大量的样本鉴定,综合成本较低,而且不需要多年的形态学鉴定经验,操作简便、可重复、易于标准化,形成了对传统形态鉴定学一个强有力的补充,得到了许多专家学者的支持。但目前DNA条形码也遇到了发展瓶颈,尽管在科属较集中时鉴别效率较高(约为90%),但是在广泛性选择物种时,鉴定成功率显著下降,尤其是近缘物种的准确鉴定,迄今没能找到通用的DNA条形码序列,导致部分专家对于DNA条形码在物种鉴定上的能力持怀疑或反对态度,认为应用DNA条形码技术会将传承已久的分类学鉴定方法还原为类型学鉴定方法,这样是一种倒退[40, 41]。
而相比于传统的DNA条形码,新近提出的以叶绿体全基因组作为条形码候选序列的“超级条形码”具有更高的鉴定效率和准确性,因而受到越来越多专家学者的关注。
基于DNA条形码自身的限制,在植物鉴定领域可能永远也不可能找到动物中CO1这样的鉴定位点,因此需要用更高效的序列来鉴别突变率较低的物种[42]。2008年在加拿大英属哥伦比亚大学召开的植物无国界(Botany without Borders)会议上,与会者指出叶绿体全基因组有着能和动物CO1序列相媲美的信息量,同时由于大规模平行测序技术(MPS)的发展,能够将叶绿体全基因组作为DNA条形码序列应用在物种鉴别上,并将其称之为“超级条形码”[43],基于叶绿体基因组的物种鉴定受到鉴定学家的逐步认可。Sucher等[44]在近缘药用植物的鉴别中提出,基于基因组的“指纹”图谱可以在种及种群的水平上对植物物种的个体进行鉴别,在相同性质的样本及伪品的检测中具有优势。相比于其他核糖体DNA序列,叶绿体DNA序列更具有“天生”的优势:叶绿体全基因组广泛存在于植物当中,避免了单基因序列带来的个别物种序列缺失的问题,同时也能够有效地解决普通DNA条形码在PCR扩增时的诸多问题[45]。其基因序列的平均长度为1.1×105~1.6×105 bp,远超常用DNA条形码300~700 bp的长度,不仅具有很高的保守性,其包含的物种进化的信息量也远大于常用DNA条形码序列,能够体现足够的种间差异,因此能够有更好的鉴别效率和准确度[46, 47, 48, 49, 50]。
1986年,Shinozaki等[51]完成了烟草和地钱的叶绿体全基因组序列测定,第一次完成了单一物种的叶绿体全基因组测序。但是由于当时的技术落后,叶绿体全基因组测序成本很高,效率很低。因此,直到2010年,NBCI上的叶绿体全基因组序列的个数还不足200,仍不足以作为物种鉴别的数据库进行使用。
Parks等[52]对松属的37个样本进行叶绿体基因组测序,除了在低分类水平上检验了叶绿体基因组的系统发育及种群遗传学研究上的能力,提出叶绿体基因组可以作为种水平上的植物条形码。Nock等[53]以总DNA为模板进行测序,以近缘同属物种叶绿体序列为参考拼接,获得了叶绿体基因组全序列,由此可以在有参考近缘物种序列时降低测序成本,并可以应用于近缘物种的鉴定。Doorduin等[54]成功对菊科植物Jacobaea vulgaris的17个个体以叶绿体基因组作为“条形码”进行鉴别分类,这是世界首次应用一条叶绿体基因序列鉴别一个物种内的多个不同样本,这证明了以测定全叶绿体基因组序列进行物种鉴别的“超级条形码”有作为所有植物物种公用“条形码”的潜力。
Kane等[55]认为基于叶绿体基因组的“超级条形码”在种下分类水平可以进行物种的鉴别,尤其是在近缘物种和不同基因型个体的区分上表现出极强的判别能力。采用序列差异进行物种的鉴别是生物鉴定学家最常用的经典方法,但Hebert等[56]认为物种鉴别最简捷的方法是根据一个基因在2个物种中是否存在进行判断。不同物种的质体基因组在基因种类上是不同的,相对目前常用的条形码序列,这是叶绿体基因组可以成为通用的植物“超级条形码”的一个明显优势。另外,不同物种的基因排列顺序也不尽相同,利用基因排序的差异进行物种鉴定也引起了研究人员的兴趣[57, 58]。
目前,“超级条形码”还具有较多的局限和挑战。随着第2代测序技术(NGS)广泛应用于叶绿体基因组测序,高纯度的叶绿体DNA成为成功测序的必要前提。虽然Diekmann等[59]提出了一套较为标准的叶绿体DNA提取方法,但是对于样本的新鲜程度和浓度梯度溶液提出了较高的要求。基于目前的条件,较难获得可用于测序的高纯度叶绿体DNA。
Roche/454、Illumina及Ion Torrent等测序技术的发展和成熟直接降低了植物叶绿体基因组的测序成本,但是全基因组测序成本仍然高于目前传统条形码技术。较高的成本直接导致叶绿体全基因组数据库的不健全。截至2013年12月,只报道了410条植物物种的叶绿体基因组序列(图 1)。
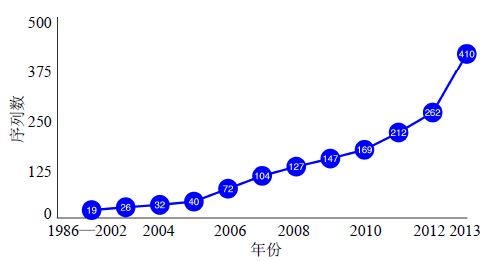 | 图 1 1986—2013年Genbank中叶绿体全基因组总数Fig. 1 Total number of complete chloroplast genome sequences submitted to Genbank from 1986 to 2013 |
尽管如此,相比传统条形码技术的种种不足,对于“超级条形码”未来良好发展前景仍为开展中药药用植物鉴定提供了一个新的思路。
5 结语综合目前研究状况而言,不同于DNA条形码在动物界的出色表现,在整个植物界,找到一个基因序列或者一段较短的DNA片段用以鉴别区分所有药用植物物种几乎是不可能的。因此寻找相应的替代方法进行物种鉴定就成为必然。DNA条形码技术已经走到了一个十字路口,如果不能尽快找出通用的条形码序列或寻求到一种完善的解决不同物种所需条形码候选序列的方式(如与其他学科专业相结合进行分析研究),那么应用DNA条形码作为药用植物物种鉴定的手段与方法必将在专家学者的争议声中迎来寒冬。随着测序技术的发展以及测序成本的降低,有关叶绿体全基因组序列测定的报道已经初见报端,以全叶绿体基因序列进行测序并分析以寻找适合作为药用植物鉴定的DNA条形码候选序列和以全长叶绿体基因组序列作为“超级条形码”将成为“后DNA条形码时代”的研究重点。
| [1] | Hebert P D, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes [J]. Proceed Royal Soc London Series B: Biol Sci, 2003, 270(1512): 313-321. |
| [2] | Gregory T R. DNA barcoding does not compete with taxonomy [J]. Nature, 2005, 434(7037): 1067. |
| [3] | Fazekas A J, Burgess K S, Kesanakurti P R, et al. Multiple multilocus DNA barcodes from the plastid genome discriminate plant species equally well [J]. PLoS One, 2008, 3(7): e2802. |
| [4] | Hebert P D, Penton E H, Burns J M, et al. Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterfly astraptes fulgerator [J]. Proc Natl Acad Sci USA, 2004, 101(41): 14812-14817. |
| [5] | 陈 念, 赵树进. 入侵种的DNA条形码鉴定 [J]. 生物技术通讯, 2009, 20(1): 135-137. |
| [6] | 陈士林, 宋经元, 姚 辉, 等. 药用植物DNA条形码鉴定策略及关键技术分析 [J]. 中国天然药物, 2009, 7(5): 322-327. |
| [7] | 陈 念, 付晓燕, 赵树进, 等. DNA条形码: 物种分类和鉴定技术 [J]. 生物技术通讯, 2008, 19(4): 629-631. |
| [8] | CBOL Plant Working Group. A DNA barcode for land plants [J]. Proc Natl Acad Sci USA, 2009, 106(31): 12794-12797. |
| [9] | Min X J, Hickey D A. Assessing the effect of varying sequence length on DNA barcoding of fungi [J]. Molec Ecol Notes, 2007, 7(3): 365-373. |
| [10] | Selvaraj D, Sarma R K, Sathishkumar R. Phylogenetic analysis of chloroplast matK gene from Zingiberaceae for plant DNA barcoding [J]. Bioinformation, 2008, 3(1): 24-27. |
| [11] | Chase M W, Cowan R S, Hollingsworth P M, et al. A proposal for a standardised protocol to barcode all land plants [J]. Taxon, 2007, 56(2): 295-299. |
| [12] | Hollingsworth P M. DNA barcoding plants in biodiversity hot spots: Progress and outstanding questions [J]. Heredity, 2008, 101(1): 1-2. |
| [13] | Lahaye R, Van Der Bank M, Bogarin D, et al. DNA barcoding the floras of biodiversity hotspots [J]. Proc Natl Acad Sci USA, 2008, 105(8): 2923-2928. |
| [14] | Cu Noud P, Savolainen V, Chatrou L W, et al. Molecular phylogenetics of Caryophyllales based on nuclear 18S rDNA and plastid rbcL, atpB, and matK DNA sequences [J]. Am J Bot, 2002, 89(1): 132-144. |
| [15] | Kress W J, Erickson D L. A two-locus global DNA barcode for land plants: The coding rbcL gene complements the non-coding trnH-psbA spacer region [J]. PLoS One, 2007, 2(6): e508. |
| [16] | Newmaster S G, Fazekas A J, Steeves R A D, et al. Testing candidate plant barcode regions in the Myristicaceae [J]. Mol Ecol Resour, 2008, 8(3): 480-490. |
| [17] | Shaw J, Lickey E B, Beck J T, et al. The tortoise and the hare II: Relative utility of 21 noncoding chloroplast DNA sequences for phylogenetic analysis [J]. Am J Bot, 2005, 92(1): 142-166. |
| [18] | Baldwin B G, Sanderson M J, Porter J M, et al. The ITS region of nuclear ribosomal DNA: a valuable source of evidence on angiosperm phylogeny [J]. Ann Mo Bot Gard, 1995, 82(2): 247-277. |
| [19] | Kress W J, Wurdack K J, Zimmer E A, et al. Use of DNA barcodes to identify flowering plants [J]. Proc Natl Acad Sci USA, 2005, 102(23): 8369-8374. |
| [20] | Pennisi E. Wanted: A barcode for plants [J]. Science, 2007, 318(5848): 190-191. |
| [21] | Newmaster S G, Fazekas A J, Ragupathy S. DNA barcoding in land plants: evaluation of rbcL in a multigene tiered approach [J]. Can J Bot, 2006, 84(3): 335-341. |
| [22] | Álvarez I, Wendel J F. Ribosomal ITS sequences and plant phylogenetic inference [J]. Mol Phylogenet Evol, 2003, 29(3): 417-434. |
| [23] | Chase M W, Salamin N, Wilkinson M, et al. Land plants and DNA barcodes: short-term and long-term goals [J]. Philos T Roy Soc B, 2005, 360(1462): 1889-1895. |
| [24] | Coleman A W. ITS2 is a double-edged tool for eukaryote evolutionary comparisons [J]. Trends Genet, 2003, 19(7): 370-375. |
| [25] | Coleman A W. Pan-eukaryote ITS2 homologies revealed by RNA secondary structure [J]. Nucleic Acids Res, 2007, 35(10): 3322-3329. |
| [26] | Chiou S J, Yen J H, Fang C L, et al. Authentication of medicinal herbs using PCR-Amplified ITS2 with specific primers [J]. Planta Med, 2007, 73(13): 1421-1426. |
| [27] | Chen S L, Yao H, Han J P, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species [J]. PLoS One, 2010, 5(1): e8613. |
| [28] | 罗 焜, 陈士林, 陈科力, 等. 基于芸香科的植物通用DNA条形码研究 [J]. 中国科学: 生命科学, 2010(4): 342-351. |
| [29] | Pang X H, Song J Y, Zhu Y J, et al. Applying plant DNA barcodes for Rosaceae species identification [J]. Cladistics, 2011, 27(2): 165-170. |
| [30] | Gao T, Yao H, Song J Y, et al. Identification of medicinal plants in the family Fabaceae using a potential DNA barcode ITS2 [J]. J Ethnopharmacol, 2010, 130(1): 116-121. |
| [31] | 朱英杰, 陈士林, 姚 辉, 等. 重楼属药用植物DNA条形码鉴定研究 [J]. 药学学报, 2010, 45(3): 376-382. |
| [32] | Gao T, Yao H, Song J Y, et al. Evaluating the feasibility of using candidate DNA barcodes in discriminating species of the large Asteraceae family [J]. BMC Evol Biol, 2010, 10(1): 324-327. |
| [33] | Pang X H, Song J Y, Zhu Y J, et al. Using DNA barcoding to identify species within Euphorbiaceae [J]. Planta Med, 2010, 76(15): 1784-1786. |
| [34] | 夏 至, 冯翠元, 高致明, 等. 黄芩及其同属近缘种的DNA条形码鉴定研究 [J]. 中草药, 2014, 45(1): 107-112. |
| [35] | 刘美子, 刘 萍, 李美妮, 等. 党参及其易混伪品的ITS2分子鉴定 [J]. 世界科学技术—中医药现代化, 2011, 13(2): 412-416. |
| [36] | Han J P, Liu C, Li M H, et al. Relationship between DNA barcoding and chemical classification of Salvia medicinal herbs [J]. Chin Herbal Med, 2010, 2(1): 16-29. |
| [37] | Hollingsworth P M, Graham S W, Little D P. Choosing and using a plant DNA barcode [J]. PLoS One, 2011, 6(5): e19254. |
| [38] | Vijayan K, Tsou C H. DNA barcoding in plants: taxonomy in a new perspective [J]. Curr Sci India, 2010, 99(11): 1530-1541. |
| [39] | Newmaster S G, Fazekas A J, Ragupathy S. DNA barcoding in land plants: evaluation of rbcL in a multigene tiered approach [J]. Can J Bot, 2006, 84(3): 335-341. |
| [40] | Sass C, Little D P, Stevenson D W, et al. DNA barcoding in the Cycadales: Testing the potential of proposed barcoding markers for species identification of Cycads [J]. PLoS One, 2007, 2(11): e1154. |
| [41] | Will K W, Mishler B D, Wheeler Q D. The perils of DNA barcoding and the need for integrative taxonomy [J]. Systematic Biol, 2005, 54(5): 844-851. |
| [42] | Heinze B. A database of PCR primers for the chloroplast genomes of higher plants [J]. Plant Methods, 2007, 3(1): 4-10. |
| [43] | Kane N C, Cronk Q. Botany without borders: barcoding in focus [J]. Mol Ecol, 2008, 17(24): 5175-5176. |
| [44] | Sucher N J, Carles M C. Genome-based approaches to the authentication of medicinal plants [J]. Planta Med, 2008, 74(6): 603-623. |
| [45] | Huang C Y, Grunheit N, Ahmadinejad N, et al. Mutational decay and age of chloroplast and mitochondrial genomes transferred recently to angiosperm nuclear chromosomes [J]. Plant Physiol, 2005, 138(3): 1723-1733. |
| [46] | Shendure J, Ji H L. Next-generation DNA sequencing [J]. Nat Biotechnol, 2008, 26(10): 1135-1145. |
| [47] | Wolfe K H, Li W H, Sharp P M. Rates of nucleotide substitution vary greatly among plant mitochondrial. chloroplast, and nuclear dnas [J]. Proc Natl Acad Sci USA, 1987, 84(24): 9054-9058. |
| [48] | Diekmann K, Hodkinson T R, Wolfe K H, et al. Complete chloroplast genome sequence of a major allogamous forage species, perennial ryegrass (Lolium perenne L.) [J]. DNA Res, 2009, 16(3): 165-176. |
| [49] | Kumar S, Hahn F M, Mcmahan C M, et al. Comparative analysis of the complete sequence of the plastid genome of Parthenium argentatum and identification of DNA barcodes to differentiate Parthenium species and lines [J]. BMC Plant Biol, 2009, 9(1): 131. |
| [50] | Wu F H, Chan M T, Liao D C, et al. Complete chloroplast genome of oncidium gower ramsey and evaluation of molecular markers for identification and breeding in Oncidiinae [J]. BMC Plant Biol, 2010, 10(1): 68-80. |
| [51] | Shinozaki K, Ohme M, Tanaka M, et al. The complete nucleotide-sequence of the tobacco chloroplast genome- its gene organization and expression [J]. EMBO J, 1986, 5(9): 2043-2049. |
| [52] | Parks M, Cronn R, Liston A. Increasing phylogenetic resolution at low taxonomic levels using massively parallel sequencing of chloroplast genomes [J]. BMC Biol, 2009, 7(1): 84-90. |
| [53] | Nock C J, Waters D L E, Edwards M A, et al. Chloroplast genome sequences from total DNA for plant identification [J]. Plant Biotechnol J, 2011, 9(3): 328-333. |
| [54] | Doorduin L, Gravendeel B, Lammers Y, et al. The Complete chloroplast genome of 17 individuals of pest species Jacobaea vulgaris: SNPs, microsatellites and barcoding markers for population and phylogenetic studies [J]. DNA Res, 2011, 18(2): 93-105. |
| [55] | Kane N, Sveinsson S, Dempewolf H, et al. Ultra-barcoding in Cacao (Theobroma spp.; Malvaceae) using whole chloroplast genomes and nuclear ribosomal DNA [J]. Am J Bot, 2012, 99(2): 320-329. |
| [56] | Hebert P D N, Penton E H, Burns J M, et al. Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterfly Astraptes fulgerator [J]. Proc Natl Acad Sci USA, 2004, 101(41): 14812-14817. |
| [57] | Luo H W, Shi J, Arndt W, et al. Gene order phylogeny of the genus prochlorococcus [J]. PLoS One, 2008, 3(12): e3837. |
| [58] | Luo H W, Sun Z Y, Arndt W, et al. Gene order phylogeny and the evolution of methanogens [J]. PLoS One, 2009, 4(6): e6069. |
| [59] | Diekmann K, Hodkinson T R, Fricke E, et al. An optimized chloroplast DNA extraction protocol for grasses (Poaceae) proves suitable for whole plastid genome sequencing and SNP detection [J]. PLoS One, 2008, 3(7): e2813. |
 2014, Vol. 45
2014, Vol. 45


