野三七Panax vienamensis var. fuscidiscus K. Komatsu,S. Zhu & S. Q. Cai是越南人参P. vietnamensis Ha et Grushv. 的一个变种,为五加科人参属植物,主要分布于我国云南省南部的红河州以及老挝等国家。最早由Zhu等[1]在云南省金平县境内发现并命名,具有活血、化瘀和止血等功效,用于治疗肿痛、伤痛和月经不调等[2]。野三七含有大量的奥克梯隆型皂苷珠子参皂苷R2[3-4],以及三七皂苷R1,人参皂苷Rg1、Rb1、Rd和Re等[5,6,7,8,9,10]。近年来研究发现,野三七也分布在红河州的元阳县、绿春县等地。民间也有从老挝等国家高价购买野三七销售,或者作为种源人工种植。一些野三七植株生长达15年以上,高温和多雨的环境下表现出明显的抗病性。因此,野三七是三七P. notoginseng (Burk.) F. H. Chen抗病育种的优良野生种质资源之一。
DNA分子标记是鉴定种质资源遗传多样性的重要手段,也是一种重要的辅助育种方法[11]。简单重复序列(simple sequence repeats,SSR),一般以1~6个碱基为核心序列,具有高度多态性。目前已利用SSR标记构建了许多物种的染色体遗传图谱,并被广泛应用于基因定位及亲缘关系分析、品种鉴定和动植物育种等领域[12]。本研究分析了野三七转录组中的SSR位点,并随机验证了SSR引物的有效性,以期为野三七分子标记辅助育种提供有力工具。
1 材料与方法 1.1 野三七转录组测序与SSR的筛选野三七根和叶片RNA混合样品的转录组测序由北京博奥公司进行,共获得126 758条unigenes。利用est_timmer.perl程序过滤unigenes,参数为:-amb=2,50;-tr5=T,5,50;-tr3=A,5,50;-cut=100,700;再利用CD_HIT去除冗余序列,参数为:-c 1.00 -n 5 -M 2 000。利用misa.pl搜索和定位SSR,搜索的标准为重复单元长度2~6 bp,二核苷酸至少重复6次,三核苷酸至少重复5次,四至六核苷酸至少重复4次;同时筛选被间隔小于或等于100 bp碱基间隔的复合型 SSR。单核苷酸重复容易发生错配而测序失败,所以没有选择。
1.2 SSR引物设计调用Primer 3.0程序对含有SSR位点的两端序列设计引物,引物设计标准:退火温度(Tm)在55~64 ℃,上、下游引物的Tm值相差不大于5 ℃;引物长度在18~27 bp;GC量在40%~60%;尽量避免出现引物二级结构如发卡结构、二聚体、错配和引物二聚体。为验证SSR引物是否可用,随机选择合成30对引物(昆明毓秀生物科技有限公司),进行PCR扩增。
1.3 野三七DNA提取与PCR扩增13株采自不同产地的野三七(表 1),按照Zhu等[1]的方法对其形态特征进行鉴定。野三七的典型特征是无托叶、无苞片,成熟果实时为红色,顶端有一黑色斑点。提取叶片DNA(北京百泰克生物技术有限公司DNA提取试剂盒)。20 μL PCR扩增体系包含2.5 mmol/L dNTPs 1 μL,EasyTaq DNA polymerase 1μL,10×EasyTaq 缓冲液 2 μL,10 μmol/L的上游和下游引物各1 μL,10 ng/μL DNA 1 μL,ddH2O 13 μL。PCR扩增程序为94 ℃预变性3 min后35个循环,每个循环包含94 ℃变性30 s,最佳Tm退火1 min,72 ℃延伸1 min。循环结束后再72 ℃延伸5 min。扩增产物用8%非变性聚丙烯酰胺凝胶检测。
|
|
表 1 不同来源的野三七植株 Table 1 PVF plants collected from different habitats |
采用人工读带的方法,将电泳图上可重复的、清晰的条带计为“1”,同一位置无带或不易分辨的弱带计为“0”,建立原始数据矩阵。利用软件NTsys 2.10e和Popgene 32计算每对引物扩增位点的等位位点数、多态性信息量、遗传距离,按UPGMA进行聚类绘图。
2 结果与分析 2.1 野三七转录组SSR位点数量与分布频率利用MISA工具搜索野三七转录组的126 758条unigenes序列,共在17 780条unigene中找到21 320个符合条件的SSRs,发生频率(含SSR的unigene数与总unigene数之比)为14.03%。14 862条unigene含有单个SSR位点,2 918条unigene(16.41%)含有一个以上SSR位点,1 207条unigene含有复合SSRs。野三七转录组中SSR位点的出现频率(SSR数目与总unigene的数目比值)为16.82%。平均每10 kb有1.29个SSR位点(表 2)。
|
|
表 2 野三七转录组中SSR搜索结果 Table 2 Searching results of SSR in transcriptome of PVF |
野三七转录组SSR种类较为丰富,从重复次数看,发现重复基元以重复6次出现的频率最高,有5 657个,占总SSR的26.53%. 其次为4、5、7次重复,分别占总SSR数量的15.74%、18.71%和16.26%(表 3)。
|
|
表 3 野三七转录组中不同基序长度和重复的SSR数量分布 Table 3 Distribution of SSRs with different motif types and repeat numbers in transcriptome of PVF |
从野三七SSR核苷酸基序类型来看(图 1),SSR以二核苷酸AG/CT和AT/AT重复基序为主要类型,占总SSR的46.25%。其次是三、四核苷酸重复基序,其出现频率分别为28.08%和11.03%,以4~7次重复为主。三核苷酸重复序列AAG/CTT最多,ACT/AGT重复序列最少;四核苷酸重复序列AAAT/ATTT为最多,AAAG/CTTT次之,ACAT/ATGT为最少。
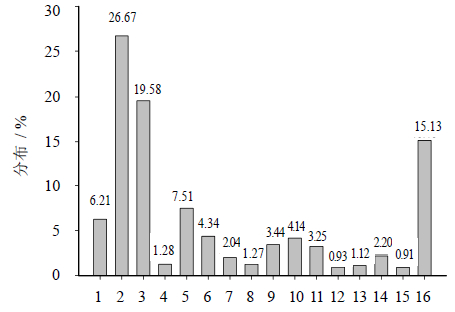 | 1-AC/GT 2-AG/CT 3-AT/AT 4-AAC/GTT 5-AAG/CTT 6-AAT/ATT 7-ACC/GGT 8-ACT/AGT 9-AGC/CTG 10-AGG/CCT 11-ATC/ATG 12-AAAC/GTTT 13-AAAG/CTTT 14-AAAT/ATTT 15-ACAT/ATGT 16-其他图 1 野三七转录组中SSR基序类型分布 Fig. 1 Distribution of SSR type in transcriptome of PVF |
SSR标记多态性是判断其可用性的重要依据。SSR长度是影响其多态性高低的重要因素,当SSR长度大于或等于20 bp时多态性较高,长度在12~20 bp的SSR多态性中等,而长度在12 bp以下时多态性极低[13]。本研究SSR筛选过程中已将多态潜能低于12 bp以下的SSR过滤去掉。野三七转录组中,长度为12~20 bp的SSR最多,共有16 510个,占非复合型SSR总数(20 113个)的82.09%,其次是长度为21~30 bp,共2 140个(11.98%)(图 2)。
 | 图 2 野三七转录组中SSR长度分布 Fig. 2 Distribution of SSR length in transcriptome of PVF |
利用Primer3共设计出39 336对SSR引物。为了验证其引物的有效性,随机挑选30对引物对13株不同来源的野三七基因组DNA进行SSR-PCR扩增。扩增结果表明,除1对引物没有扩增产物外,其余29对(96.67%)引物均能扩增出可重复的、清晰的条带。其中24对(80.00%)PCR扩增产物与预期大小复合,有4对(13.33%)扩增产物长度超过预期,1对引物(3.33%)扩增产物小于预期。
15对(50.00%)表现出多态性差异(表 4),多态性信息量(PIC)范围为0.29~0.50,平均值为0.44。利用SSR多态性对13个野三七材料进行聚类分析,13个野三七材料被聚成2类(图 3),一类是购买自老挝,另一类是采自云南省红河州的金平县、元阳县,说明老挝的野三七种质和国内种质遗传距离较远。
|
|
表 4 SSR-PCR筛选引物 Table 4 Filtering primers of SSR-PCR |
 | 图 3 野三七的UPGMA聚类图 Fig. 3 Dendrogram for tested PVF plants by UPGMA |
野三七转录组中存在丰富的SSR位点。本研究从野三七转录组中共搜索出21 320个SSR,分布于17 780条unigenes。SSR出现频率为16.86%,高于玉米(1.5%)、水稻(4.7%)、大麦(3.4%)、高粱(3.6%)和小麦(3.2%)等作物[14],这种差异可能与SSR搜索标准、数据库大小和物种等有关。
多数植物EST-SSR以二核苷酸和三核苷酸重复为主,但主要的重复基元不同。野三七转录组SSR重复基元以二核苷酸为主,占所有SSRs的52.52%,其次是三核苷酸重复基元。这与大豆、番茄、棉花、杨树、拟南芥、大麦等作物不同,这些植物中SSR的主要类型是三核苷酸重复[15,16]。GC重复基元在多数植物中很难见到,出现频率都很低[17,18,19]。本研究也发现,野三七转录组中,SSR位点最多的是二核苷酸的重复基元AT/AT和AG/CT,仅有1个GC重复基元。
总的来说,野三七转录组SSR不但出现频率高,而且类型丰富;从多态性潜能的角度考虑,搜索到的这些SSR也具有较高的可用性。因此,本研究的结果为进一步开发新的野三七功能基因SSR标记奠定了基础,这种标记的建立对于加速野三七功能基因资源的开发利用、丰富其分子标记类型、遗传资源评价、绘制遗传图谱、实现特定性状的辅助选择和进行比较基因组学研究都具有重要的意义。
| [1] | Zhu S, Fushimi H, Cai S Q, et al. A new variety of the genus Panax from Southern Yunnan, China and its nucleotide sequences of 18S ribosomal RNA gene and matK gene [J]. Jap J Bot, 2003, 78: 86-94. |
| [2] | 杨兴仓, 司民真, 刘仁明, 等. 野三七的红外光谱分析 [J]. 光散射学报, 2011, 23(2): 162-167. |
| [3] | Takao K, Midori T, Harukuni T, et al. Anti-tumor-promoting activity of majonoside-R2 from Vietnamese Ginseng, Panax vietnamensis Ha et Grushv. [J]. Biol Pharm Bull, 1998, 21(8): 834-838. |
| [4] | Takao K, Midori T, Eiichiro I, et al. Cancer chemopreventive activity of majonoside-R2 from Vietnamese Ginseng, Panax vietnamensis Ha et Grushv. [J]. Cancer Lett, 1999, 147: 11-16. |
| [5] | Zhu S, Zou K, Fushimi H, et al. Comparative study on triterpene saponins of ginseng drugs [J]. Planta Med, 2004, 70: 666-677. |
| [6] | Zhu S, Zou K, Fushimi H, et al. Comparative study on triterpene saponins of ginseng drugs [J]. Planta Med, 2004, 70: 666-677. |
| [7] | Nguyen M D, Nguyen T N, Ryoji K, et al. Saponins from Vietnamese ginseng, Panax vietnamensis Ha et Grushv. collected in central Vietnam: I [J]. Chem Pharm Bull, 1993, 41(11): 2010-2014. |
| [8] | Nguyen M D, Ryoji K, Kazuhiro O, et al. Saponins from Vietnamese ginseng, Panax vietnamensis Ha et Grushv. collected in central Vietnam: II [J]. Chem Pharm Bull, 1994, 42(1): 115-122. |
| [9] | Nguyen M D, Ryoji K, Kazuhiro O, et al. Saponins from Vietnamese ginseng, Panax vietnamensis Ha et Grushv. collected in central Vietnam: III [J]. Chem Pharm Bull, 1994, 42(3): 634-640. |
| [10] | Quan L T, Ketut A, Yasuhiro T, et al. Triterpene saponins from Vietnamese ginseng (Panax vietnamensis) and their hepatocytoprotective activity [J]. J Nat Prod, 2001, 64(4): 456-461. |
| [11] | 黄映萍. DNA分子标记研究进展 [J]. 中山大学研究生学刊: 自然科学医学版, 2010, 31(2): 27-36. |
| [12] | 闫秋良. 基于生物信息学方法从牛和绵羊表达序列标签中筛选SSR标记的初步研究 [D]. 西安: 西北农林科技大学, 2007. |
| [13] | Temnykh S, de Clerck G, Lukashova A, et al. Computational and experimental analysis of microsatellites in rice (Oryza sativa L.) [J]. Genome Res, 2001, 11: 1441-1452. |
| [14] | Kantety R V, Rota M L, Matthews D E, et al. Data mining for simple sequence repeats in expressed sequence tags from barley, maize, rice, sorghum and wheat [J]. Plant Mol Biol, 2002, 48: 501-510. |
| [15] | Cardle L, Ramsay L, Mibourne D, et al. Computational and experimental characterization of physically clustered simple sequence repeats in plants [J]. Genetics, 2000, 156: 847-854. |
| [16] | Varshney R K, Graner A, Sorrells M. Genic miorosatellite markers in plants: features and applications [J]. Trends Biotechnol, 2005, 23(1): 48-55. |
| [17] | Lidan S, Weiru Y, Qixiang Z, et al. Genome-wide characterization and linkage mapping of simple sequence repeats in Mei (Prunus mume Sieb. et Zucc.) [J]. PLoS One, 2013, 8(3): e59562. |
| [18] | Cardle L, Ramsay L, Milbourne D, et al. Computational and experimental characterization of rhysically clutered simple sequence repeats in plants [J]. Genetics, 2000, 156: 847-854. |
| [19] | Marshall H, Wayne W X, Brian W K, et al. Identification of differential gene expression in Brassica rapa Nectaries through expressed sequence tag analysis [J]. PLoS One, 2010, 5(1): e8782. |
 2014, Vol. 45
2014, Vol. 45


